
Введение
С учётом актуальности Multiple instance learning (далее: MIL) и, в частности, наличия преимуществ данного метода для анализа гистологических изображений, решил попробовать обучить модели с целью классификации наборов данных, на те, которые содержат только нормальные ткани и те, в которых встречаются изображения со светлоклеточным раком почки.
В основном ориентировался на 2 проекта по данной тематике :
Имплементация MIL Attention layer на Keras - ссылка
Проект реализации Attention-based Deep Multiple Instance Learning для анализа гистологических изображений - github
Датасет
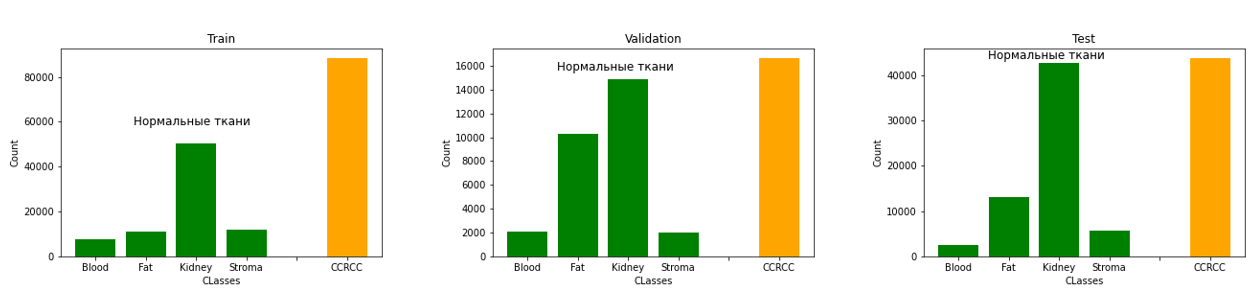
Для обучения моделей использовались датасеты, содержащие 500, 1000 и 2000 наборов (bags of instances). Соотношение позитивных (содержащих изображения со светлоклеточным раком почки) и негативных (содержащих только нормальные ткани) было 1:1. В каждом наборе присутствовало 40 цветных изображений в формате .jpeg с разрешением 256х256 пикселей, полученных с полнослайдовых изображений исследования CPTAC-CCRCC (WSI можно найти в свободном доступе на сайте Cancer Imaging Archive). В позитивных наборах 20 из 40 изображений были со светлоклеточным раком почки.
Аннотацию WSI проводил я самостоятельно ( т.к. по профессии являюсь патологоанатомом) и подробнее процесс описал в другой статье (ссылка) .
Все изображения в датасете можно разделить на 2 класса : нормальные ткани (кровь, строма, жировая ткань, ткань почки) и светлоклеточный рак почки (CCRCC).
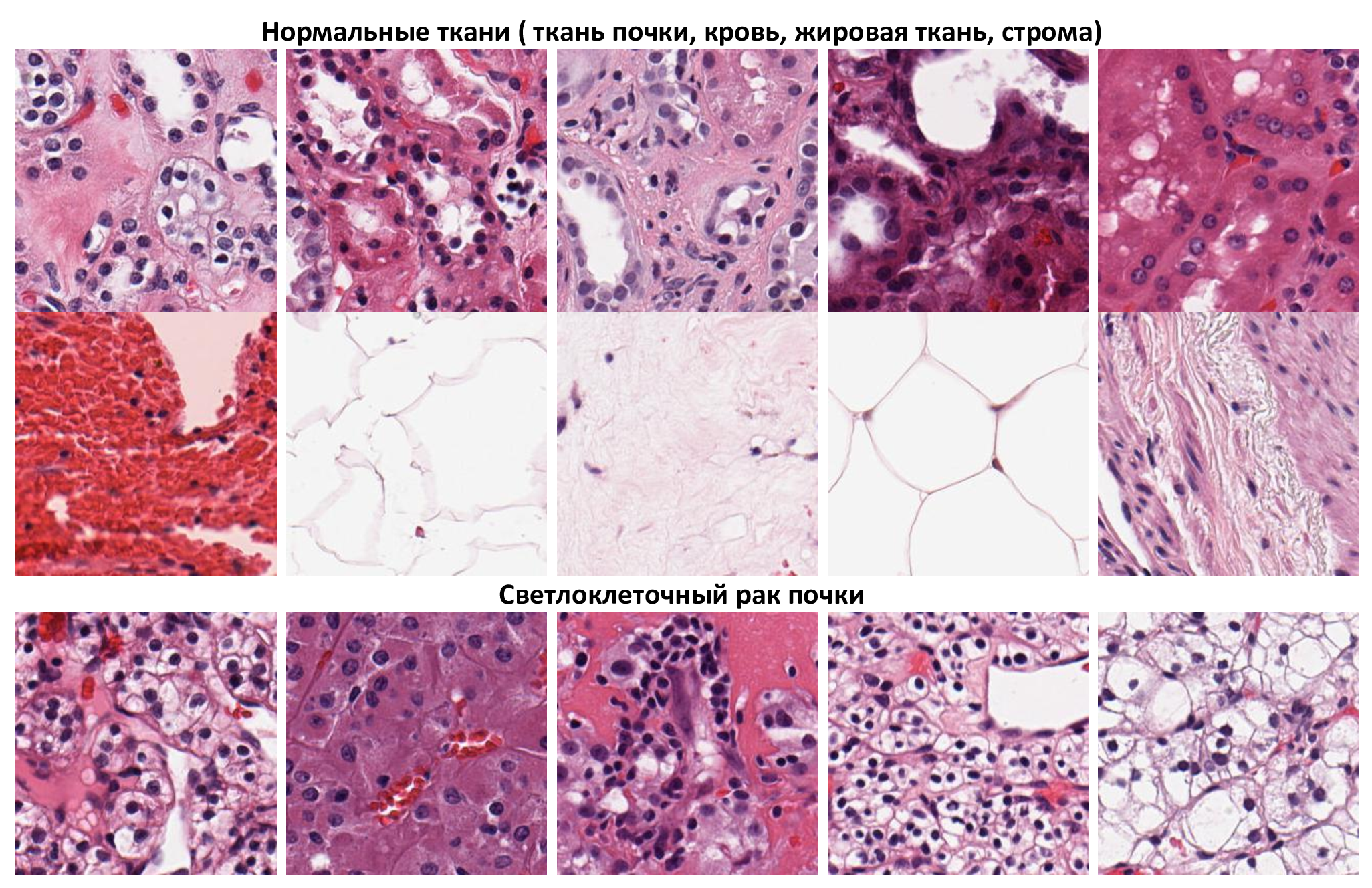
Датасеты были сгенерированы искусственно, путём случайного выбора изображений из пула изображений нормальных тканей и рака, без замены (т.е. одно изображение могло попасть только в один набор).
Модель
Код модели
from tensorflow import keras
from tensorflow.keras import layers
from keras.layers import Flatten
from keras.layers import Input, Dense, Layer, Dropout, Conv2D, MaxPooling2D, Flatten, multiply
from MILAttentionLayer import MILAttentionLayer
def SimpleModel(instance_shape,bag_size):
""" Create Keras model for Multiply Instance Learning
Parameters
-------------------
instance_shape (tuple) - shape of 1 instance in the bag
bag_size (int) - size of the bag
Returns
-------------------
keras.Model
"""
# Extract features from inputs.
inputs, embeddings = [], []
conv1_1 = Conv2D(16, kernel_size=(2,2), activation='relu')
conv1_2 = Conv2D(16, kernel_size=(2,2), activation='relu')
mpool_1 = MaxPooling2D((2,2))
conv2_1 = Conv2D(32, kernel_size=(2,2), activation='relu')
conv2_2 = Conv2D(32, kernel_size=(2,2),activation='relu')
mpool_2 = MaxPooling2D((2,2))
fc0 = Dense(512, activation='relu', name='fc0')
fc1 = Dense(512, activation='relu', name='fc1')
fc2 = Dense(256, activation= 'relu', name='fc2')
for _ in range(bag_size):
inp = layers.Input(instance_shape)
inputs.append(inp)
x = conv1_1(inp)
x = conv1_2(x)
x = mpool_1(x)
x = conv2_1(x)
x = conv2_2(x)
x = mpool_2(x)
x = Flatten()(x)
x = fc0(x)
x = Dropout(0.5)(x)
x = fc1(x)
x = Dropout(0.5)(x)
x = fc2(x)
x = Dropout(0.2)(x)
embeddings.append(x)
# Аttention layer.
alpha = MILAttentionLayer(
weight_params_dim=1024,
kernel_regularizer=keras.regularizers.l2(0),# previous - 0.01
use_gated=True,
name="alpha",
)(embeddings)
# Multiply attention weights with the input layers.
multiply_layers = [
layers.multiply([alpha[i], embeddings[i]]) for i in range(len(alpha))
]
# Concatenate layers.
concat = layers.concatenate(multiply_layers, axis=1)
# Classification output node.
output = layers.Dense(2, activation = 'softmax')(concat)
return keras.Model(inputs, output) С целью эксперимента модель была обучена на трёх датасетах с различным количеством наборов данных :
Model_500 - модель обученная на датасете, содержащем 500 наборов данных
Model_1000 - модель обученная на датасете, содержащем 1000 наборов данных
Model_2000 - модель обученная на датасете, содержащем 2000 наборов данных
Код обучения моделей
import tensorflow as tf
from CustomDataGenerator import CustomDataGenerator
from SimpleModel import SimpleModel
def train_model (train_df, validation_df, model_save_path):
"""
Train SimpleModel
Parameters
-------------------
train_df (pandas DataFrame) - DataFrame with the training data. X (bag of instances) - list of images paths. y -label
validation_df (pandas DataFrame) - DataFrame with the validation data. X (bag of instances) - list of images paths. y -label
model_save_path (str) - path for model saving
Returns
-------------------
"""
# create generator of the training and validation data
train_generator = CustomDataGenerator(df = train_df, shuffle = True, augmentations = True )
validation_generator = CustomDataGenerator (df = validation_df, shuffle = False, augmentations = False )
# Callbacks
model_checkpoint = tf.keras.callbacks.ModelCheckpoint(
model_save_path,
monitor="val_loss",
verbose=1,
mode="min",
save_best_only=True,
save_weights_only= False)
es = tf.keras.callbacks.EarlyStopping(
monitor="val_loss",
patience=10,
verbose=1,
mode="min")
# optimizer
opt = tf.keras.optimizers.Adam(learning_rate=1e-3, decay=0.0005, beta_1=0.9, beta_2=0.999)
# create and compile model
model = SimpleModel(bag_size = 40, instance_shape = (256, 256, 3) )
model.compile(optimizer = opt,
loss='categorical_crossentropy', metrics=["accuracy",tf.keras.metrics.AUC(name = 'AUC'),
tf.keras.metrics.AUC(curve = 'PR',name = 'PR_AUC'),
tf.keras.metrics.Precision(name = 'Precision', class_id = 1),
tf.keras.metrics.Recall(name = 'Recall',class_id = 1)])
# model fitting
model.fit(
train_generator,
validation_data = validation_generator ,
epochs=100,
batch_size= 1,
callbacks=[model_checkpoint,es],
verbose=1)Результаты обучения
Model |
Set |
Loss |
Accuracy |
PR_AUC |
ROC_AUC |
Precision |
Recall |
Model_500 |
Train |
0.017599 |
0.9940 |
0.99985 |
0.99985 |
0.9940 |
0.9940 |
Val |
0.013762 |
1 |
1 |
1 |
1 |
1 |
|
Model_1000 |
Train |
0.0069 |
0.9980 |
1 |
1 |
0.9980 |
0.9980 |
Val |
0.00123 |
1 |
1 |
1 |
1 |
1 |
|
Model_2000 |
Train |
0.0117 |
0.9970 |
0.9992 |
0.9994 |
0.9970 |
0.9970 |
Val |
0.00010 |
1 |
1 |
1 |
1 |
1 |
Лучшие результаты при обучении показала Model_2000, обученная на датасете, содержащем наибольшее количество данных.
Тестирование моделей
Для тестирования каждой модели было подготовлено 4 датасета с различным распределением изображений в наборе
Тестовые датасеты:
Test_40_20 - датасет, в позитивных наборах которого, из 40 изображений, 20 составляли изображения со светлоклеточным раком почки.
Test_40_10 - датасет, в позитивных наборах которого, из 40 изображений, 10 составляли изображения со светлоклеточным раком почки.
Test_40_5 - датасет, в позитивных наборах которого, из 40 изображений, 5 составляли изображения со светлоклеточным раком почки.
Test_40_1 - датасет, в позитивных наборах которого, из 40 изображений, 1 составляли изображения со светлоклеточным раком почки.
С целью эксперимента хотел посмотреть, какие результаты покажут модели на датасетах с меньшим количеством позитивных изображений в наборе.
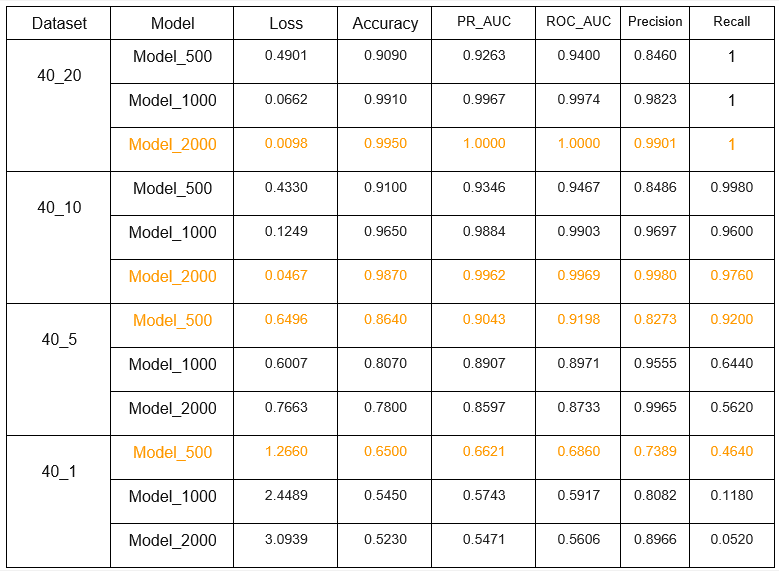
Результаты тестирования
Confusion matrix
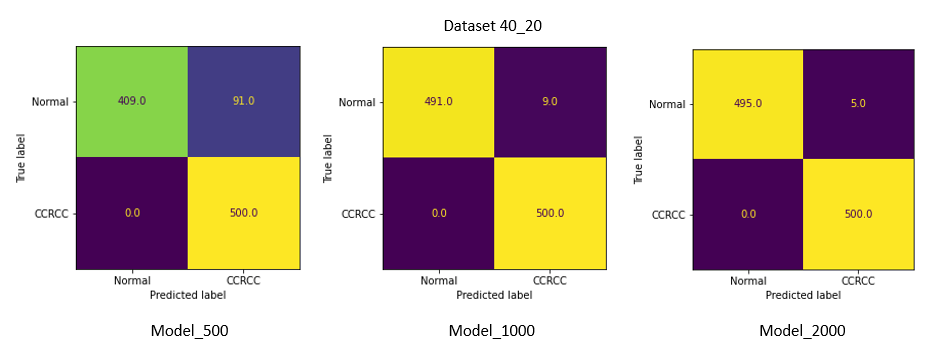
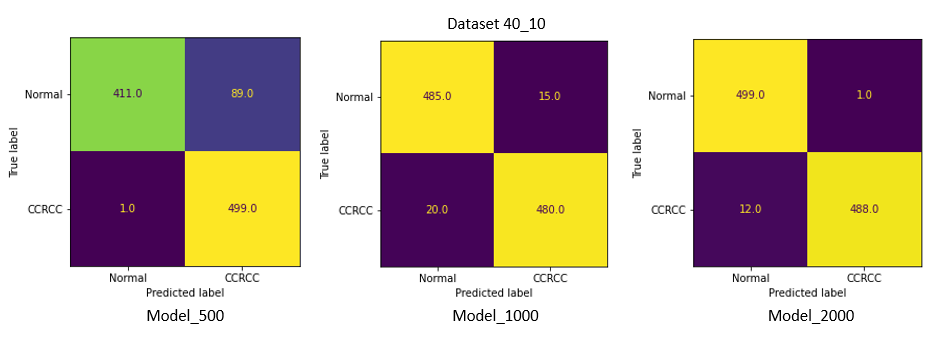
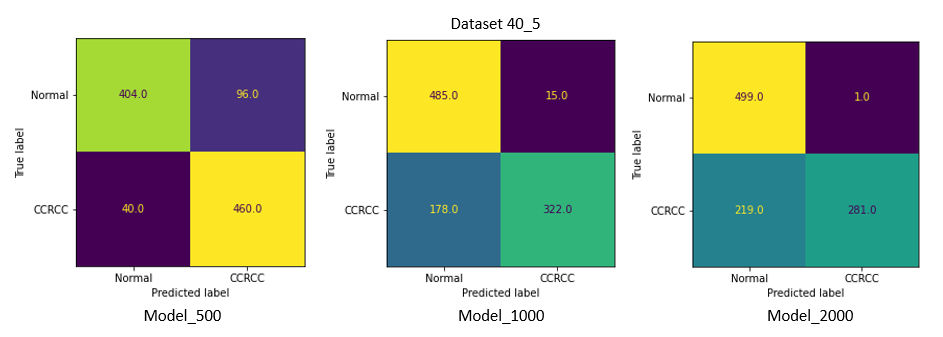
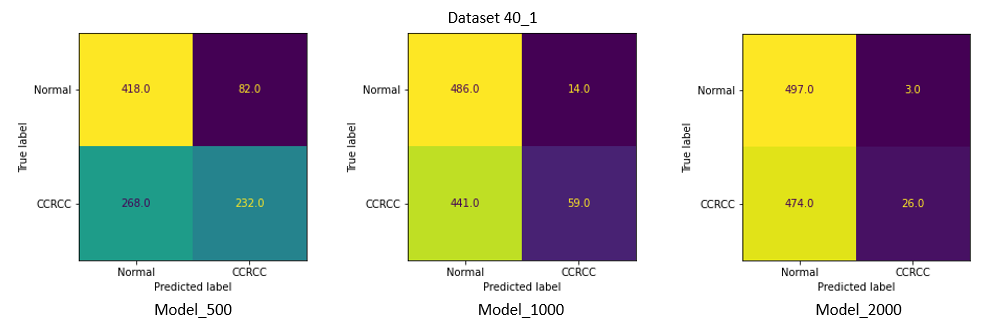
Наилучшие результаты на 40_20 и 40_10 показала Model_2000 с точностью в 99.5 % и 98.7 % соответственно. Recall (в данной задаче приоритетнее, чем точность, из-за нежелательных ложно-негативных срабатываний) составил 1 и 0.976.
Однако на датасетах 40_5 и 40_1, которые содержали наименьшее количество изображений светлоклеточного рака, качество всех моделей сильно снизилось, и лучшие результаты уже у модели, обученной на наименьшем количестве данных (Model_500) .
Комментарии (3)

shadrap
31.10.2022 18:25с какой стадии визуальный контроль, становится в принципе возможным? я так понимаю что где-то с 3й стадии они приобретают названные свойства, типа светлой цитоплазмы , увеличенного ядра и тп

shadrap
спасибо интересно. я так понимаю, основной критерий идентификации клетки как имеющий СКР это эозинофильный окрас?
Hardrockmaniac Автор
В большинстве изображений опухолевые клетки крупнее, со светлой цитоплазмой,не формируют канальцевых ( как в ткани почки) структур.
Иногда,действительно,может быть трудно отличить от почки,хотя нейросети обученные для классификации каждого изображения показывали неплохие результаты,иногда неправильно классифицируя рак как ткань почки или,что удивительно, строму.