В предыдущей подробной статье про Полный геном мы обещали опубликовать три задачи и подарить тест тому, кто первым решит все три правильно. Заодно в этих задачах мы даем примеры, как можно работать с генетическими данными. Сегодня публикуем первую.

В первой статье мы делились полезной информацией и ссылками, которые пригодятся для работы с биоинформатическими данными. Советуем сначала прочитать ее, если вы пропустили.
Дисклеймер
Работа с генетическими данными проводится на Unix системах (Linux, macOS), так как некоторые команды и ПО недоступны на Windows. Поэтому для пользователей Windows одним из самых простых решений будет арендовать виртуальную машину с Linux.
Все описанные ниже операции выполняются в командной строке — терминале. Перед началом выполнения узнайте, как работать в терминале под управлением вашей ОС и использовать команды, так как некоторые из них могут потенциально навредить ОС и вашим данным.
Мы собрали образ виртуальной машины (ВМ) со всем необходимым ПО на Яндекс.Облаке. Зарегистрируйтесь в Яндекс.Облаке, в личном кабинете в разделе Compute Cloud нажмите «Создать ВМ». В качестве публичного образа выберите из каталога «Атлас Анализ Данных 1000 Геномов».
Конфигурация ВМ: 100% 2vCPU, 8GB RAM, 20GB HDD. При создании ВМ необходимо ввести платежные данные, но со счета ничего не спишется. Стартового и дополнительного гранта по кодовому слову достаточно, чтобы работать c ВМ и образом от Атласа до 31 декабря 2019 года бесплатно. Чтобы получить грант на прохождение задач, отправьте службе поддержки Яндекс.Облака кодовое слово «АТЛАС».
Примечание: грант действителен для новых пользователей Яндекс.Облака, которые завели аккаунт с 18 декабря 2019 года или для тех, у кого еще действует пробный период и имеется стартовый грант. Кодовое слово «АТЛАС» действует только один раз.
Предварительно создайте ssh ключ на локальном компьютере, с которого вы планируете подключаться к ВМ:
Не забудьте скопировать содержимое
Если вы хотите установить ПО на свой компьютер, ниже есть вся информация по установке. Если вы решили воспользоваться Яндекс.Облаком — создайте ВМ и переходите к следующему разделу.
Plink — программный пакет для манипуляций с генетическими данными и широкогеномного поиска ассоциаций (GWAS). Его разработал генетик Шон Перселл (Shaun Purcell). С помощью Plink с 2008 года были выполнены сотни GWAS’ов по всему миру, результаты лучших из которых Атлас использует как источник данных для алгоритмов расчета рисков заболеваний.
Plink предлагает набор инструментов для хранения и конвертации данных генотипирования и поиска по ним. Также Plink позволяет производить статистическую обработку, анализ неравновесного сцепления (linkage disequilibrium, LD), анализ идентичности по происхождению (identity by descent, IBD) и по состоянию (identity by state, IBS), популяционную стратификацию и тесты на эпистаз — взаимодействие нескольких генетических вариаций между собой.
IBD и IBS используются для анализа популяционного состава и определения родства.
Пример эпистаза — вариации rs7412 и rs429358 в гене APOE, определенное сочетание вариантов которых резко повышает риск развития болезни Альцгеймера, тогда как каждый вариант по отдельности вносит лишь незначительный вклад в риск.
Скачайте стабильную версию Plink с официального сайта.
BCFtools — набор утилит для манипулирования генетическими данными в формате VCF и его бинарном аналоге BCF. Список возможных применений пакета BCFtools включает аннотацию, фильтрацию, слияние и разделение VCF/BCF файлов, нахождение их пересечения, индексирование, выборочный поиск, сортировку, подсчет статистики и т.п.
Для установки выполните:
Подробнее процесс установки описан здесь.
Пакет KING (Kinship-based INference for Gwas) используется в популяционных исследованиях при работе с данными полногеномного поиска ассоциаций для определения родственных связей в исследуемых данных. В этой задаче KING поможет определить степень родства нескольких образцов из проекта «1000 Геномов».
Скачать можно здесь. Для решения задач пригодится руководство к KING по ссылке.
Для руководства мы используем открытые данные проекта «1000 Геномов». Для анализа мы выбрали 10 образцов с информацией о генотипах около 85 миллионов вариаций, полученных путем анализа данных NGS с выравниванием на версию референсного генома GRCh37. Родственные связи и популяции образцов указаны на Рисунке 1.

Рисунок 1. Родословная используемых в VCF образцов. Квадрат соответствует мужскому полу, круг — женскому. Пунктирная линия означает неустановленное родство второго порядка.
Формат VCF позволяет хранить информацию о поле человека в виде одного числа, если эта информация была известна при генерации VCF. Выглядит это так: поле GT (genotype, генотип) для записей с Х хромосомы содержит одно численное значение, которое соответствует одной аллели, для мужчин и два — для женщин. Если информация о биологическом поле секвенируемого образца отсутствует, то поле GT будет по умолчанию содержать два числовых значения (выделены красным на рисунке 2).
В используемых в данном руководстве VCF файлах Y-хромосома исключена, но и наличие хромосомы Y в VCF файле далеко не всегда говорит о том, что она действительно есть у секвенируемого образца. Это происходит из-за псевдоаутосомных регионов (PARs), которые идентичны для Х и Y хромосом и расположены на их концах.
Разные хромосомы в норме не имеют длинных идентичных (гомологичных) участков, однако хромосомы Х и Y обладают такими участками длиной по несколько миллионов пар оснований в самом начале (PAR1) и конце (PAR2). Поэтому при анализе данных NGS у мужчин в PAR регионах обнаруживаются два аллеля (по одному с каждой половой хромосомы), а у женщин могут появиться генотипы в PAR регионах Y хромосомы, хотя на самом деле это генотипы с их X-хромосомы.
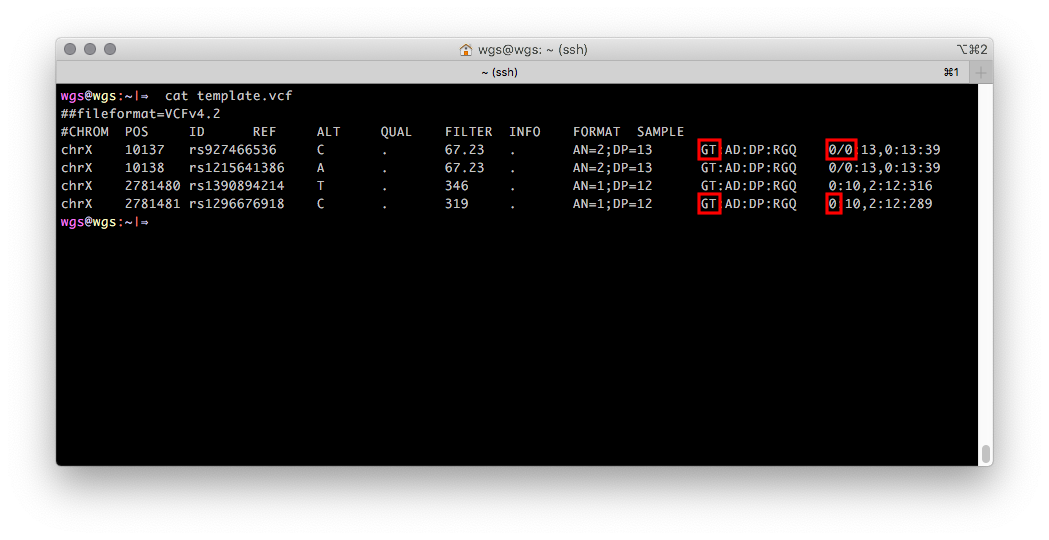
Рисунок 2. VCF файл с генотипами с Х хромосомы мужчины из региона PAR1 (первые две записи) и непсевдоаутосомного региона (последние две записи).
VCF файлы для выполнения заданий в неархивированном виде занимают около 19 гигабайт каждый, поэтому для экономии места мы рекомендуем работать только с архивами. Все перечисленные выше программы уже умеют работать со сжатыми данными VCF. Дополнительно ничего делать не нужно.
Чтобы определить пол субъекта, нужно посмотреть на генотипы на Х хромосоме и исключить регионы PAR1 и PAR2, расположенные в ее начале и конце. Это интервалы позиций 60001–2699520 и 154931044–155260560 в GRCh37 версии генома. Если генотип будет содержать одно численное обозначение — это мужской биологический пол, иначе — женский. Нужно иметь в виду, что обозначение пола в VCF файле зависит от наличия информации о биологическом поле при генерации VCF, поэтому использовать данный подход можно не всегда.
Используйте следующую команду для каждого из образцов в датасете. Подставьте идентификатор образца после аргумента
При выполнении команд вы увидите часть содержимого VCF файла для указанного идентификатора образца. Параметр
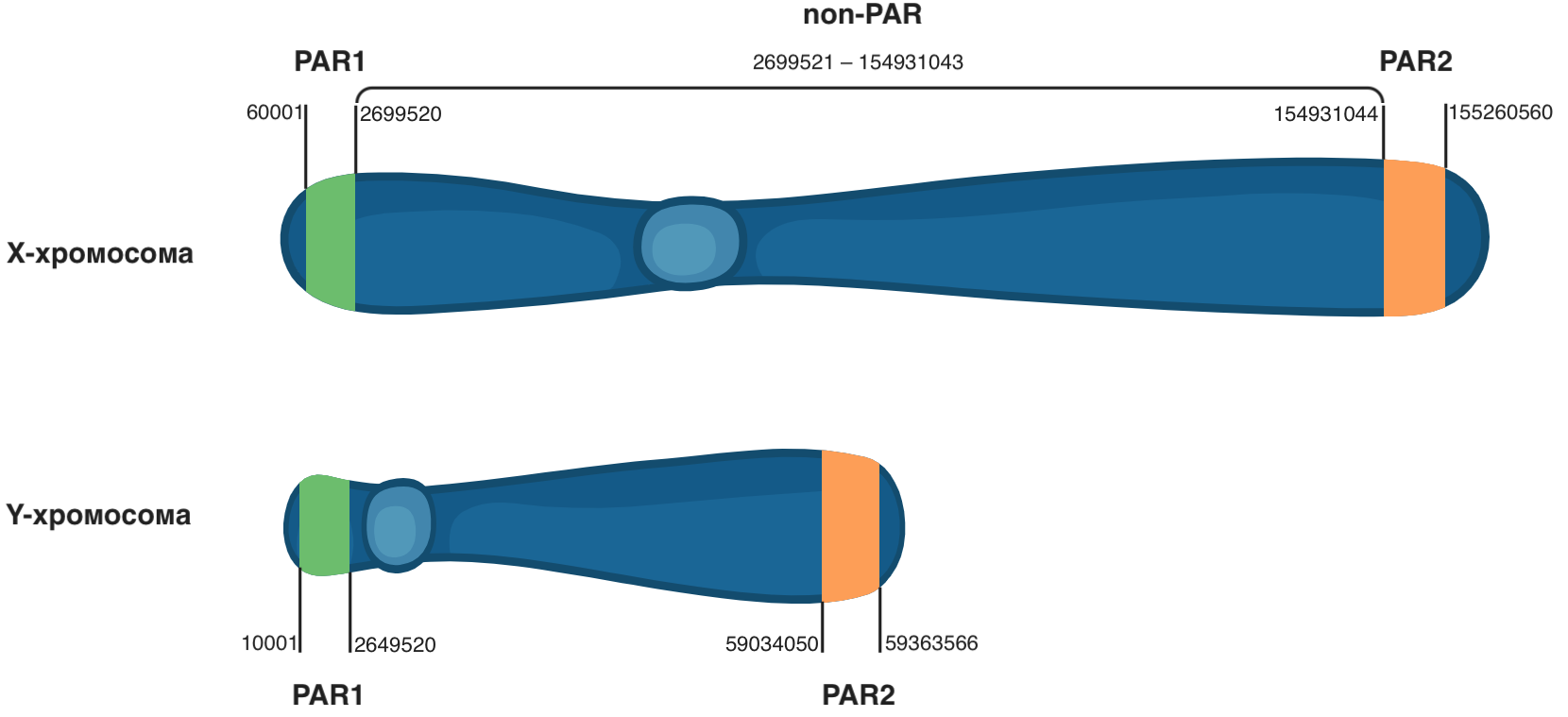
Рисунок 3. Расположение псевдоаутосомных регионов PAR1 и PAR2 на половых хромосомах.
Посмотреть список всех идентификаторов образцов в VCF файле можно на Рисунке 1 или в последней строке шапки VCF файла. Они будут перечислены после наименования колонки FORMAT:
Истинный пол данных образцов также указан на Рисунке 1.
Для определения родства нам нужно попарно сравнить генетические данные всех образцов. Делать это по полному геному сложно: VCF файл в таком случае занимает десятки гигабайт. Используемый нами VCF занимает всего около 2 гигабайт, но мы все равно отфильтруем его по списку идентификаторов генетических вариаций (rsIDs), генотипируемых на чипах от компании Illumina: GSA v1, GSA v2, HumanOmniExpress v1.0, HumanOmniExpress v1.3, InfiniumExome v1.1 и InfiniumOmniExpressExome v1.4. Это наиболее популярные чипы в коммерческом генотипировании.
Мы собрали список всех идентификаторов генетических вариаций с этих чипов в отдельный файл со списком rsIDs. Он содержит 1,4 миллиона идентификаторов. Для фильтрации VCF файла выполните следующую команду:
При каждом использовании BCFtools и других пакетов для работы с VCF файлами в шапку файла добавляется история предыдущих команд. Независимо от способа фильтрации VCF файла и выполненных ранее команд, можно проверить целостность и идентичность основного содержимого VCF подсчетом хеш-суммы:
Команда
В случае совпадения хеш-суммы можно двигаться дальше и конвертировать VCF во внутренний формат Plink (по умолчанию формат Plink — это три файла с расширениями bed, bim и fam). В этих файлах остается только генотип, хромосома, позиция и некоторые другие данные, а остальное — отсеивается. С таким форматом гораздо проще работать и решать различные задачи, не требующие дополнительной информации из VCF. Например проводить GWAS.
Эта команда создаст в папке три файла:
Определить попарное родство можно для всех 10 образцов. Используем следующую команду для анализа Plink файлов:
Просмотрите файл
Сравните полученные вами коэффициенты в файле
Используя тестовый датасет из 12 образцов
Как мы писали выше, kinship коэффициенты не могут однозначно охарактеризовать родство того или иного порядка: одинаковые kinship коэффициенты получаются при сравнении пар родитель — ребенок и брат — сестра (родство первого порядка). При невозможности установить характер родственной связи, укажите любую из возможных. Обратите внимание, что образцы в тестовом датасете имеют отличные от используемых в датасете для обучения идентификаторы.
Ответы присылайте на почту wgs@atlas.ru до 26 декабря до 23:59. Еще две задачи будут скоро опубликованы, а итоговые результаты по задачам появятся 28 декабря. Победитель получит тест Полный геном, а второе и третье места — генетический тест «Атлас». Также будут специальные призы от Яндекс.Облако. Бывшие и настоящие сотрудники Атласа в конкурсе не участвуют ;)

В первой статье мы делились полезной информацией и ссылками, которые пригодятся для работы с биоинформатическими данными. Советуем сначала прочитать ее, если вы пропустили.
Дисклеймер
Работа с генетическими данными проводится на Unix системах (Linux, macOS), так как некоторые команды и ПО недоступны на Windows. Поэтому для пользователей Windows одним из самых простых решений будет арендовать виртуальную машину с Linux.
Все описанные ниже операции выполняются в командной строке — терминале. Перед началом выполнения узнайте, как работать в терминале под управлением вашей ОС и использовать команды, так как некоторые из них могут потенциально навредить ОС и вашим данным.
Необходимое ПО
Мы собрали образ виртуальной машины (ВМ) со всем необходимым ПО на Яндекс.Облаке. Зарегистрируйтесь в Яндекс.Облаке, в личном кабинете в разделе Compute Cloud нажмите «Создать ВМ». В качестве публичного образа выберите из каталога «Атлас Анализ Данных 1000 Геномов».
Конфигурация ВМ: 100% 2vCPU, 8GB RAM, 20GB HDD. При создании ВМ необходимо ввести платежные данные, но со счета ничего не спишется. Стартового и дополнительного гранта по кодовому слову достаточно, чтобы работать c ВМ и образом от Атласа до 31 декабря 2019 года бесплатно. Чтобы получить грант на прохождение задач, отправьте службе поддержки Яндекс.Облака кодовое слово «АТЛАС».
Примечание: грант действителен для новых пользователей Яндекс.Облака, которые завели аккаунт с 18 декабря 2019 года или для тех, у кого еще действует пробный период и имеется стартовый грант. Кодовое слово «АТЛАС» действует только один раз.
Предварительно создайте ssh ключ на локальном компьютере, с которого вы планируете подключаться к ВМ:
ssh-keygen -o -t rsa -b 4096 -C "my-local-machine" -f ~/.ssh/yandex-cloud -a 100
Не забудьте скопировать содержимое
~/.ssh/yandex-cloud.pub файла в соответствующее окно при создании ВМ.Если вы хотите установить ПО на свой компьютер, ниже есть вся информация по установке. Если вы решили воспользоваться Яндекс.Облаком — создайте ВМ и переходите к следующему разделу.
Plink
Plink — программный пакет для манипуляций с генетическими данными и широкогеномного поиска ассоциаций (GWAS). Его разработал генетик Шон Перселл (Shaun Purcell). С помощью Plink с 2008 года были выполнены сотни GWAS’ов по всему миру, результаты лучших из которых Атлас использует как источник данных для алгоритмов расчета рисков заболеваний.
Plink предлагает набор инструментов для хранения и конвертации данных генотипирования и поиска по ним. Также Plink позволяет производить статистическую обработку, анализ неравновесного сцепления (linkage disequilibrium, LD), анализ идентичности по происхождению (identity by descent, IBD) и по состоянию (identity by state, IBS), популяционную стратификацию и тесты на эпистаз — взаимодействие нескольких генетических вариаций между собой.
IBD и IBS используются для анализа популяционного состава и определения родства.
Пример эпистаза — вариации rs7412 и rs429358 в гене APOE, определенное сочетание вариантов которых резко повышает риск развития болезни Альцгеймера, тогда как каждый вариант по отдельности вносит лишь незначительный вклад в риск.
Скачайте стабильную версию Plink с официального сайта.
BCFtools
BCFtools — набор утилит для манипулирования генетическими данными в формате VCF и его бинарном аналоге BCF. Список возможных применений пакета BCFtools включает аннотацию, фильтрацию, слияние и разделение VCF/BCF файлов, нахождение их пересечения, индексирование, выборочный поиск, сортировку, подсчет статистики и т.п.
Для установки выполните:
git clone git://github.com/samtools/htslib.git
git clone git://github.com/samtools/bcftools.git
cd bcftools
# The following is optional:
# autoheader && autoconf && ./configure --enable-libgsl --enable-perl-filters
makeПодробнее процесс установки описан здесь.
KING
Пакет KING (Kinship-based INference for Gwas) используется в популяционных исследованиях при работе с данными полногеномного поиска ассоциаций для определения родственных связей в исследуемых данных. В этой задаче KING поможет определить степень родства нескольких образцов из проекта «1000 Геномов».
Скачать можно здесь. Для решения задач пригодится руководство к KING по ссылке.
Почти все ошибки, которые могут возникнуть в процессе работы с инструментами, описаны на Stackoverflow или его биоинформатическом аналоге — Biostars.
Используемые данные
Для руководства мы используем открытые данные проекта «1000 Геномов». Для анализа мы выбрали 10 образцов с информацией о генотипах около 85 миллионов вариаций, полученных путем анализа данных NGS с выравниванием на версию референсного генома GRCh37. Родственные связи и популяции образцов указаны на Рисунке 1.

Рисунок 1. Родословная используемых в VCF образцов. Квадрат соответствует мужскому полу, круг — женскому. Пунктирная линия означает неустановленное родство второго порядка.
Взять на заметку
Формат VCF позволяет хранить информацию о поле человека в виде одного числа, если эта информация была известна при генерации VCF. Выглядит это так: поле GT (genotype, генотип) для записей с Х хромосомы содержит одно численное значение, которое соответствует одной аллели, для мужчин и два — для женщин. Если информация о биологическом поле секвенируемого образца отсутствует, то поле GT будет по умолчанию содержать два числовых значения (выделены красным на рисунке 2).
В используемых в данном руководстве VCF файлах Y-хромосома исключена, но и наличие хромосомы Y в VCF файле далеко не всегда говорит о том, что она действительно есть у секвенируемого образца. Это происходит из-за псевдоаутосомных регионов (PARs), которые идентичны для Х и Y хромосом и расположены на их концах.
Разные хромосомы в норме не имеют длинных идентичных (гомологичных) участков, однако хромосомы Х и Y обладают такими участками длиной по несколько миллионов пар оснований в самом начале (PAR1) и конце (PAR2). Поэтому при анализе данных NGS у мужчин в PAR регионах обнаруживаются два аллеля (по одному с каждой половой хромосомы), а у женщин могут появиться генотипы в PAR регионах Y хромосомы, хотя на самом деле это генотипы с их X-хромосомы.

Рисунок 2. VCF файл с генотипами с Х хромосомы мужчины из региона PAR1 (первые две записи) и непсевдоаутосомного региона (последние две записи).
Образовательный блок
Генетический пол — это набор половых хромосом, соответствующих проявлению первичных и вторичных половых признаков по мужскому или женскому типу. В норме мужчины имеют одну хромосому Х и одну хромосому Y, в то время как женщины имеют две хромосомы X. При различных нарушениях образования половых клеток, яйцеклеток и сперматозоидов, у родителей может родиться ребенок с отличным набором половых хромосом, что часто приводит к нарушениям развития первичных и вторичных половых признаков.
Две самых частых хромосомных половых аномалии — это синдром Тёрнера (набор хромосом X0, то есть всего одна X-хромосома) и синдром Клайнфельтера (набор хромосом XXY).
Аллель — один или более нуклеотид, который находится в какой-либо позиции генома и имеет альтернативный вариант. Понятие используют при описании генотипов. Различают референсные аллели и альтернативные. Все они хранятся в VCF файле в полях REF и ALT, соответственно.
Определяем пол
Для пользователей Яндекс.Облака
Все данные для выполнения руководства и самостоятельных заданий хранятся на Яндекс.Облаке с использованием структуры, изображенной ниже. Папка
В каталоге
Tutorial содержит VCF файл, необходимый для выполнения руководства, папка Test — для выполнения самостоятельных заданий. В папке Technical содержится два файла со списком идентификаторов генетических вариаций: rsids_for_subsetting.txt используется в руководстве и заданиях для самостоятельного выполнения, external_interpretation_rsids.txt может понадобиться в будущем при приобретении полногеномного секвенирования в Атлас для загрузки данных генотипирования в сторонние сервисы. Папка Tools содержит помимо прочего два скрипта, используемые в заданиях 2 и 3.home
L-- ubuntu
+-- Data
¦ +-- Test
¦ ¦ +-- CEI.1kg.2019.test.vcf.gz
¦ ¦ L-- CEI.1kg.2019.test.vcf.gz.tbi
¦ L-- Tutorial
¦ +-- CEI.1kg.2019.demo.vcf.gz
¦ L-- CEI.1kg.2019.demo.vcf.gz.tbi
+-- Technical
¦ +-- external_interpretation_rsids.txt
¦ L-- rsids_for_subsetting.txt
L-- Tools
+-- convert_plink_delimiter.sh
L-- create_23andme.shВ каталоге
/home на ВМ Яндекс.Облака будет создана папка, название которой соответствует имени пользователя, указанному на этапе создания ВМ. Скопируйте всё из каталога /home/ubuntu в свой каталог через следующие команды:cd ~
cp -r /home/ubuntu/* ./Для остальных
При работе на личном ПК скачать необходимые файлы для выполнения первой задачи вы можете по ссылке. В скачиваемом архиве поддерживается структура хранения файлов, аналогичная используемой на Яндекс.Облаке:
Распакуйте
home
L-- ubuntu
+-- Data
¦ +-- Test
¦ ¦ +-- CEI.1kg.2019.test.vcf.gz
¦ ¦ L-- CEI.1kg.2019.test.vcf.gz.tbi
¦ L-- Tutorial
¦ +-- CEI.1kg.2019.demo.vcf.gz
¦ L-- CEI.1kg.2019.demo.vcf.gz.tbi
+-- Technical
¦ +-- external_interpretation_rsids.txt
¦ L-- rsids_for_subsetting.txt
L-- Tools
+-- convert_plink_delimiter.sh
L-- create_23andme.shРаспакуйте
atlas_wgs_contest.tar.gz архив командойtar -xvzf atlas_wgs_contest.tar.gzVCF файлы для выполнения заданий в неархивированном виде занимают около 19 гигабайт каждый, поэтому для экономии места мы рекомендуем работать только с архивами. Все перечисленные выше программы уже умеют работать со сжатыми данными VCF. Дополнительно ничего делать не нужно.
Чтобы определить пол субъекта, нужно посмотреть на генотипы на Х хромосоме и исключить регионы PAR1 и PAR2, расположенные в ее начале и конце. Это интервалы позиций 60001–2699520 и 154931044–155260560 в GRCh37 версии генома. Если генотип будет содержать одно численное обозначение — это мужской биологический пол, иначе — женский. Нужно иметь в виду, что обозначение пола в VCF файле зависит от наличия информации о биологическом поле при генерации VCF, поэтому использовать данный подход можно не всегда.
Используйте следующую команду для каждого из образцов в датасете. Подставьте идентификатор образца после аргумента
-s:(/Data/Tutotrial/CEI.1kg.2019.demo.vcf.gz):
# перейдите в нужную вам директорию
bcftools view -O v -H -s HG00731 -r chrX:2699521-154931043 CEI.1kg.2019.demo.vcf.gz | headПри выполнении команд вы увидите часть содержимого VCF файла для указанного идентификатора образца. Параметр
-r chrX:2699521-154931043 в BCFtools ограничивает просмотр содержимого файла до региона хромосомы Х с позиции 2699521 до позиции 154931043 (регион non-PAR на Рисунке 3). Эти границы исключают ненужные в данном случае псевдоаутосомные регионы (PAR1 и PAR2). По численным значениям в поле GT определите пол каждого образца.
Рисунок 3. Расположение псевдоаутосомных регионов PAR1 и PAR2 на половых хромосомах.
Посмотреть список всех идентификаторов образцов в VCF файле можно на Рисунке 1 или в последней строке шапки VCF файла. Они будут перечислены после наименования колонки FORMAT:
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT HG00731 HG00732 HG02026 HG02025 HG02250 HG02373 HG00733 HG02024 HG02377 HG02381Истинный пол данных образцов также указан на Рисунке 1.
Определяем родство
Для определения родства нам нужно попарно сравнить генетические данные всех образцов. Делать это по полному геному сложно: VCF файл в таком случае занимает десятки гигабайт. Используемый нами VCF занимает всего около 2 гигабайт, но мы все равно отфильтруем его по списку идентификаторов генетических вариаций (rsIDs), генотипируемых на чипах от компании Illumina: GSA v1, GSA v2, HumanOmniExpress v1.0, HumanOmniExpress v1.3, InfiniumExome v1.1 и InfiniumOmniExpressExome v1.4. Это наиболее популярные чипы в коммерческом генотипировании.
Мы собрали список всех идентификаторов генетических вариаций с этих чипов в отдельный файл со списком rsIDs. Он содержит 1,4 миллиона идентификаторов. Для фильтрации VCF файла выполните следующую команду:
bcftools view -O z -i 'ID=@rsids_for_subsetting.txt' CEI.1kg.2019.demo.vcf.gz > CEI.1kg.2019.demo.subset.vcf.gzПри каждом использовании BCFtools и других пакетов для работы с VCF файлами в шапку файла добавляется история предыдущих команд. Независимо от способа фильтрации VCF файла и выполненных ранее команд, можно проверить целостность и идентичность основного содержимого VCF подсчетом хеш-суммы:
# перейдите в нужную вам директорию
gunzip -c CEI.1kg.2019.demo.subset.vcf.gz | grep -v "^#" > CEI.1kg.2019.demo.subset.body.vcf
md5sum --tag CEI.1kg.2019.demo.subset.body.vcf
# Вывод последней команды должен совпадать с d609eb330908d4eb5feb9f1860fd508bКоманда
gunzip -c производит декомпрессию файла с выводом его содержимого в stdout, из которого далее удаляются строки шапки VCF файла, начинающиеся с # (поэтому используется команда grep -v "^#"). Шапка удаляется для того, чтобы сравнивать целостность только самих генетических данных, а не метаданных о том, какие инструменты и когда были использованы для работы с этим VCF файлом.В случае совпадения хеш-суммы можно двигаться дальше и конвертировать VCF во внутренний формат Plink (по умолчанию формат Plink — это три файла с расширениями bed, bim и fam). В этих файлах остается только генотип, хромосома, позиция и некоторые другие данные, а остальное — отсеивается. С таким форматом гораздо проще работать и решать различные задачи, не требующие дополнительной информации из VCF. Например проводить GWAS.
# Перейдите в нужную директорию
plink --vcf CEI.1kg.2019.demo.subset.vcf.gz --make-bed --out CEI.1kg.2019.demo.subset
Эта команда создаст в папке три файла:
CEI.1kg.2019.demo.subset.bed
CEI.1kg.2019.demo.subset.bim
CEI.1kg.2019.demo.subset.famОпределить попарное родство можно для всех 10 образцов. Используем следующую команду для анализа Plink файлов:
king -b CEI.1kg.2019.demo.subset.bed --kinship --prefix CEI.1kg.2019.demo.subset.kinship_analysisПросмотрите файл
CEI.1kg.2019.demo.subset.kinship_analysis.kin0 и обратите внимание на колонку Kinship, в которой содержатся kinship коэффициенты для пар образцов, указанных соответственно в ID1 и ID2.Сравните полученные вами коэффициенты в файле
CEI.1kg.2019.demo.subset.kinship_analysis.kin0 для всех пар образцов с родословной, представленной на Рисунке 1 (пунктирная линия соответствует родству второго порядка, однако точные данные о родстве отсутствуют, т.е. это могут быть двоюродные брат и сестра, тетя/племянник или дядя/племянница). Попробуйте самостоятельно сделать вывод о том, какие значения kinship коэффициентов могут соответствовать родству первого и второго порядка.Подсказка
Выдержка из документации KING: значения kinship коэффициентов >0.354 соответствуют дубликатам образцов либо однояйцевым близнецам, от 0.177 до 0.354 — родству первого порядка (родители-дети, братья-сестры), от 0.0884 до 0.177 — родству второго порядка (двоюродные братья-сестры, тети/дяди-племянники), а от 0.0442 до 0.0884 — родству третьего порядка (бабушки/дедушки-внуки, троюродные братья/сестры). Все, что меньше 0.0442, сложно однозначно интерпретировать.
Первое задание конкурса
Используя тестовый датасет из 12 образцов
Data/Test/CEI.1kg.2019.test.vcf.gz, составьте их родословную, руководствуясь результатами определения пола и kinship анализа. Образцы, которые по результатам анализа не находятся в родственных связях с кем-либо, запишите рядом, не соединяя их линией с другими образцами. Родословная может быть составлена в стилистике, аналогичной Рисунку 1, однако это остается на ваше усмотрение. Мужчины обозначаются квадратом, женщины — кругом, брак — горизонтальной линией, ребенок — вертикальной линией, несколько детей — горизонтальным разветвлением вертикальной линии (в виде буквы П). Подробнее про эти обозначения можно почитать здесь.Как мы писали выше, kinship коэффициенты не могут однозначно охарактеризовать родство того или иного порядка: одинаковые kinship коэффициенты получаются при сравнении пар родитель — ребенок и брат — сестра (родство первого порядка). При невозможности установить характер родственной связи, укажите любую из возможных. Обратите внимание, что образцы в тестовом датасете имеют отличные от используемых в датасете для обучения идентификаторы.
Ответы присылайте на почту wgs@atlas.ru до 26 декабря до 23:59. Еще две задачи будут скоро опубликованы, а итоговые результаты по задачам появятся 28 декабря. Победитель получит тест Полный геном, а второе и третье места — генетический тест «Атлас». Также будут специальные призы от Яндекс.Облако. Бывшие и настоящие сотрудники Атласа в конкурсе не участвуют ;)
Комментарии (20)

shadrap
20.12.2019 18:47Чипы хорошо зарекомендовали себя в агро-секторе, на животных и растениях. Там они идут как пирожки. Видимо не так критична валидность точечной ошибки, если в целом все совпадает.
Специалисты считают, что чипы для генотипирования человека, в скором времени станут узко направленными. Такие как Human 24 или HumanCoreExome, на которых и Атлас и 23иМе и прочие делают генотипирование окончательно перейдут в класс «развлекательных» или уйдут.
У Атласа, если я не ошибаюсь, свой, кастомизированный чип, в целом он совпадает с стандартным Illumina вским, но есть различия.

solarplexus
95 т.р. за количество отчетов (получение информации, которая не пригодится) — многовато, учитывая, что обычный тест (здоровье + родословная) стоит всего-то 8 т.р.
synka
А просто на FF сейчас скидка, всего 49$. До 26 декабря.
asyaaam Автор
Вы сравниваете совсем разные продукты. Основа тестов 23andme — технологий ДНК-микрочипа. Она позволяет определить около 700 000 вариантов генов, это меньше 0,1% ДНК. У этой технологии есть определенные ограничения.
Основа теста Полный геном — полногеномное секвенирование NGS. Эта технология позволяет прочитать 95% всей ДНК. По исходным данным ДНК-микрочипа нельзя оценить риски онкологических заболеваний, эффективность лекарств, провести поиск мутаций, связанных со здоровьем, который вручную делает биоинформатик. Также по мере появления новых исследований, мы будем добавлять новые данные в интерпретацию. С ДНК-микрочипом так работает не всегда, так как на нем, опять же, ограниченное число вариантов генов.
kablag
"По исходным данным ДНК-микрочипа нельзя оценить риски онкологических заболеваний, эффективность лекарств, провести поиск мутаций, связанных со здоровьем, который вручную делает биоинформатик."
Разве 23andme даёт исходные данные чипа без обработки?
asyaaam Автор
23andme выдает сырые данные генотипирования, но их недостаточно для таких отчетов. Для них подходят только данные полногеномного секвенирования с глубиной покрытия не менее х30
kablag
что значит сырые данные генотипирования? чипы выдают аллельные варианты в конкретных точках. Полногеномное секвенирование принципиально выдаёт тоже самое только для большего количества точек (при этом Вы решили, что для достоверного определения хватает 30х покрытия – ваше право, но к сравнению точности методов это не применимо: Вы сами правильно отметили, что это разные методики).
Поэтому если кто-то выбирает между методами, то единственный критерий: нужно ему столько данных или нет. Полногеномное секвенирование – это возможный задел на будущее, но в наших реалиях и данных чипов с излишком.
А по поводу интерпретации данных – те же 23andme уже обжигались на этом. Слишком это «щепетильная» информация давать выводы о возможных предрасположенностях к заболеваниям неподготовленным пользователям.
asyaaam Автор
Да, «сырые» обработанные данные генотипирования — это аллельные варианты в конкретных точках. Набор этих конкретных точек определяется чипом, на котором проводится генотипирование. В силу этого обстоятельства, а также того, что все интересные/важные/клинически значимые генетические вариации на один чип «не засунуть», возникает необходимость использовать технологии, позволяющие максимально полным образом описать генетическую вариабельность индивидуума при необходимости или наличии интереса.
Помимо этого, существуют мультиаллельные снипы (которых, на самом деле, очень много), правильное генотипирование которых сложно проводить на чипах, так как чип способен различить только две заранее заданные аллели для каждой генетической вариации. Эта проблема особенно остро встает в случае генетических вариаций, которые являются сложными инделами (complex INDELs) и мультиаллельными инделами, где вариабельность вставляемого (или удаляемого) фрагмента приводит к разным клиническим последствиям.
Решением этих проблем генотипирования на чипах является генотипирование с использованием в том числе данных высокопроизводительного секвенирования. Более того, все современные научные исследования и проекты (например, UKBB), которые оказали и продолжают оказывать огромный вклад в понимание и развитие геномики и смежных областей, были сделаны в том числе с использованием высокпроизводительного секвенирования, так как необходиомсть понимания взаимосвязей генетики и фенотипа уже давно вышла за пределы ~600000-1000000 вариаций, доступных на ДНК чипах. Также определение HLA-аллелей и многих других признаков, связанных с гипервариабельными участками ДНК, невозможно на чипе, но возможно при анализе данных высокопроизводительного секвенирования.
Среднее покрытие 30х часто используется как референс в разработках программного обеспечения для генотипирования с использованием данных NGS и рекомендовано к использованию в программных продуктах Broad Institute of MIT and Harvard. Такое значение средней глубины при условии высокого качества выравнивания (mapping quality) достаточно для высокого уровня чувствительности и специфичности определения альтернативных и референсных аллелей наследственных (germline) вариаций.
Исследования соматических вариаций требуют намного более высокого покрытия, так как частота вариантного аллеля может оказаться неопределимо низкой из-за межклеточной вариабельности геномов клеток опухолей. Такие исследования доступны в проекте Атлас Соло.
По поводу сравнения точности генотипирования на чипах и генотипирования с использованием NGS: вы можете ознакомиться с содержимым вот этой статьи, авторы которой проводили аналитическую валидацию генотипирования на чипах с использованием полногеномного и полноэкзомного секвенирования и показали высокую степень идентичности получаемых результатов.
gecube
raw файл с указанием какой аллель в какой позиции (прошу прощения, если не очень точно выразился) — даёт. Или что Вы имеете под исходными данными? График сигнала по каждой из позиций? Или фотографию мирочипа? )))
Я так понимаю, что нет. Полногеномное — это условно, множим Вашу ДНК, режем на кусочки, далее считываем, а потом клеим поверх референсного генома человека. С какой-то вероятностью осознаем, что в позиции Х находится нуклеотид У.
Микрочипирование по идее точнее — т.к. оно проверяет, если, правильно понимаю, конкретное основание в конкретной позиции. Сигнал либо есть, либо его нет.
kablag
чип в этом плане не точнее. В чипе есть синтетическая ДНК, к которой «прилипает» Ваша похожая ДНК, но если такая похожая есть в нескольких участках генома, то может быть ошибка. Просто чипы делают на заранее выбранные точки и они уже проверены на такие ошибки, а при NGS нахождение какой-то новой неизученной и вероятно значимой мутации – это повод для перепроверки другими методами.
gecube
Ну, я себе это так и представлял ) Вопрос в том — насколько вероятна ошибка?
kablag
это нужно смотреть аналитические характеристики конкретного теста. В общем случае, если нашлось что-то критичное и материал остался, можно попробовать перепроверить секвенированием по Сэнгеру – этот метод «золотой стандарт».
solarplexus
Конечно, я буду сравнивать, ибо я в этом не шарю, как и большинство из нас. Сравнительная информация на сайте Атласа предсавлена между двумя продуктами самого Атласа. И выглядит, скажем, как очередная непонятная реклама. То, что вы написали — выглядит более убедительно. Но, опять-таки, требуется дополнительная проверка, т.к. вы, очевидно, заинтересованый человек в продаже этого продукта, а продукт не из дешевых.
gecube
А чего сравнивать? Если читать не профессиональную, а хотя бы любительскую литературу, то Вы узнаете, что даже полногеномное сканирование бывает разное. И какое-нибудь х5 для медицины вообще не годится. Т.к. будут пропуски серьезные и некоторые участки будут нестабильно распознаны. Касательно сравнения с 23andme — это вообще разные технологии. И 23andme делают тест на микрочипа. Таком же как и ancestry, ftdna, myheritage.
kablag, интересный факт — я так понял, что эти микрочипы изначально были заточены на медицину, но потом внезапно оказалось, что они родство между людьми определяют очень хорошо. Почему и стали использоваться для "нахождения своих корней" и "этноотчетов". Что сейчас и является целевой услугой указанных лабораторий. Я попробовал было сам поинтепретировать результаты… ну, например, есть классный сервис Promethease. И был очень разочарован, потому что интерпретации, как Вы сами заметили, очень противоречивые, а риски болезней очень зависят от множества позиций и их комбинаций, в том числе и не включенных в эти обследуемые этими микрочипами. Поэтому мое мнение, что попытка узнать свое здоровье по ним… Это как гадание на кофейной гуще.
kablag
не совсем так, FDA запретил им заниматься выдачей диагнозов или результатов напоминающих диагнозы. Сейчас они могут выдавать результаты по ограниченному списку заболеваний customercare.23andme.com/hc/en-us/articles/211831908-23andMe-and-the-FDA
Кстати у Атласа, как я понимаю, регистрационных удостоверений минздрава на тесты нет и на них врачи ориентироваться не могут. Собственно и назначать не могут, так как в схемах лечения этого нет. Просто пока на это смотрят сквозь пальцы.
NGS в этом плане не сильно лучше. На самом деле заболеваний, при которых определённая мутация приводит к достоверному проявлению не так много. Большая часть изменений или дают какой-то небольшой вклад, или настолько критичны, что являются летальными и такие люди просто не рождаются. Кроме того нужно знать, как информация об этих изменениях повлияет на терапию: и тут тоже, в большинстве случаев – никак.
Поэтому если есть желание и деньги: делайте полногеномное секвенирование – хуже не будет, но чуда ждать не стоит. Только если Вы не слишком мнительный человек и потом не начнёте у себя диагностировать то, чего нет :) А так, то что здоровый образ жизни и ежедневная зарядка улучшает самочувствие понятно и без NGS :) Особенно это забавно в части спортивных достижений: генетика там работает в лучшем случае для спортсменов уровня олимпийской сборной.
gecube
ну, правильно, потому что генетика — это не 100%, есть еще фактор среды. Не рассматриваем строго детерминированные болезни вроде гемофилии или дальтонизма. Но я к тому, что этнокалькуляторы и определение родства появилось, по-моему, существенно раньше, чем запрет FDA на "диагнозы" 23andMe.
Это точно!
Я думаю, что мотивация какая. Если сделать сделать NGS, то результаты остаются с вами, а, следовательно, спустя некоторое время можно будет получить более уточненную интерпретацию, не переделывая тест, т.к. наше понимание того за что какие гены отвечают будет только улучшаться. С другой стороны, спешки никакой нет — по идее с развитием технологий цены должны только падать, а технология становиться более массовой.
asyaaam
Вот очень интересно — есть ли "истории успеха", когда человек сдал полногеномный тест, и это радикально помогло ему улучшить жизнь? Я не говорю про кейсы, когда сирота нашла своих родителей — это уже избитые случаи и не особо интересные. Речь именно про состояние организма.
Benis
У генотека более глубокие тесты на технологии ДНК микрочипа. На распродажах генетический паспорт стоил 10 000р https://www.genotek.ru/genetics/health/
asyaaam Автор
Если мы сравниваем полногеномные исследования, то вы правы. У Генотека оно тоже есть. Только полная расшифровка генома у них стоит 249 000. Кстати, у нас тоже была распродажа на Черную пятницу. Наш генетический тест «Атлас» на ДНК-микрочипе стоил 9900.
Benis
Нет, я имел ввиду именно про тест ДНК по средствам чипа. Сколько вы генов исследуете в тесте ДНК-микрочип?
asyaaam Автор
С помощью ДНК-микрочипа не исследуют гены полностью. Мы исследуем около 660 000 вариантов генов. Подробнее в нашей первой статье.