Сегодня я хочу рассказать о том, как я писал отчеты на R, с чем сталкивался и как решал проблемы, которые возникали по ходу разработки. Отчеты были в формате PDF и запускались из Python в Camunda.
1. Запуск отчетов на R
Первое, с чем пришлось столкнуться - это желание заказчика запускать генерацию отчетов на R из Python. Точнее из Python запускали subprocess.Popen, который запускал R script. R script, в свою очередь, запускал одну из функций генерации того или иного отчета, которая была частью нашего проекта, написанного как пакет на R. Разницу между пакетом и обычными скриптами на R я описал тут - ссылка. Можно сразу дергать функции R и мой коллега пытался переписать вызов отчетов из Python, но то ли из-за проблем с окружением на сервере, то ли из-за Camunda, в которой это все запускалось, такой способ отлично работал локально, но не работал на нашем боевом окружении. Минусом же вызова R через subprocess.Popen является то, что Python вызывает R скрипт через shell, а уже внутри этого скрипта дергается функция пакета R.

Параметры для отчета тоже приходится передавать через shell и уже в R script файле так же их читать как будь то их передают через командную строку.
Часть кода на Python:
parameters = {
"param1": param1,
"param2": param2,
"param3": param3
}
...
command_list = ["Rscript", path_to_r_script] + [f"--{key}={value}" for key, value in parameters.items()]
with subprocess.Popen(
command_list, stdout=subprocess.PIPE, stderr=subprocess.PIPE, cwd=work_dir
) as sub_prc_response:
stdout, stderr = sub_prc_response.communicate()
return_code = sub_prc_response.returncode
Script на R, где ourpackage название нашего пакета:
options(warn=-1)
library(ourpackage)
library(optparse)
options = list(
make_option("--param1", help = "first parameter"),
make_option("--param2", help = "second parameter"),
make_option("--param3", help = "third parameter"),
)
cli_arguments = parse_args(OptionParser(option_list = options))
generate_report(param1 = cli_arguments$param1,
param2 = cli_arguments$param2,
param3 = cli_arguments$param3)
Возникает вопрос как это все тестировать и отлаживать. У нас часть кода на Python, а часть на R. Можно использовать JupiterHub, но наши администраторы настроили его так, что код на Python мы могли вызвать, а вот код на R нет. Мотив был - экономия памяти и ресурсов. В итоге тестировали через RStudio. В ней можно создать файл типа Python:
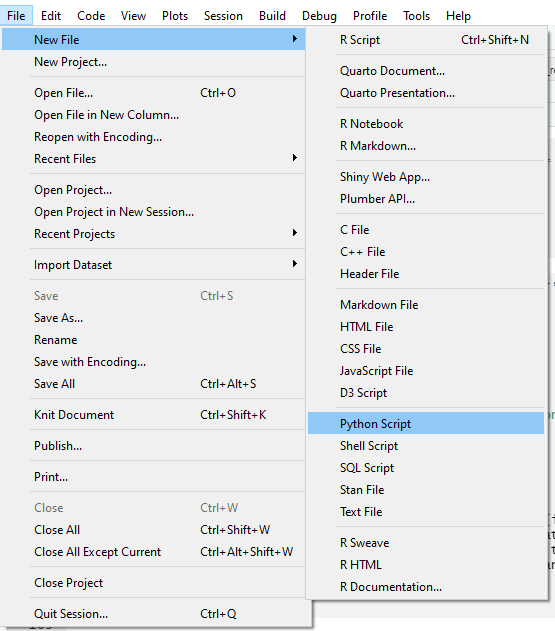
И уже внутри этого файла вызвать функцию python, которая, в свою очередь, вызовет генерацию отчета на R.
Пример вызова:
from our_project.workflow.generate_report_one import report_one_executor
task = {
"variables": {
"param1": {"value": "value1"},
"param2": {"value":"value2"},
"param3": {"value":"value3"}
}
}
report_one_executor(task)Чтобы проверить и отладить сам R script файл, его можно запустить из Terminal в RStudio. Для этого его вам придется положить внутрь вашего проекта, хотя бы на время теста и отладки. Можно держать его и вне проекта, если вам это позволяют настройки окружения, в котором вы работаете. У меня не было такой возможности, поэтому я размещал script файл в подпапке внутри R папки проекта, так как в пакет содержимое подпапок не попадает и, конечно же, не коммитил эту попдпапку в репозиторий. Команду запуска отчета через script надо писать в Terminal. Это вкладка на нижней панели в RStudio.
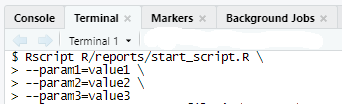
Пример запуска из shell:
Rscript R/reports/start_script.R \
--param1=value1 \
--param2=value2 \
--param3=value3В коде, для генерации отчетов, надо указать путь к RMD файлу, на основе которого будет строиться отчет. Пример функции, которая строит отчет ниже, при условии что RMD файл лежит в inst/reports:
output_rmd <-
function(output_report_file = "test",
output_report_dir = NULL,
output_type = "DOCX",
params_list = NULL) {
rmd_file = "test-report.Rmd"
report_rmd_folder <- system.file("reports", package = "myRTestPrj")
rmd_input <- paste(report_rmd_folder, rmd_file, sep = "/")
rmarkdown::render(
rmd_input,
output_file = output_report_file,
output_dir = output_report_dir,
params = params_list
)
}2. Конфликт библиотек
При сборке пакета часто можно увидеть сообщения типа:
replacing previous import ‘flextable::rotate’ by ‘ggpubr::rotate’ when loading ‘our_project’В этом сообщении говорится, что если явно для функции rotate не указать название библиотеки, то будет вызываться ggpubr::rotate. Это происходит из-за того, что в разных пакетах есть функции с одинаковыми именами. Чтобы эти сообщения не выводились можно указать какие функции из каких библиотек должны грузиться, а какие нет. У нас в проекте для этого в папке R был создан файл global_imports_pkg.R в котором прописывались именно те функции, которые нам нужны.
# Global libraries
#' @importFrom data.table dcast melt rbindlist
#' @import ggplot2
#' @rawNamespace import(ggpubr, except=c(font))
#' @importFrom grDevices rgb colorRampPalette
#' @rawNamespace import(huxtable, except=c(add_rownames,theme_grey))
NULLНо это потенциально может привести к другой, более коварной ошибке. Пусть у вас в global_imports_pkg.R написанно так:
#' @importFrom data.table melt rbindlistА в коде вы вызываете функцию dcast, которой нет в строчке выше. Есл вы запустете функцию из под RStudio, просто написав в консоли:
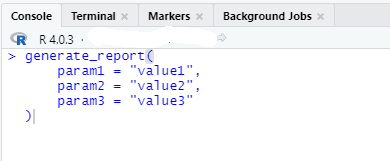
У вас все будет работать, так как локально вы себе инсталлировали весь пакет data.table, а вот при попытке вызвать эту же функцию из Python, у вас будет ошибка, так как в проекте у вас написанно, что в наш пакет нужно подгрузить только 2 функции из data.table - это melt и rbindlist. Ту же ошибку вы можете получить на сервере, хотя локально все будет работать.
Похожее поведение можно так же получить, если забыть добавить используемый вами сторонний пакет в файл DESCRIPTION:
...
Imports:
highcharter,
statnet.common,
hablar
Suggests:
devtools,
...При запкуске локально в RStudio все будет работать, так как вы явно установили этот пакет локально, но вот при вызове вашего пакета сторонним кодом или средой вы получите ошибку.
3. Утечка памяти в Rmarkdown::render
При генерации больших отчеов в PDF формате возникает ошибка, если посмотреть в терминале на процесс генерации шаблона, то видно как он отъедает всю доступную память и падает. Для решения этой проблемы мы резали отчет на части и потом их склеивали. Для этого использовали qpdf::pdf_combine. Проблема с утечкой памяти обсуждается как минимум тут. Какого-то более простого решения найти не получилось, хотя сил и времени было потраченно немало.
4. Latex
Для форматирования pdf отчетов использовали Latex. Начальный шаблон для отчета был взят тут.
Код на R для генерации отчета с шаблоном Latex:
rmarkdown::render(
rmd_input,
output_format = "pdf_document",
output_file = output_file,
output_dir = output_report_dir,
params = params,
output_options = list(
template = paste0(report_rmd_folder,
"/assets/custom.latex"),
clean = TRUE
)
)Так же непосредственно в шапку RMD файла можно добавить команды для форматирования документа:
---
title:
header-includes:
\usepackage{graphicx}
\usepackage{fancyhdr}
\pagestyle{fancy}
\color{ecbdarkblue}
\color{ecbbgr}
% Head
\fancyhead[C]{}
\fancyhead[L]{}
\fancyhead[R]{}
% Remove header line
\renewcommand{\headrulewidth}{0pt}
% Page margins
\usepackage{geometry}
\geometry{
a4paper,
left=30px,
top=10mm,
headsep=5mm,
right=50px
}
% Titles
\usepackage{titlesec}
\titlespacing\section{0mm}{7pt plus 4pt minus 2pt}{28pt plus 2pt minus 2pt}
\titlespacing\subsection{6mm}{-7pt plus 4pt minus 2pt}{0pt plus 2pt minus 2pt}
\titlespacing\subsubsection{6mm}{12pt plus 4pt minus 2pt}{0pt plus 2pt minus 2pt}
% Section and subsection titles
\sectionfont{\fontfamily{phv}\selectfont\LARGE\bfseries\color{ecbdarkblue}}
\subsectionfont{\fontfamily{phv}\selectfont\normalsize\mdseries\color{white}}
\subsubsectionfont{\fontfamily{phv}\selectfont\scriptsize\mdseries\color{white}}
\color{black}
% Line spacing
\usepackage{setspace}
\setstretch{0.6}
% Section color
\usepackage{xcolor}
\usepackage{framed}
\colorlet{shadecolor}{ecbbgr}
% Footer
\fancyfootoffset[R]{-2mm}
\renewcommand{\footrulewidth}{0.4pt}\color{ecbdarkblue}
\rfoot{\vspace{0.5mm}\Large\color{ecbdarkblue}\colorbox{ecbbgr}{ S \color{white}\thepage}}
\cfoot{\vspace{0.01mm}\scriptsize\bfseries\hspace{420px} ECB \\ \scriptsize\bfseries\color{black}\hspace{390px} DG-S/EA/GBS\\ \scriptsize\bfseries\color{ecbdarkblue}\hspace{395px} `r format(Sys.Date(), format="%d %b %Y")`}
\lfoot{}
output:
pdf_document:
keep_md: no
keep_tex: no
latex_engine: pdflatex
number_sections: false
---
```{r setup, include=FALSE}
# some code here...
```Подробно команды Latex я не буду разбирать, так как достаточно посмотреть на код в тех или иных блоках и понять, как надо его поправить, чтобы изменить форматирование. Во многом я опирался на этот сайт когда форматировал документ.
Можно так же гибко настроить какие ошибки и сообщения будут писаться в отчет. Это делается командой:
knitr::opts_chunk$set(echo = FALSE,
warning = FALSE,
message = FALSE,
error = TRUE)Можно эти настройки так же включать и отключать в шапке chunk:
```{r our_chunk, eval = TRUE, results='asis', echo=FALSE, warning=FALSE, message=FALSE, error=TRUE}5. Заглушки для внешних библиотек при написании Unit тестов
Для вызова вункций из внешних библиотек, написанных на Python мы использовали reticulate.
Пример вызова внешней функции:
get_python_func <- function(param1) {
pm <- reticulate::import("our_project.core.python_functions")
return(pm$get_python_func(param1))
}Тест - заглушка для такой функции:
test_that("get_python_func", {
stub(get_python_func, "reticulate::import", list(get_python_func = function(...) TRUE))
res <- get_python_func(param1 = "value1")
expect_equal(res, TRUE)
})6. Отладка кода
При отладке кода есть возможность скопировать значения внутренних переменных в глобальную область памяти и напрямую вызвать нужную нам часть кода с определенными значениями переменных для этого кода.

На картинке выше в верхнем правом углу в поле Data есть переменная df. Чтобы скопировать её в глобальную область надо написать в Console:
assign("df", value = df, envir = .GlobalEnv)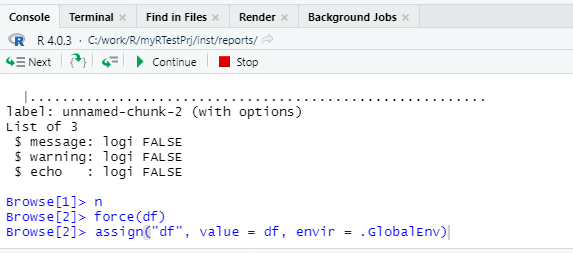
И выполнить эту команду - нажать ENTER.
После этого можно выделить тот код который мы хотим протестировать с этой локальной переменной и запустить его - CTRL+ENTER.
Дело в том, что отладка в RMD файле не работает и вся логика по возможности выносится в функции, которые находятся в файлах *.R. К этому ещё добавляется не высокая скорость генерации отчетов из RMD файла. В таких условиях каждый раз делать DEBUG не удобно. Работает все медленно и участки кода в RMD не отладить. А с установкой значений переменных в глобально области можно проверить любой участок кода, даже если он находится непосредственно в RMD файле.
7. Использование классов в проекте
Мы использовали ReferenceClasses, решив, что они больше всего нам подходят. Правда, их невозможно отладить инструментами RStudio. Но есть стандартные функции отладки, с помощью которых сама отладка возможна:
s$trace(class_method, browser)
s$untrace(class_method, browser)
debug(s$class_method)Где s - это экземпляр класса, а class_method - это метод класса. Но он оказался не очень удобным и мы отлаживали с помощью переменных в глобальной области видимости.
8. Прочие нюансы проекта
Библиотека для работы с данными для отчетов - dplyr;
Чтобы создать список из входящих параметров функции использовали следующий код:
# create list with input parameters
params <- c(as.list(environment()))Удалить параметры или параметр из глобального окружения можно с помощью команды rm;
Статический анализатор кода - lintr;
Для автоматической записи документации в Confluence использовали библиотеку conflr;
Таблицы в отчетах делали с помощью felxtable.
Заключение
Это статья - памятка, в которой я описал ключевые вещи проекта, которые я не хотел бы забыть и которые, на мой взгляд, были бы полезны тем, кто работает с аналогичными проектами на языке R. Возможно что-то можно улучшить, а чего-то избежать, но не хотелось бы "Изобретать велосипед заново", начиная новый проект на R. В заключении хотел бы пожелать всем успехов, а также хороших и интересных проектов.

nonickname227
Не пробовали подключать R к питону через rpy2? Наверно пробовали, а чем тогда не устроило?
alex_29 Автор
Пробовали, я же писал про это. Локально работало, а на окружении нет. Скорее всего проблема с окружением, но я описывал то что у нас работало, чтобы достоверность соблюсти.
nonickname227
Теперь более понятно. Просто rpy2 не обязательно только с Jupyter использовать. Можно же было бы просто использовать импорт пакетов R и непосредственно использовать функции и другие объекты из R в питоне. Я про такой вариант поинтересовался...