
Доброго времени суток всем уважаемым хабровчанам. Меня зовут Алексей, и в данный момент я работаю в “Филиале №11 ООО "ОЦРВ" Сириус”. В этой статье я хотел бы поделиться с вами опытом своего участия в соревновании на достаточно известной соревновательной платформе по Data Science’у - Kaggle. Перейдем к сути.
Описание соревнования.
Соревнование называлось RSNA 2022 Cervical Spine Fracture Detection и проходило с 28 июля по 28 октября 2022 года. Для его организации Радиологическое общество Северной Америки (RSNA) объединилась с Американским обществом нейрорадиологии (ASNR) и Американским обществом радиологии позвоночника (ASSR), дабы выяснить, возможно ли при помощи искусственного интеллекта обнаружить переломы в шейном отделе позвоночника.
Для создания эталонного набора данных была собрана рабочая группа, которой удалось собрать 3000 КТ исследований. Радиологи из ASNR и ASSR сделали разметку на уровне каждого изображения в исследовании (на аксиальном срезе), указав наличие, номера позвонков и местоположение любых переломов шейного отдела позвоночника.
Данные были представлены в следующем виде:
Папка train_images, содержащая в себе 2019 исследований пациентов. Каждое исследование содержало в себе ~200-600 .dcm файлов, представляющих собой аксиальные проекции шейного отдела. Саму структуру DICOM’a разберем отдельно.
Папка test_images, содержащая всего данные о 3 пациентах для отладки. Скрытый тест предполагал ~1500 исследований, прямого доступа к которому не было.
Табличка train.csv, содержащая в себе бинарную отметку, свидетельствующую о наличии переломов для каждого из шейных позвонков (С1-С7) + бинарная метка о том, есть ли перелом шейного отдела у пациента вообще.

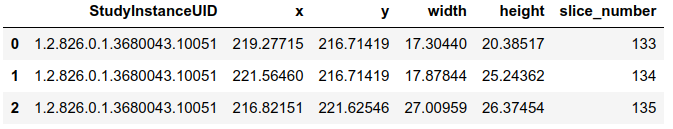
train_bounding_boxes.csv - информация о локализации переломов для 235 исследований. Как правило, на одном снимке не больше 1 перелома.
Папка segmentations с сегментационной разметкой для позвонков (NIFTI файлы с расширением .nii). Часть получена при помощи сегментационной модели 3D-UNET, доработанной впоследствии радиологами. При этом значения сегментационных меток варьируются от 1 до 7 для С1 до С7 (семь шейных позвонков), от 8 до 19 для Т1 до Т12 (двенадцать грудных позвонков, расположенных в центре верхней и средней части спины) и 0 для всего остального. Поскольку задача сформулирована для шейного отдела позвоночника, все сканы имеют метки от C1 до C7, но не все - грудные метки. Стоит также отметить, что NIFTI содержат разметку, сделанную в сагиттальной плоскости, в то время как файлы DICOM представлены в аксиальной плоскости. Это важно, поскольку необходимо их верно сматчить. При неправильном матчинге есть вероятность перевернуть маску по оси Z и отразить ее по оси X.
test.csv, который, по факту, содержал лишь полезную информацию об id исследования, для которого нужно делать предсказание.

sample_submission.csv - пример формата, в котором ожидается запись предсказаний.

Всего ~ 700 тыс. файлов общим весом ~350 Гб.
Метрика.
Нам необходимо предсказать вероятность перелома для каждого из семи шейных позвонков (C1-C7), а также общую вероятность любых переломов шейного отдела позвоночника (patient_overall). Это означает, что для каждого исследования у нас будет 8 строк с предсказаниями. При этом переломы основания черепа, грудного отдела позвоночника, ребер и ключиц в расчет не принимаются. Метрика оценки качества решений представляет собой взвешенную логарифмическую потерю с несколькими метками (weighted multi-label logarithmic loss), усредненную по всем пациентам:

где i - номер исследования, а j - метка в рамках исследования.
Веса (w_j) при этом были следующими: 1 - если перелома конкретного позвонка нет; 2 - если перелом у конкретного позвонка имеется; 7 - если в исследовании переломов не имеется; 14 - если перелом в исследовании имеется.
Пример вычисления метрики представлен на картинке, взятой отсюда:

Краткий обзор данных.
Как уже писалось выше, .dcm файлы представляли собой аксиальный срез <1 мм толщиной, внутри которых присутствует как визуальная информация (пиксели), так и некоторые метаданные. Полный набор метаинформации представлен на скриншоте ниже.

Описание метаданных можно найти по ссылке, правда на английском языке. Распишу тут некоторые из них, которые мне кажутся важными для понимания.
Slice Thickness - толщина среза в мм.
Instance Number - порядковый номер среза.
Image Position - координаты x, y, z верхнего левого угла (центр первого переданного вокселя) изображения в мм.
Image Orientation - Значения направляющих косинусов для первой строки и первого столбца относительно положения пациента.
Pixel Spacing - Физическое расстояние между центрами двух смежных пикселей, причем первое число пары характеризует расстояние в мм между соседними “строками” пикселей, а второе -расстояние между соседними столбцами.
Rescale Intercept - Слагаемое, которое можно использовать для перевода численных значений в юниты Хаунсфилда (HU).
Rescale Slope - множитель для возможного перевода значений в HU.
Итоговая формула - HU = Rescale_Slope * Stored_Value + Rescale_Intercept
Описание следующих двух значений носит несколько общих характер, псевдокод преобразований исходных значений с их использованием можно найти в описании стандарта DICOM’а по ссылке выше.
Window Center - значение центра окна, к которому будет приведено изображение для сжатия представления.
Window Width - ширина окна “сжатия”.
Оконные значения используются уже после преобразований с Rescale Slope и Rescale Intercept. Значения меньше, чем WC-WW/2 присваивается минимальное значение в изображении, а больше, чем WC+WW/2 - максимальному. Но это упрощенный вариант. Более сложный, с преобразованиями из VOI LUT, описан в стандарте.
Что же насчет визуальных данных? А вот и они.

Здесь уже применены преобразования из VOI LUT таблицы (при использования библиотеки pydicom конвертация может быть осуществлена вызовом функции apply_voi_lut).
Данные по сегментированным позвонкам были закодированы в формат .nii (NIFTI), с которым прекрасно работает такая библиотека в python как nibabel. Каждый файл содержит сегментационные маски для всех срезов, однако информация по сегментации представлена в сагиттальной плоскости, поэтому для матчинга .dcm с масками нужно было делать промежуточное преобразование. В маски также была закодирована информация о том, какой позвонок она отображает. Благодаря этому можно было достать информацию для классификации среза по типу представленного позвонка. К сожалению, такая информация была предоставлена только для 87 пациентов, что составляет всего ~4% от всех пациентов.

Также, как я уже писал выше, в соревновании была представлена информация по локализации переломов для 235 пациентов, а это около 12% от общего числа пациентов. Примеры их визуализации можете видеть ниже.

Визуализация любезно взята из данного публичного ноутбука. В нем вы также сможете найти аналитические графики для предоставленных организаторами соревнования данных.
Приступим к делу.
На соревнование у меня был всего месяц, а из ресурсов была доступна одна А100. Это не очень-то густо для такого набора данных, но и куда лучше того, что сейчас нам предоставляет сама платформа Kaggle (~36 GPU-часов в неделю на P100). Ограничение по времени и ресурсам сыграло важную роль при выборе гипотез, которые были протестированы в первую очередь.
Итак, полный список гипотез, которые пришли в голову, были следующими:
Классификация аксиальных срезов картиночной сетью с двумя ветками на определение позвонка + определение перелома с последующей постобработкой (ибо на выходе от нас все же ожидается ответ по пациенту, а не по срезу).
Отдельная сеть на классификацию срезов по типу позвонка + отдельная сеть на object detection + постпроцессинг.
Подход №2, но без отдельной сети на классификацию, а просто с отдельной классификационной веткой.
Предыдущий подход, но с предварительной сегментацией отдельной сетью позвонка и вырезанием изображения по его описанному вокруг маски прямоугольнику.
End-2-end классификация 3д снимка КТ каждого пациента.
End-2-end object detection (либо с веткой на классификацию, либо с переформатированной разметкой от сегментации).
Стоит также отметить, что для успешного сабмита инференс для ~1500 пациентов должен быть завершен за 9 GPU-часов на той же P100. Если предположить, что кол-во срезов для одного пациента в среднем равно 400, то обнаружится, что эти данные должны обработаться за ~22 секунды, а один срез - за ~54 мс.
В силу ресурсных ограничений было принято решение сосредоточиться на первых двух подходах. Итак, приступим к описанию реализации и результатам.
Попытка номер раз.
Собственно, после анализа сообщений на форуме и публичных ноутбуков, было замечено, что возможно построить классификатор позвонков по метаданным, причем с достаточно высоким качеством (~0.88 по точности на кросс валидации по 5 фолдам). Именно такой классификатор я и принял решение создать как бейзлайн перед тем, как скармливать срезы детектору.
В качестве признаков были отобраны следующие пять: x, y, z координаты из Image Position, толщина среза из Slice Thickness и нормализованное значение номера среза (отношение порядкового числа к максимальному в рамках пациента). Из моделей были запущены “с ноги” Catboost и RandomForest из легендарного scikit-learn. Итоговые результаты оказались сопоставимыми, классификатор неплохо усваивал монотонность возрастания порядка позвонков.
В качестве детектора изначально был выбран YOLOv7, ибо как бы SotA в соотношении скорость и качество. Однако в процессе обучения обнаружилось, что обучение очень нестабильно: из 5-6 итераций только 1 итерация имела mAP (Mean Average Precision) отличный от нуля. Анализ issues на гитхабе показал, что с такой проблемой некоторые пользователи все же сталкивались. Однако те предложения, что были там представлены, не изменили ситуацию для меня. Тогда я решил перейти на более надежный с инженерной точки зрения фреймворк - YOLOv5. О нем уже писали на хабре, например, вот тут. С точки зрения подготовки входных данных, из .dcm был экспортирован набор пикселей (512х512), с предварительным переводом в HU с отсечением по порогам от 300 до 1900 единиц (такие значения характерны для костной ткани). В качестве входного размера изображения использовались значения от 512 до 1280, аугментации стандартные, согласно уже имеющимся в репозитории конфигам. В итоге, максимальное значения mAP при пороге IoU в 0.5, которое удалось достичь, было равно 0.235.

Визуальный анализ предсказаний показал, что изрядная доля ошибок приходилась вне области интереса (вне позвонка).

Казалось бы, стоит добавить кроп под позвонку, чтобы исключить лишние реагирования на костную ткань вне области интересов. Однако эта часть потребовала бы еще дополнительных затрат временных и вычислительных ресурсов. А поскольку время таяло прямо на глазах, было принято решение взять публичный бейзлайн и что-то сделать с ним. Ах да, стоит перед этим еще упомянуть постобработку, ибо от нас все же ожидают вероятности перелома позвонков по пациентам, а не по срезам.
Алгоритм можно описать следующими шагами:
Для каждого среза находим кол-во bounding box’ов и тип(ы) позвонков, что на нем изображены. Делим кол-во переломов на число найденных позвонков на изображении (как правило, не больше 2).
Сохраняем полученные вероятности в словарь со списками, где ключом будет выступать номер позвонка.
Задаем параметр значений найденных переломов для пациента, после которого считается, что вероятность перелома позвонка равна 1 (в своем эксперименте я использовал значение, равное 50).
Для каждого позвонка суммируем значения в списке и делим на параметр, указанный выше и ограничиваем максимальным числом, равным 1.
На этом все. Результат на платформе следующий: публичный лидерборд - 0.75, приватный - 0.91. Стоит также отметить, что сабмит вероятностей 0.05 для каждого пациента по всем позвонкам давал скор в 0.66 и 0.76 соответственно. Это оказалось куда хуже лучшего публичного решения, о котором поговорим далее.
Тюним публичный бейзлайн.
Прежде чем описать процесс тюнинга, стоит сказать, что из себя представлял бейзлайн.

Данные: нормализованные min-max нормализацией картинки в рамках одного среза, сжатые до размера 384х384 при помощи билинейной интерполяции. Затем использовалась нормализация по статистикам ImageNet’a. Что же касается разметки, то в качестве меток использовались два вектора, один из которых содержал в себе данные по переломам для каждого из 7 позвонков, а второй - классификационная multi-label разметка по тому, какие позвонки изображены на срезе, полученная из сегментационной разметки. Вектор с переломами был поэлементно умножен на вектор позвонков, чтобы разметка по переломам была представлена только для видимых на срезе позвонков.
Модель: EfficentNetv2-s из пакета torchvision, с замененным финальным слоем на 2 линейных с кол-вом нейронов, равным 7. Сама сеть вышла в середине 2021 года, став на тот момент тем, что именуют SotA (State-of-the-art). Помимо высокой точности данный тип сетей характеризует и относительно высокая скорость как обучения, так и инференса. Основными нововведениями относительно первой версии выглядели так:
Обучение на картинках меньшего размера, но с большим батчем.
Замена MBConv блоков на Fused-MBConv на первых стадиях (внутри заменили Depthwise convolution и последующее расширение сверткой 1х1 на свертку 3х3).
Параметры расширения сети от базовой к более сложным сетям вместо фиксированных были заменены на адаптивные, полученные в процессе оптимизации.
Прогрессивное обучение (progressive learning), в рамках которого размер картинки постепенно увеличивался наряду с регуляризацией (в статье описаны 3 главных параметра в рамках регуляризации: Dropout, RandAugment и Mixup).
Функция потерь: Поскольку модель состояла из двух веток, то и функция потерь была составная. Ветка на определение типа позвонка использовала стандартную бинарную перекрестную энтропию. Ветка на определение перелома позвонков использовала взвешенную бинарную перекрестную энтропию. Что же из себя представляли веса? По сути, это попытка инкорпорировать в себя веса из метрики оценки качества решения. Т.е была группа позитивных весов (14, 2, 2, 2, 2, 2, 2, 2) и негативных (7, 1, 1, 1, 1, 1, 1, 1), которые и были мультипликаторами для соответствующих позиций в результатах выдачи бинарной кросс-энтропии. Затем лосс нормировался по совокупной сумме весов и суммировался для каждого пациента. Итоговый лосс - это удвоенная сумма функции потерь на ветке с определением переломов и функции потерь с ветки определения типа позвонков.
Постобработка: Как можно отметить, классификация проводилась по слайсам, а предсказания необходимо делать по пациентам. Переход проходил по следующему алгоритму:
Сначала мы поэлементно умножаем предсказания вероятностей переломов позвонков и их номеров. Здесь предсказания номера позвонка является весом, позволяющим отфильтровать неточные предсказания, когда у нас, например, предсказан перелом третьего позвонка, но на слайсе представлены только первые два. Тогда небольшой вес на определении третьего позвонка позволит снизить степень уверенности в том, что третий позвонок все же имеет перелом.
Взвешенные предсказания переломов усредняются по всем срезам внутри каждого исследования.
Осталось определить общую вероятность того, что пациент имеет перелом в шейном отделе. Для этого мы сформулируем задачу следующим образом: имея вероятности переломов каждого из семи позвонков, нам нужно найти вероятность того, сломан ли хотя бы один из позвонков. Более формально, решение будет выглядеть следующим образом:
Режим обучения: Обучение проходило в течение одной неполной эпохи (а если быть точнее, то 4к батчей при размере батча в 32 - это приблизительно 128к срезов при наличии сегментационной разметки для ~ 700к срезов на 87 кейсах, т.е 20% от общего кол-ва масок). Оптимизатор - Адам, вшитый в one cycle политику изменения шага обучения.
Данное решение, усредненное по 5 фолдам дает 0.47 на паблике и 0.52 на привате соответственно.
Итак, перейдем к улучшениям.
Аугментации.
Первое, что было сделано - это добавлены аугментации вместо тех, что использовались при обучении на ImageNet’e. Среди них были следующие: HorizontalFlip, RandomBrightnessContrast, ShiftScaleRotate, что-то одно из списка геометрических преобразований (ElasticTransform, GridDistortion, OpticalDistortion), Resize (512->768), RandomCrop(512). На инференсе использовался только ресайз и CenterCrop(512).
Использование этих аугментаций вместе со сменой модели на m-версию efficientnet’a дало после усреднения по 3 фолдам 0.46 на паблике и 0.49 на привате.
Постобработка №1 (неудачная).
Была мысль, а что если мы немножко подчистим вероятности, срезав вероятности 0.05 до нуля и округлив вероятности выше 0.95 до 1. Результат оказался неудовлетворительным. Примененный к бейзлайну, метод солидно ухудшил показатели до 0.59 на паблике и 0.82 на привате. Увеличение порогов лишь ухудшало и так не очень-то достойный результат.
Увеличение длительности тренировки.
Увеличение длительности тренировки для модели effnetv2-s до одной полноценной эпохи с тем же набором аугментаций при усреднении по 5 фолдам дало 0.41 на паблике и 0.46 на привате.
SSE блок.
Идея получена из решения по одному из предыдущих соревнований.

По сути, сеть режется после последнего MBConv блока, после чего сверху вешаются 2 ветки (на переломы и позвонки), где идет sSE блок + Dense + ReLU + Dropout + последний линейный с софтмаксом. Также сделан скип с определения позвонков на переломы. Полученный результат для m-модели effnetv2 по 5 фолдам - 0.45 на паблике и 0.49 на привате. Т.е версия с блоком немного лучше на паблике, а на привате +- такой же результат. А вот s-версия показала результаты похуже, чем без блока - 0.42 на паблике и 0.47 на привате. При этом увеличение длительности тренировки до 3 эпох лишь ухудшило результаты (~0.6).
Увеличение картинки без изменения размера кропа.
Неудачная попытка сконцентрировать внимание на центральной части картинки, где находится под окном. Картинка была увеличена до 1024 с кропом в 512. Правда, по-видимому, случайный кроп иногда не захватывал целевую область, что и повлияло на ухудшение качества. EfficientNetv2-s с sSE на 5 фолдах выдал 0.44 на паблике и 0.49 на привате.
Предобработка по порогам HU.
На этом этапе была изменена предобработка: вместо стандартной min-max нормализации в рамках картинки производился перевод в значений пикселей в HU и отсечение по порогам в 300 слева и 1900 справа. Данное изменение для лучшей модели на тот момент сказалось не лучшим образом. Скор ухудшился примерно на одну сотую.
Постобработка №2 (тоже не сильно удачная).

В итоге, выбор пал на максимальное значение, среднее и медиану, а сглаживание проходило скользящим окном размером шириной в 20 (эмпирически подобранный параметр). Но последние 2 характеристики, казалось, не совсем корректно использовать на всем распределении, а лишь на той ее части, что образует фигуру. Поэтому было принято решение использовать только те значения в распределении, что выше 0.05. Сейчас уже кажется, что это достаточно высокая величина, и стоило бы понизить порог хотя бы на 1 порядок. Итоговые результаты можно будет увидеть в финальной таблице, но, спойлер, все плохо.
Другая сеть.
В качестве альтернативной архитектуры было принято решение попробовать ResNeXt50 (где-то она проскакивала в обсуждениях среди тех, кто был в топе). Результат по 5 фолдам с sSE блоком и новым препроцессингом - 0.43 на паблике и 0.48 на привате.
Увеличение кропа вкупе с общим размером изображения.
Одна из гипотез была связана с тем, что кроп области интересов слишком мал для того, чтобы получить более детальную информацию о переломе, особенно если учитывать дополнительные инструменты для аккумулирования этой информации в виде sSE блока. Поэтому было принято решение увеличить размер картинки до 1024 с кропом в 768. Данное улучшение вместе с sSE блоком и новым препроцессингом показало наилучший результат.
Итого.
Ниже представлена таблица с результатами, по которой можно увидеть 3 лучших эксперимента.

В принципе, это все, что удалось сделать за отведенное время, а если точнее, то финальные пару недель. Учитывая то, что один фолд для лучшего результата обучался около 5 часов, кажется, что получился неплохой результат. Ничего нового, к сожалению, привнести в мир науки не удалось, но свое серебро я забрал, попав в топ-4% лучших решений.
А теперь давайте же узнаем, чем примечательны оказались решения других конкурсантов.
Обзор лучших решений.
И напоследок я постараюсь привести саммари трех наиболее интересных для меня решений, которые вошли в финальную десятку. Начнем с мест пониже и закончим первым местом.
7 место.
Полное описание решения можно найти здесь.

Основу данного решения составляло использование двух 3D-Unet’ов для сегментации. Правда, в первом случае сегментировались целевые позвонки (используя 87 кейсов из трейна + ~200 кейсов из внешних датасетов), а во втором случае - сами переломы. Спросите, откуда же разметка для сегментации переломов? Здесь использовалось два подхода для генерации данных, на основе псевдолейблинга. Первый способ основан на написании кастомной аугментации, которая создает видимость перелома на здоровом позвонка, делая маску, заливая ее с запасом от нарисованного перелома. Второй способ попроще - это залитый bounding box с переломами из представленной разметки для 235 пациентов, но с немного поправленной маской, убирая лишнюю заливку за пределами кости.

Обучение второй модели шло в несколько этапов, во время которых маски обрабатывались при помощи дополнительной постобработки (например, можно было убирать маски переломов, предсказания которых противоречили классификационной разметке), после чего снова подавались на обучение.
Полученные результаты из двух сетей конкатенируются и подаются на вход небольшой 3д сверточной сети, которая уже и выдает финальный результат.
Стоит еще отметить то, что в качестве финальной постобработки на кросс-валидации были подобраны минимальные и максимальные пороги для вероятностей, т.к logloss чувствителен к крайним значениям.
4 место.
Полное описание находится тут.
Основной идеей здесь служило использование двустадийного обучения. На первой стадии обучался мультилейбл 3д сегментатор позвонков наподобие U-Net’a, который в качестве бэкбона использовал одну из топовых cnn для action classification’a, а на декодере (2+1D) свертки. При этом использовались 2д аугментации, которые повторялись при помощи ReplayCompose’a. Во второй стадии использовался 3д классификатор, который на вход принимал набор слайсов, которые были организованы в трехканальный блок (слайс+слайс+сегментационная маска из первой стадии, перекодированная в 2д формат целочисленным кодированием). На выходе был вектор из 8 значений (7 на позвонки + общий). Если целевой позвонок был виден менее чем на 30%, значение для перелома позвонка обнулялось, а общий скор был пропорционально пересчитан. Остальные подробности и код доступны по ссылке в начале блока.
1 место.
Решение можно найти по ссылке.
Как и предыдущее решение, оно состоит из двух стадий. Первая стадия - привычная для нас 3д сегментация шейных позвонков. Массивы входных данных были собраны из 128 слайсов и сжаты до размера 128х128. Затем обученная модель была использована для предсказания позвонков на 2 тысячах пациентов с классификационной разметкой.

Теперь полученные маски можно использовать для того, чтобы подготовить данные для второй стадии. Подготовка выглядит следующим образом: для каждого целевого позвонка нужно вытащить 15 равномерно распределенных по z-оси слайсов. Для каждого из этих слайсов нужно захватить по 2 слайса слева и справа, чтобы сформировать 5-канальное изображение. Также в 6 канал записывается сегментационная маска, дабы минимизировать эффект того, что на изображении будет присутствовать отличный от целевого позвонок.
Теперь у нас есть для каждого пациента 15 3д изображений для каждого позвонка и 7х15=105 изображений для всего пациента. Самый простой способ - использовать 3д сверточную сеть для классификации, но у автора данного решения такой подход не оправдал себя. Поэтому я решил использовать подход с 2д сверточной сеткой + lstm.
Всего он привел 2 типа моделей. Первая модель принимала на вход 15 изображений для каждого позвонка, извлекала 512-размерные признаки и подавала их в lstm.
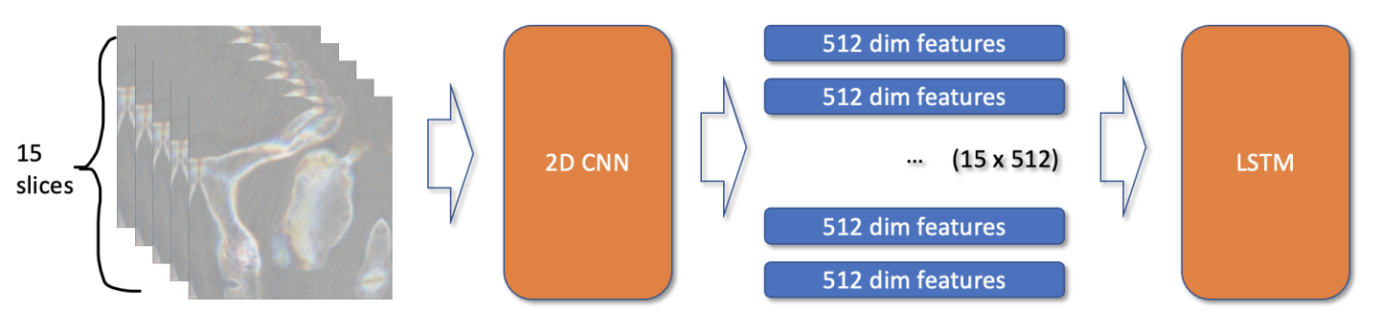
Минусы такого подхода в том, что здесь у нас нет возможности в end-2-end формате делать предсказания для пациента. Поэтому автор доработал модель, подавая 105 изображений по всем позвонкам для конкретного пациента. Из минусов такого подхода - это огромные требования к gpu-памяти, ибо для того, чтобы держать в памяти 105 6-канальных изображений нужны солидные ресурсы (у автора было 2 A6000, у которых на борту в сумме 96 Гб видеопамяти).
Собственно, на этом все. Если будут какие-то вопросы или пожелания - добро пожаловать в комментарии. До новых встреч.
