
У вас продакшн нейронные сети, терабайты данных? Вам хочется понять, как работает нейронная сеть, но на таком объеме это сложно сделать? Сложно, но можно. Мы в NtechLab находимся именно в той ситуации, когда данных так много, что привычные инструменты интроспекции нейронных сетей становятся не информативны или вовсе не запускаются. У нас нет привычной разметки для обучения атрибутов. Но нам удалось вытащить из нейронной сети достаточно, чтобы классифицировать все имеющиеся данные на понятные человеку и учтенные нейронной сетью атрибуты. В этом посте мы расскажем, как это сделать.
Методов для интроспекции нейронных сетей придумано достаточно. Первое, что приходит в голову:
(Еще можно посмотреть здесь).
Преимущественно все эти методы исследуют и объясняют предсказание только одного объекта. Методов, которые изучают нейронку целиком, пытаются выяснить, что вообще выучила сетка, какие концепты и высокоуровневые признаки содержатся в данных, критически мало. Для понимания мест, где качество нейронки (Feature Extractor) может проседать, нужна информация обо всех примерах в структурированном виде.

В ходе решения задачи распознавания лиц у нас возникла такая гипотеза, что в данных содержится гораздо больше информации, чем остается после сжатия в вектор признаков (Feature Vector). К примеру, у лиц, несомненно, есть атрибуты. Принято полагать, что на каждом следующем слое нейронной сети выделяются все более и более высокоуровневые признаки: начиная с уголков, черточек и других примитивов, заканчивая прической, полом, возрастом (применительно к распознаванию лиц). Мы не знаем заранее, какие высокоуровневые признаки на самом деле выделяются, из-за отсутствия разметки. Например, это могут быть: ношение очков, пол, ракурс съемки, прочие визуальные препятствия на фото. Если получить разметку таких скрытых атрибутов удастся, то можно фильтровать данные по ним, собирать малопредставленные в данных признаки (очки, маски) и так далее. В общем, вещь полезная.
Мы провели анализ собственной сетки распознавания лиц, и это помогло лучше понять, что происходит в обучении, и занять первое место в NIST. Мы очень вдохновились полученными результатами и решили поделиться методологией с сообществом.
Идея
Как достать больше информации из нейронной сети? Хочется взглянуть на ее внутреннее представление до сжатия в вектор признаков. Однако размерность пространства достаточно большая, и анализировать его сложно. На помощь могли бы прийти PCA или TSNE, которые отлично справляются со сжатием в ограниченное число размерностей. Рассмотрим PCA:
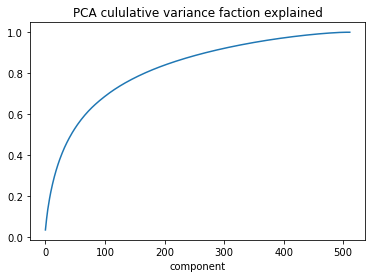
Рассмотрим компоненты PCA и визуализируем первые 10 из них картинками из датасета. Выясняется, что:
Чтобы объяснить 80% вариативности, нужно достаточно много (200) компонент.
-
Анализ главных компонент PCA не оказался информативным, преобладание компоненты не означало наличия интерпретируемого признака (зато там есть Гарри Поттер).

Анализ главных компонент на признаках нейронной сети
Теперь TSNE. Его проблемы состоят в том, что:
Он не масштабируется на размер датасета, который предполагается исследовать: мы не дождемся результатов.
Если мы сделаем сжатие на меньшей выборке, экстраполировать на остальную выборку не представляется возможным: есть fit, нет predict.
Предположим, мы запустили TSNE. Далее нам пригодится сделать кластеризацию, чтобы выявить похожие примеры из датасета. Тут надеемся, что кластеризация сможет выявить необходимые нам атрибуты.
Масштабируем TSNE + Кластеризацию
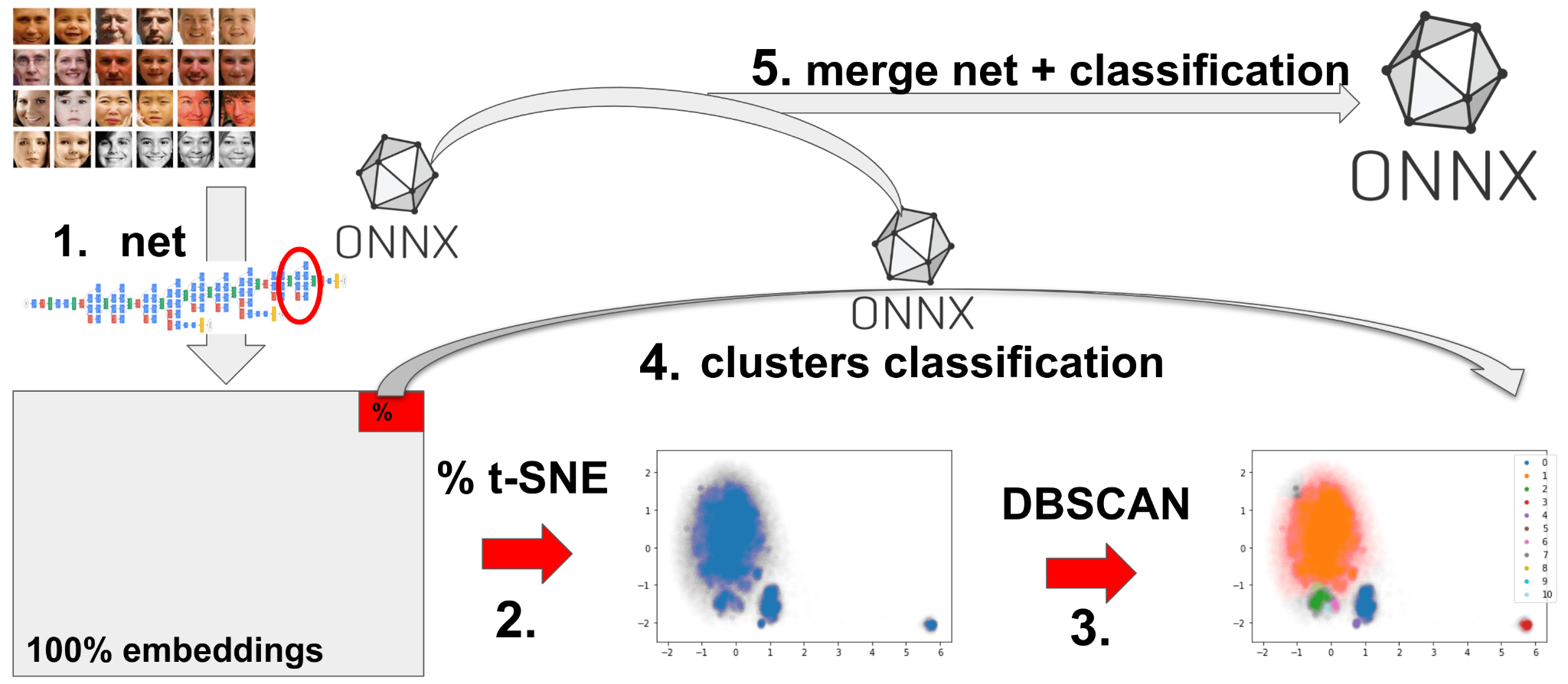
Решение кроется в том, что в действительности нам не так важен результат работы TSNE – куда важнее получить метки кластеров. Будем использовать внутреннее представление нейронной сети – эмбеддинги. Если нам надо получить только метки, мы можем, например, сделать следующий трюк (нумерация соответствует картинке выше):
Прогоняем нейронную сеть и делаем подвыборку из эмбеддингов датасета.
Делаем TSNE на подвыборке (2% – в нашем случае).
Делаем кластеризацию на результате п.2.
Обучаем классификатор «эмбеддинг – номер кластера». Сохраняем модель в ONNX.
Объединяем ONNX-граф нейронной сети с ONNX-графом классификатора.
Прогоняем классификатор на полной выборке или новых данных.
При таком подходе мы сможем аппроксимировать результат понижения размерности + кластеризации на неограниченный объем данных. Приступим.
Готовимся
Предполагается, что проделанный анализ может провести любой желающий, поэтому необходимые ресурсы для эксперимента были весьма ограниченными.
NVIDIA GeForce RTX 2080 Ti
Intel(R) Core(TM) i7-9700 CPU @ 3.00GHz
64G RAM
Такой конфигурации будет более чем достаточно, чтобы и извлечь эмбеддинги, и запустить TSNE и провести кластеризацию.
Весь используемый код находится в этом репозитории: https://github.com/NTech-Lab/dl-tsne
В посте будут только важные сниппеты без очень технических подробностей, за деталями лучше идти сразу в репозиторий и читать.
Подготовка нейронной сети
Перед началом работы необходимо, конечно, обучить нейронную сеть на задаче (в нашем случае – это распознавание лиц) и сконвертировать ее в формат ONNX. В формате ONNX, оказывается, значительно проще работать с вычислительным графом и извлекать промежуточные слои. Более того, пайплайн хорошо переносится на широкий спектр фреймворков обучения.
Для открытого эксперимента мы взяли нейронную сеть распознавания лиц из репозитория InsightFace: webface_r50_pfc.onnx. Одну из самых лучших, доступных для свободного скачивания в академических целях.
Извлечение признаков
Имея ONNX-модель, нам необходимо научиться доставать скрытые слои этой нейронной сети на новых картинках. Оказалось, что этого можно добиться средствами ONNX без привязки к фреймворку обучения (полезная ссылка). Пересохраняем модель с промежуточными слоями.
Модификация ONNX
model = onnx.load_model(model_file)
intermediate_tensor_name = model.graph.node[-4].output[0]
intermediate_layer_value_info = onnx.helper.ValueInfoProto()
intermediate_layer_value_info.name = intermediate_tensor_name
model.graph.output.extend([intermediate_layer_value_info])
onnx.save(model, "interim+"+model_file)О выборе промежуточного слоя. Посмотреть список промежуточных слоев в нейронной сети можно через model.graph.node – это лист из нод ONNX. Для желаемого слоя нам надо узнать имя тензора, где сохраняется результат выхода. Интуиция такая, что:
чем глубже слой, тем более высокоуровневые признаки там содержатся;
беспроигрышным вариантом – будет выбрать последний слой перед сжатием информации (любое понижение размерности, перевод в логиты классификации, регуляризационные боттлнеки и т.д.).
Конечно, хочется использовать сам вектор признаков лица, однако это будет не оптимально. Если посмотреть на устройство нейронной сети, увидим, что вектор признаков размерности 512 получается из тензора размерности 512х7х7 и сжимает информацию. В нашем случае – у сетки webface_r50_pfc.onnx сжатие информации происходит для создания эмбеддинга лица. Последний слой перед сжатием это:
model.graph.node[-4]
input: "679"
input: "bn2.weight"
input: "bn2.bias"
input: "bn2.running_mean"
input: "bn2.running_var"
output: "680"
name: "BatchNormalization_126"
op_type: "BatchNormalization"
attribute {
name: "epsilon"
f: 9.999999747378752e-06
type: FLOAT
}
attribute {
name: "momentum"
f: 0.8999999761581421
type: FLOAT
}Подготовка данных
Для эксперимента мы скачали уже подготовленные данные glint360k, ссылку на скачивание можно найти в репозитории InsightFace (распакован в data/glint360). Для использования своих датасетов можно обратить внимание на скрипт, который мы подготовили (он задействует пайплайн инсайта для детекции и нормализации). Мы сложили все в папку data/, чтобы можно было, в случае чего, подменить данные.
Для однообразного доступа к каждой картинке можно использовать простые списки файлов. Например, файл glint360.txt был создан, как
cd data
find ./glint_orig/ -name '*.jpg' > ../lists/glint360.txtГотовим признаки
Обязательно выясните, как нормализовать картинки для нейронной сети, потому что неправильная нормализация испортит весь анализ, и его придется переделывать.
def prepare_batch(imgs):
if not isinstance(imgs, list):
imgs = [imgs]
blob = cv2.dnn.blobFromImages(
imgs, 1.0 / input_std, input_size,
(input_mean, input_mean, input_mean),
swapRB=True
)
return blobДля хранения признаков настоятельно рекомендуем использовать HDF5 формат, он удобен, переносим и выдерживает огромные размеры датасетов. Из недостатков самый неприятный – случайный доступ к конкретным элементам, что понадобится в дальнейшем. Имеет смысл сразу сделать как полный дамп эмбеддингов, так и подвыборку, чтобы потом сэкономить время.
Как мы организовали дамп в HDF5
Важно: для скрытого слоя берем центральный пиксель. Эмпирически???? выяснено, что это работает в среднем лучше, чем другие попытки (пробовали maxpool и avgpool).
P = 0.02
files = np.asarray(list(map(str.strip, open("../lists/glint360.txt").readlines())))
subset = np.random.RandomState(2463426724).random(len(files)) < P
subset_files = files[subset]
root = "../data/"
with tqdm.tqdm(subset_files) as _files, h5py.File(model_file + f".{P}-embeddings.h5", "w") as f:
prefacen = f.create_dataset("prefacen", (0, 512), maxshape=(None, 512), chunks=(512, 512))
facen = f.create_dataset("facen", (0, 512), maxshape=(None, 512), chunks=(512, 512))
for images in more_itertools.chunked(
map(cv2.imread, map(root.__add__, _files))
, 512):
batch = prepare_batch(images)
facen_i, prefacen_i = session.run(output_cfg, {input_name: batch})
prefacen.resize((prefacen.shape[0]+prefacen_i.shape[0], prefacen.shape[1]))
facen.resize((facen.shape[0]+facen_i.shape[0], facen.shape[1]))
prefacen[-prefacen_i.shape[0]:] = prefacen_i**[..., prefacen_i.shape[-1] // 2, prefacen_i.shape[-1] // 2]**
facen[-facen_i.shape[0]:] = facen_i
Можно всячески оптимизировать извлечение признаков, но это не было основной целью демонстрации. Мы просто оставили на ночь считаться, и вернулись к задаче на следующий день. Распараллелить цикл на несколько GPU, сохранять результаты в разные HDF5 файлы и потом объединять было бы гораздо быстрее.
TSNE + Кластеризация
Понижение размерности
Когда мы подготовили данные, у нас получилось два файла с эмбеддингами:
-rw-rw-r-- 1 user user 1.4G Sep 16 12:16 webface_r50_pfc.onnx.0.02-embeddings.h5
-rw-rw-r-- 1 user user 66G Sep 16 22:31 webface_r50_pfc.onnx.1-embeddings.h5Уже упоминалось, что случайную подвыборку делать ужасно долго, поэтому webface_r50_pfc.onnx.0.02-embeddings.h5 – то, что нам надо. Это 2%-я подвыборка из всего датасета, на ней можно проводить анализ, чтобы потом кластеризовать весь оставшийся датасет. Этот размер датасета выбран не случайно: он помещается в GPU (RTX 2080ti) для подсчета TSNE. Если у вас GPU пожирнее, можно увеличить подвыборку, но это не принципиально.
subset_embeddings = h5py.File("webface_r50_pfc.onnx.0.02-embeddings.h5", "r")
prefacen = subset_embeddings["prefacen"][()]В этом виде данные уже можно отправлять в TSNE (выбор гиперпараметров был произведен вручную):
tsne = tsnecuda.TSNE(
num_neighbors=1000,
perplexity=200, n_iter=4000, learning_rate=2000
).fit_transform(prefacen)Получаем вот такие двумерные признаки tsne из изначальных эмбедднигов (была размерность 512). Кластера визуально отличимы друг от друга, как и хотелось для проведения анализа.

Уже видны намеки на то, что это можно как-то кластеризовать. Прежде чем пойдем дальше, – пара слов о процессе обучения TSNE.
Подбор гиперпараметров (про них подробнее здесь и здесь) оказался исключительно ручным процессом. Не сильно долгий, чтобы быть неэффективным и безысходным, но и не настолько быстрый, чтобы оставить его без внимания. Особенно чувствительными параметрами TSNE оказались те, что показаны в сниппете. Отдельно хочется заметить, что при большой выборке параметр num_neighbors пришлось увеличить, без этого все остальные параметры были нечувствительны к изменениям – стабильно давали плохой результат. Потом опытным путем, чуть-чуть увеличив perplexity и увеличив количество итераций, получили достаточно многообещающую картинку (видны скопления объектов, кластера!). Все найденные параметры специфичны под эту конкретную сетку и данные. Для сетки в NtechLab оптимальными были чуть другие, но идея та же.
Кластеризация
Для кластеризации напрашивается DBSCAN с его порогом для разделения кластеров по плотности. Про DBSCAN можно почитать здесь.
Мы немного его адаптировали, чтобы он работал не так долго на таком объеме данных. Исходный DBSCAN работает несколько минут на всей подвыборке, а это слишком долго, чтобы быстро итерироваться и подобрать хорошие гиперпараметры. Идея такая:
берем подвыборку;
запускаем DBSCAN, получаем метки кластеров;
обучаем KNN на метках кластеров, размечаем все остальное;
получаем быструю версию DBSCAN????.
KNNDBSCAN(sklearn.cluster.DBSCAN):
class KNNDBSCAN(sklearn.cluster.DBSCAN):
"""DBSCAN worked well when I sample down points. But gives no prediction.
So I train KNN on top of cluster labels
"""
def __init__(self, *args, subset=1, knn_params=None, random_seed=42, **kwargs, ):
super().__init__(*args, **kwargs)
knn_params = knn_params or dict()
self.knn = sklearn.neighbors.KNeighborsClassifier(
n_jobs=kwargs.pop("n_jobs", None),
**knn_params
)
self.subset_ = subset
self.rng = np.random.RandomState(random_seed)
def subset(self, X, y=None):
train_idx = np.arange(len(X))
self.rng.shuffle(train_idx)
train_idx = train_idx[:int(len(X) * self.subset_)]
if y is None:
return X[train_idx]
else:
return X[train_idx], y[train_idx]
def fit(self, X):
train_X = self.subset(X)
super().fit(train_X)
train_labels = self.labels_
train_kidx = np.where(train_labels >= 0)
self.knn.fit(train_X[train_kidx], train_labels[train_kidx])
del self.labels_
return self
def predict(self, X):
return self.knn.predict(X)
def fit_predict(self, X):
return self.fit(X).predict(X)Подбор гиперпараметров DBSCAN – еще более долгий процесс, чем TSNE. Подобрать гиперпараметры было непросто, чтобы разбивка на кластеры выглядела визуально хорошо. Если у кого есть идея, как сделать лучше, – велком.
ntsne = (tsne - tsne.mean(0)) / tsne.std(0)
y = KNNDBSCAN(min_samples=120, subset=0.5, eps=0.05, knn_params=dict(n_neighbors=5)).fit_predict(ntsne)
display_labels(ntsne, y, slc=slice(None, None, 5), alpha=0.002)
Предварительные результаты
Для текущей подвыборки мы можем найти наиболее явные кластеры и посмотреть на фото, которые им соответствуют.

Если присмотреться, то маленькие кластеры (10, 8, 6, 4 и 3) очень репрезентативны. Например, третий – кластер с очками. С маленькими кластерами и связана вся сложность подбора гиперпараметров. Они яркие, но выделить их бывает сложно.
Переносим модель на терабайты
Мы получили разметку для наших данных, но у нас осталось 98% данных без этой разметки по кластерам. Для применения модели на практике необходимо все это дело довести до ума. В идеале – иметь ONNX-модель, которая делает все сразу. Это достижимый результат, давайте его реализуем.
Доменная регрессия
Как ни странно, но для применения модели нам будет достаточно обычной линейной регрессии. Обучим ее.
from sklearn.linear_model import LogisticRegressionCV
cluster_model = LogisticRegressionCV(n_jobs=-1, max_iter=1000)
cluster_model.fit(prefacen, y)
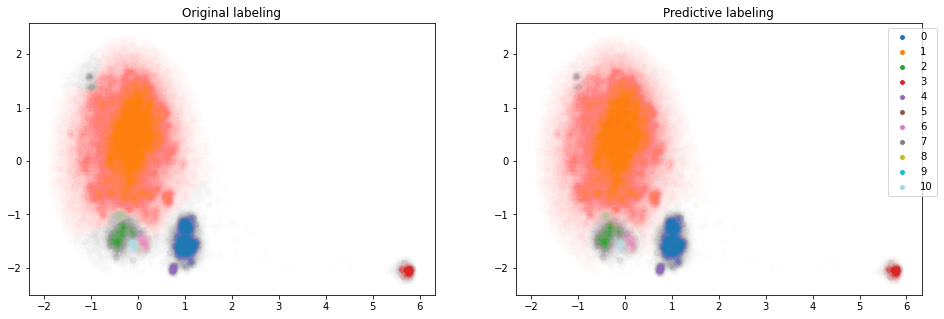
score = cluster_model.score(prefacen, y)В моем случае score=0.9623, что достаточно неплохо. На нашей практике проблемы наблюдались когда score<0.7, так что 0.96 – достойный результат. ROC AUC (ovo/ovr; weighted/macro) больше 0.975, что тоже не вызывает подозрений. Визуальное сравнение тоже в порядке. Идем дальше!
Конвертируем в ONNX:
from skl2onnx import convert_sklearn
from skl2onnx.common.data_types import FloatTensorType
initial_type = [('float_input', FloatTensorType([None, 512]))]
options = {id(cluster_model): {'raw_scores': True}}
onx = convert_sklearn(cluster_model, initial_types=initial_type, options=options)
with open("cluster_model.webface_r50_pfc.680.onnx", "wb") as f:
f.write(onx.SerializeToString())Применение модели
Итак, на данном этапе мы обучили модель классификации кластеров по эмбеддингам сетки. Но, чтобы применить это на практике эффективно, нам надо сделать несколько дополнительных шагов. Нерационально иметь две модели, одна из которых зависит от другой. Для инференса лучше сделать одну общую ONNX-модель под капотом, запуская модель кластеризации по эмбеддингам. Приступим.
У нас имеются:
cluster_model.webface_r50_pfc.680.onnxwebface_r50_pfc.onnx
Для манипуляции графами ONNX была использована библиотека sclblonnx. Она позволяет склеивать графы, подменять входы и выходы, то что нужно.
Код конвертации:
# load 2 models to merge
# note that all operations below mutate the input graph
model = so.graph_from_file("webface_r50_pfc.onnx")
cluster = so.graph_from_file("cluster_model.webface_r50_pfc.680.onnx")
# prepare nodes to extract the correct slice
node_slice = onnx.helper.make_node(
'Slice',
inputs=['680', "680.start", "680.end", "680.axes"],
outputs=['680.slice.nd'],
name="Slice.680"
)
node_squeeze = onnx.helper.make_node(
'Squeeze',
inputs=['680.slice.nd'],
axes=[2, 3],
outputs=['680.slice'],
name="Squeeze.680"
)
# constants are required to pass to slice parameters, need to be added to the graph
model = so.add_constant(model, "680.start", value=np.asarray([3, 3]), data_type="INT64")
model = so.add_constant(model, "680.end", value=np.asarray([4, 4]), data_type="INT64")
model = so.add_constant(model, "680.axes", value=np.asarray([2, 3]), data_type="INT64")
# extracting slice from 680 layer
model = so.add_node(model, node_slice)
model = so.add_node(model, node_squeeze)
# merging
model12 = so.merge(model, cluster, io_match=[("680.slice", "float_input")], complete=False)
# fix weird shape warning, complained on shape 1 output
out0 = onnx.helper.ValueInfoProto()
out0.name = model12.output[0].name
model12.output.remove(model12.output[0])
model12.output.insert(0, out0)
# saving
so.graph_to_file(model12, "webface_r50_pfc+cluster.onnx")Теперь можно запускать модель на новых данных и получать метки кластеров. К примеру, третий кластер с очками уже из исходного датасета, где большей части не было в обучении модели кластеризации:

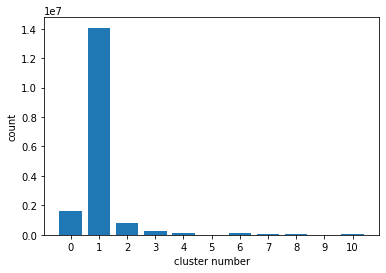
Вот так выглядит распределение кластеров в датасете:
Выводы
Кластеры можно разделить условно на желаемые и нежелаемые. Желаемые кластеры – это, например, пол, где разные кластеры не могут быть одним человеком. Нежелаемые кластеры, наоборот, объединяют людей по признаку, который мы хотим игнорировать. Например, качество снимка, угол съемки, очки, маска.
В исследуемой сетке наблюдается повышенное внимание к очкам, и хочется дальше исследовать это направление в поиске решения связанной адаптации модели к разным доменам (про это писали здесь).
Схожие результаты анализа на наших данных мы использовали для:
подбора обучающей выборки для тренировки новой модели;
фильтрации нежелательных, на наш взгляд, кластеров (картинки с «мусором»).
Что можно было сделать лучше
В целом результат получился хороший: даже маленькие домены (недостаточно представленные, но отличные от общей массы) выделены, и они репрезентативны. Как всегда, есть нюансы, которым стоило бы уделить больше времени на доработку, полировку:
разделить получше большой первый кластер, так как там достаточно смешанный домен;
использовать другой алгоритм кластеризации: думаем, OPTICS должен быть более гибким, по сравнению с DBSCAN (реализация в sklearn здесь).


ChePeter
Отличная статья.
"Принято полагать, что на каждом следующем слое нейронной сети выделяются все более и более высокоуровневые признаки: начиная с уголков, черточек и других примитивов, заканчивая прической, полом, возрастом (применительно к распознаванию лиц). "
Я тоже так думал, но затеял проверить.
Стал создавать исскуственные картинки из примитивов ( отрезки прямые ) и их комбинаций ( проекции многоугольников, полигоны )
Потом просто выкинул примитивы и раскрасил полигоны шумом, только с разными характеристиками.
Сеть всё равно отличала проекцию семиугольника от проекции квадрата заполненные одинаковым(!) шумом.
Т.е. линий нет, примитивов нет, есть области разной формы. Но она всё равно их отличает.