
Доброго времени суток, дорогой Habr, надеюсь вы успели заскучать после 3-х месяцев паузы (ссылка на прошлую статью). На связи снова Николай Иванов и сегодня вы узнаете, что общего между раком крови, лазерами и машинным обучением. В этой статье мы поговорим с вами о методе проточной цитофлуориметрии, как он работает и как врачи определяют рак костного мозга и крови. Далее обсудим причём тут машинное обучение, как его можно использовать для ускорения работы врачей, а также чего мы достигли и есть ли смысл этим вообще заниматься? Рассказ я буду вести именно в том порядке, в котором мы двигались, при решении данной задачи. Приятного чтения!
Начнём с проточной цитофлуориметрии

Проточная цитометрия (проточная цитофлуориметрия) — метод исследования дисперсных сред в режиме поштучного анализа элементов дисперсной фазы по сигналам светорассеяния и флуоресценции. На картинке выше схематично изображен принцип работы проточной цитометрии: как движутся клетки, зеркала, лазеры и АЦП. Если просто и кратко: мы помечаем клетки специальными красителями и, пока они движутся в потоке, облучаем лазером. В зависимость от красителя, светофильтра и положения клетки — получаем разные картинки на детекторе. Подробнее тут (ссылка).
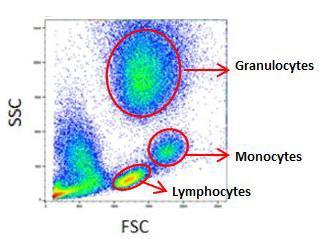
Данные метод является стандартным для детекции различных видов лейкозов. Есть определенный набор каналов, которых хорошо изучен и позволяет определять группы раковых клеток. Врачи обычно строят попарные графики либо с различными красителями, либо с различными положениями клеток, попавших на детектор (например, FSC (Forward scatter) — рассеивание под прямым углом, SSC (Side scatter) — рассеивание под углом 90). Вот примеры картинок (взято отсюда), которые анализируют врачи:
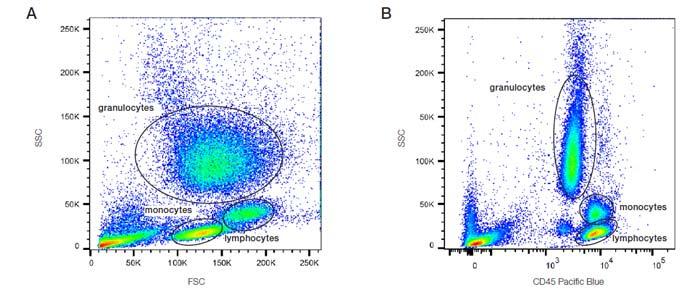
Видно, что клетки формируют различные группы, которые можно отделять друг от друга визуально, чем и занимаются врачи.
А зачем что‑то менять? Метод рабочий, куча исследований и статей по тому, как правильно работать с этими изображениями, точность врачей высока, зачем тут машинное обучение? А я вам отвечу!)
Время. На поиск кластеров и просмотр таких попарных взаимодействий, врач тратит достаточно много времени.
Размерность. Врач смотрит на двумерные графики. Да он может достаточно точно найти информацию по двумерному графику, но такие данные изначально представлены в многомерном пространстве, где больше информации. Посмотреть на картинку из 10-мерного пространства трудновато)))
Точность. А вдруг у нас получится улучшить точность и сделать этот процесс автоматизированным?
Квалификация. Не у всех врачей есть достаточная квалификация для диагностики редких форм лейкозов. В данном случае модель, обученная на данных размеченных экспертами может служить хорошим подспорьем для менее опытных врачей.
Как минимум эти 4 причины заставили нас заняться этой сложной, но интересной задачей. Приступим!
Какие данные у нас есть?
Источником данных является один из крупнейших центров детской гематологии и онкологии, который любезно предоставила нам обезличенные данные для исследования. Данные хранятся в специальных форматах “.fcs” (подробнее тут). Этот формат данных используется для хранения большого массива информации которые внутри себя содержать коэффициенты компенсации (о них я расскажу ниже), коэффициенты для различных преобразований, сами детектированные точки, наборы флуоресцентов, а также некоторую разметку врачей, которую они использовали для постановки диагнозов. Для каждого пациента также имеется поставленный диагноз (Т‑лимфобластный лейкоз, Лейкоз В-2 и т. д.) и доля бластных клеток в крови. В данных имеется естественный для профиля данной клиники перекос в плане количества больных и здоровых пациентов, так как больница являются ведущим федеральным центром по диагностики детских онкогематологических заболеваний.
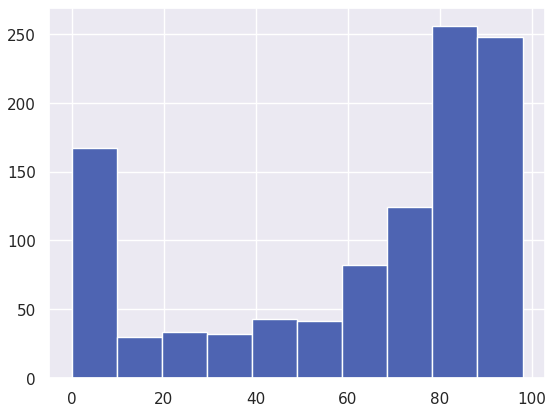
В целом, в выборке 150 здоровых пациентов, больных — 906. Количество бластных клеток от 0 до 100%. У здоровых 0% бластных клеток, чем тяжелее болен человек тем больше процент. Распределение пациентов по количеству бластных клеток показано на диаграмме. Такой дисбаланс здоровых и больных будет достаточно сложно победить, но мы пытались). Тут сразу и задачу, и метрику можно выбрать. Если задача регрессии, тогда метрики RMSE или MAE. Можно решать также задачу классификации, есть рак или нет, тогда подходящие метрики ROC AUC, Precision, Recall для обоих классов.
Для каждого пациента имеется своя таблица данных. Она представляет из себя точки и то как они детектировались в зависимости от красителей. Размер впечатляет — около 28 тысяч точек на 13 колонок (это те самые красители и различные углы измерения, под которыми клетки попадают на детектор). Пример:
Для быстроты обработки, мы преобразовали данные в parquet, так как записывать в данные мы ничего не собираемся — это приемлемый вариант с точки зрения скорости чтения.
Нужно сказать пару слов про разбиение данных. Пациенты были разделены по в соотношении 70/15/15. 70% — Данные для тренировки, 15% — валидация, 15% — тест, их модель не видела и под них не настраивались параметры. Важно отметить, что данные были отобраны по принципу out‑of‑time — стандартный прием при работе с данными, зависящими от времени. Отметим также что наши данные могут немного варьироваться, в более новых образцах могут быть удалены некоторые каналы; может использоваться более современное и точное оборудование. Также мы фиксировали сиды и считали все метрики на 10 сидах, для более справедливого сравнения моделей. Очень важно отметить, что для нашего алгоритма мы использовали лишь скрининговые данные.
Как их анализировать?
Когда мы впервые увидели данные и решили посмотреть на них в 2D, увидели вот это.
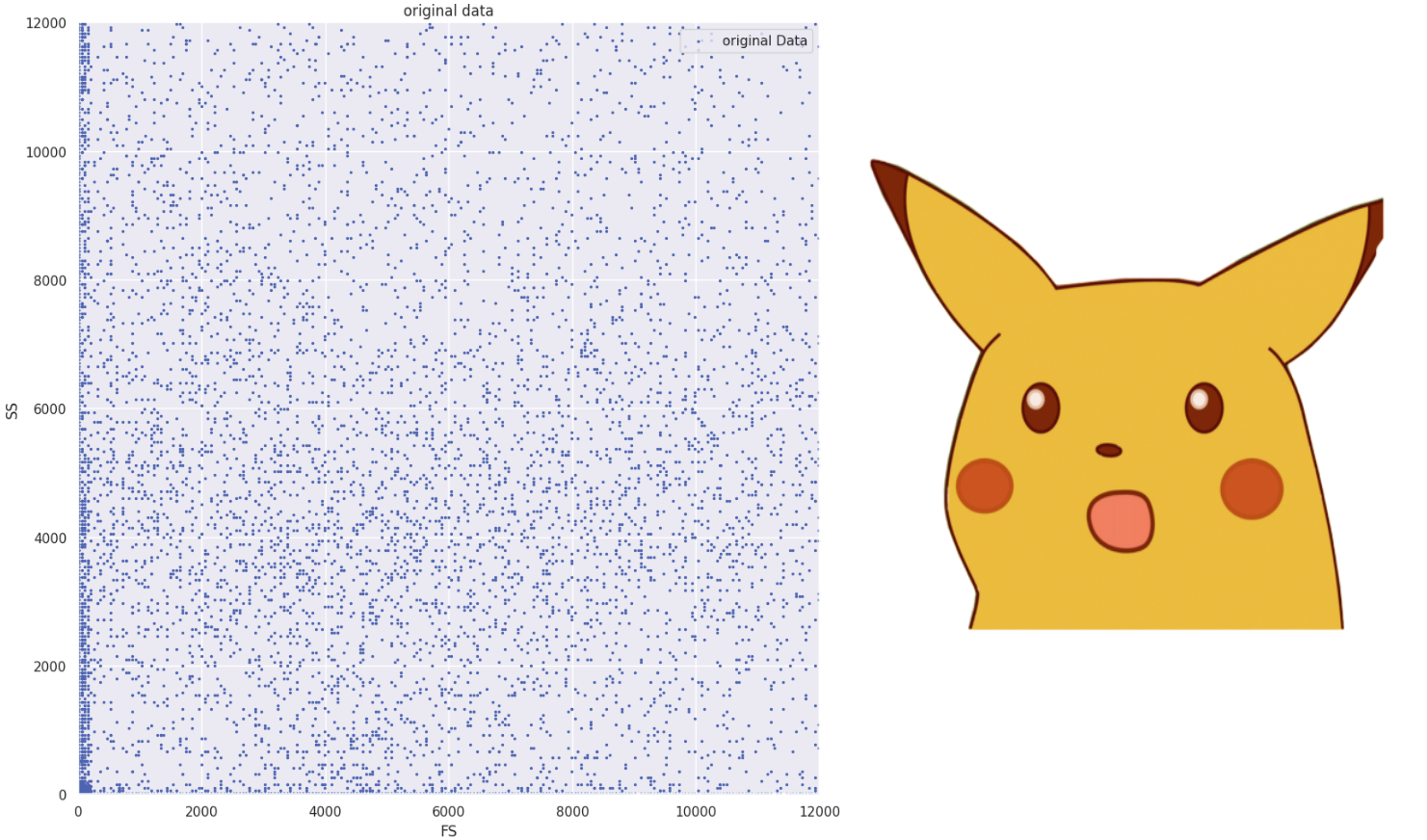
Стоп, что? У врачей такие красивые картинки были, а где кластеры??? Что происходит — первая моя реакция. Немного заспойлерю: более 80% данных оказались выбросами).
Почему это именно выбросы? Если посмотреть на картинки, на которые смотрит врач, по ним явно определяются различные группы клеток, такие как лимфоциты (ссылка), моноциты (ссылка) и другие. Тут мы видим полный хаос. Хотя сначала мы пробовали обучать и без удаления выбросов ????????.
Теперь необходимо сказать, что ко всем необходимым трансформациям, мы пришли не сразу, это был длительный и трудный процесс.
Как мы убрали эти выбросы и получили читаемые картинки?
Компенсация
Трансформация
Эвристики
Начнём с компенсации.
После возбуждения лазером флуоресцентные реагенты, связанные с клетками или частицами, излучают свет, который обнаруживается в соответствии с их конкретным диапазоном длин волн. При использовании комбинации флуоресцентных красителей эти реагенты должны иметь отличающиеся спектры излучения. Происходит спектральное перекрытие между флуоресцентными каналами, иначе говоря, мы получаем искажённую картину для каждого из каналов, зашумлённую если удобно, другими каналами. Подробнее о этом процессе написано тут. Для того чтобы это учесть такие эффекты искажения существуют специальные матрицы компенсации, обычно они зашиваются прямо в прибор. И для того, чтобы получить исходный сигнал, полученные на детекторе без шума, необходимо обратить эту матрицу и умножить на данные, полученные на приборе. Опять же подробнее можно почитать здесь. Пример матрицы компенсации выглядит так:
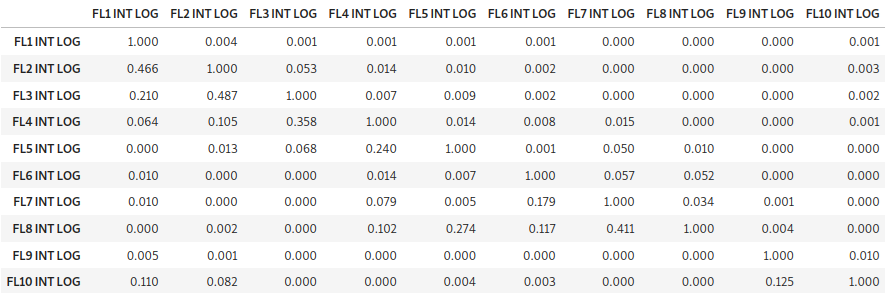
После применения компенсации обычно удаётся удалить шум в соседних каналах.
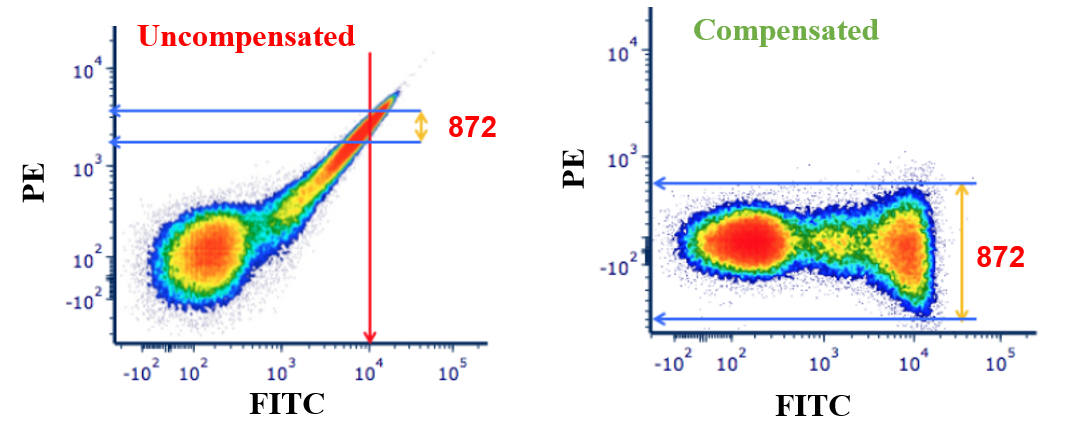
Трансформация.
Однако, применяя только лишь операцию компенсации, добиться чётких точечных диаграмм не получится. Так как даже после компенсации, выбросы никуда не пропали, в данных всё ещё могут встречаться огромные значения 1e256 и др. Используем трансформацию! Она позволит изменить масштаб и форму данных, ну, и к тому же сами врачи её используют. Основные трансформации, которые используют врачи — это log transform и logicle transform.
Log Transform
Как его считать:
Logicle Transform
Как его считать:

Сравним эти два способа трансформации на тех же самых данных после компенсации, все коэффициенты вшиты в прибор и берутся для каждого пациента свои. Подробнее про то, как появились эти трансформации можно найти тут.
Log transform слева, Logicle transform справа
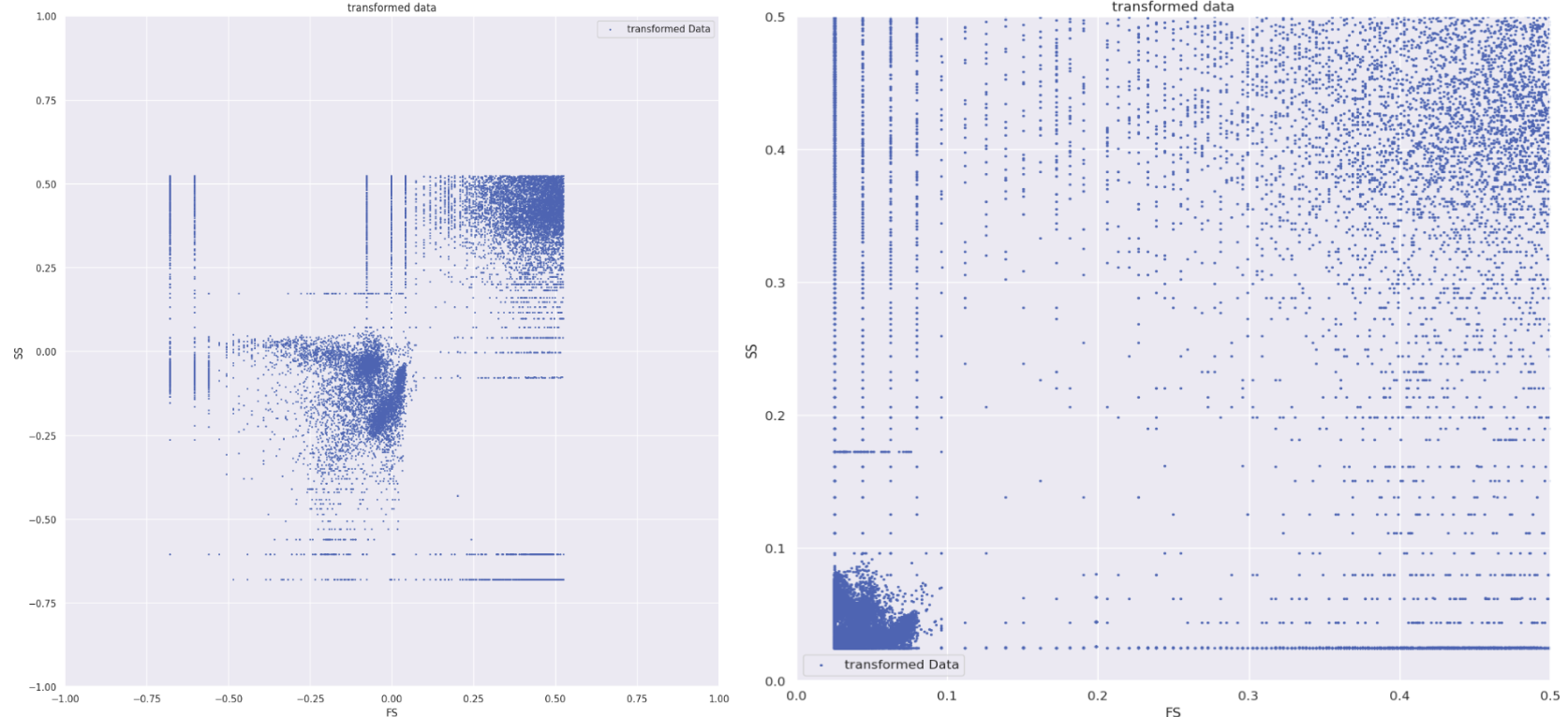
Как можно заметить, Logicle transform преобразовывает картину более «интересно»:
во‑первых, мы получаем нормированные данные со значениями от 0 до 1, что является предпочтительным при работе во многих моделях;
во‑вторых, при использовании log трансформации мы видим, что «полоски» остаются и снизу и сверху относительно тех данных, которые нам нужны;
в‑третьих, не совсем понятно, как делать «срез» относительно этих данных.
В итоге было принято решение использовать Logicle, кстати, сами врачи его чаще и используют, следуем по их стопам.
А теперь про эвристику.
Да, данные после преобразования стали нормированными, но всё ещё видны эти «полосы», которые явно не являются клетками (это выбросы, огромные значения, которые после трансформации превращаются в полосы). Тут в комнату входит эвристика! Если приблизить данные после Logicle transform в диапазоне от 0 до 0.1 можно увидеть такую картину:
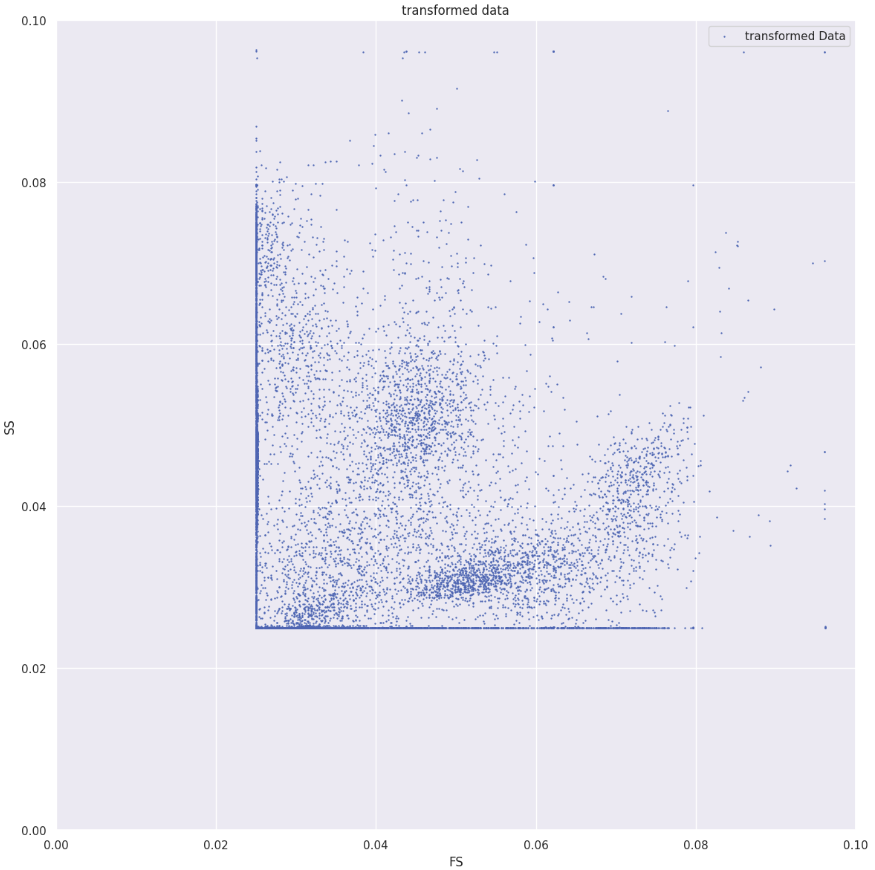
Вот референс с профильных форумов по цитометрии:
Другое дело! Мы получили то, что хотели, удалили все выбросы и получили адекватную картину, с точки зрения врачей. Вперёд к анализу!
Наивный подход
У нас теперь есть данные без шума. Давайте их анализировать! Стоп. А что нам с ними теперь делать? Для одного пациента у нас огромная таблица, с сырыми данными работать не хочется, хочется как‑то агрегировать эти данные в вектор, чтобы подать какому‑то классическому алгоритму машинного обучения.
Самая наивная и первая идея, которая пришла в голову: а что если посчитать средние, медианы, процентили и другие статистические характеристики данных для каждого пациента по каждому каналу и использовать их как признаки? Ну а давайте попробуем! В качестве регрессора мы пробовали разные модели, но лучше всего сработал бустинг. Вот таблица результатов.
Регрессия:
Модель |
Test RMSE |
Test MAE |
Pearson corr |
Spearman corr |
XGBoost |
23.01 |
14.49 |
0.75 |
0.8 |
Классификация:
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
XGBoost |
0.92±0.06 |
0.58±0.11 |
0.966±0.09 |
0.705±0.12 |
0.916±0.13 |
P.S. Все таблицы с метриками, представлены для тестовых данных.
Результаты, мягко говоря, не впечатляющие. Наивный подход плох тем, что мы смешиваем все группы клеток в один мешок, а можно ли лучше?
Кластеризация
Стоп, так врачи же выделяют кластеры? А давайте попробуем также использовать кластеризатор, и будем показывать ему сразу все 13 колонок. В качестве кластеризатора был выбран алгоритм «Kmeans++» (ссылка). Почему именно он? Мы полагаем, что количество кластеров фиксировано и примерно одинаково для всех больных и всех здоровых. Следовательно мы можем обучить его на всех пациентах сразу, чтобы найти центроиды кластеров, запоминая положение центроидов, мы можем при появлении новых клеток, отнести нашу клетку к одному из кластеров. А дальше, зная полученные кластеры, мы можем посчитать те же характеристики/признаки, что и в наивном подходе. Тогда сколько кластеров выбрать? Мы искали в промежутке от 10 до 100 кластеров, и выбирали по наилучшим метрикам. Самый лучший вариантом оказалось 50 кластеров. То есть мы имеем какие‑то кластеры клеток (в идеале мы хотим получить именно те кластеры клеток, которые имеются у нас в крови по типу лимфоцитов, моноцитов и др.) и имеем статистики по кластерам, то есть их размер, разброс и др. Такие признаки, чисто интуитивно, должны вносить больше информации.
Такой вектор признаков размерности 9150 для одного пациента, мы и подавали в регрессор и классификатор. И каков же результат? Ниже таблица для 10 сидов со средними и их стандартными отклонениями.
Модель |
Test RMSE |
Test MAE |
Pearson corr |
Spearman corr |
XGBoost +кластеры |
16.01±0.65 |
10.61±0.23 |
0.89±0.02 |
0.86±0.02 |
Мы видим значительное улучшение всех метрик. Идея сработала на ура!
Также посмотрим на метрики классификации, то есть если бы мы решали задачу предсказания, есть рак или нет, по тем же фичам, что и для регрессии(0 класс — это здоровые, 1 — больные).
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
XGBoost +кластеры |
0.952±0.002 |
0.68±0.07 |
0.966±0.01 |
0.773±0.05 |
0.947±0.01 |
Как можно заметить, из-за сильного дисбаланса, для здоровых пациентов метрики хуже.
Нейронные сети
А куда же без нейронных сетей. Попробуем на тех же признаках по кластерам, что и в предыдущем пункте обучить полносвязный персептрон (клац) со следующей архитектурой: 5 полносвязных слоёв + Dropout + GeLU. Также в процессе экспериментов пробовали добавлять, удалять слои, менять активацию, пока что лучшие результаты выглядят так.
Регрессия:
Модель |
Test RMSE |
Test MAE |
Pearson corr |
Spearman corr |
NN + кластеры |
17.4±0.2 |
12.98±0.31 |
0.88±0.04 |
0.85±0.03 |
Классификация:
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
NN + кластеры |
0.93±0.02 |
0.54±0.12 |
0.943±0.017 |
0.69±0.11 |
0.931±0.09 |
Нейронная сеть ошибается на других объектах нашей выборки, что в потенциале может привести нас к более успешной классификации для класса здоровых и больных.
Детекция аномалий
Идея с детекцией аномалии была не супер гениальная. У нас лейкозные клетки должны отличаться от обычных (условно нормальных). А не попробовать ли нам детекцию аномалий, подумали мы? И попробовали! Пробовали две самых популярных метода: Local Outlier factor и Isolation Forest. Подробнее о них можно почитать в документации библиотеки sklearn. Ниже представлены результаты для задачи классификации от наших двух кандидатов.
LocalOutlier
Модель |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
L_Out. + кластеры |
0.96±0.07 |
0.76±0.09 |
0.41±0.13 |
0.99±0.04 |
IsoForest
Модель |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
Iso + кластеры |
0.36±0.23 |
0.91±0.01 |
0.41±0.09 |
0.89±0.07 |
Как можно заметить, LocalOutlierFactor очень хорошо справляется с классом здоровых. Так что данный алгоритм будет нам полезен.
Стэкинг
Я думаю, опытные машин лёрнеры уже догадались к чему мы идём. Используем все наши замечательные алгоритмы вместе, и подадим их предсказания на вход решающему регрессору и классификатору, и интуиция подсказывает, что должно получиться что‑то суперхорошее. Проверим? (ссылка для ознакомления)
Регрессия:
Модель |
Test RMSE |
Test MAE |
Pearson corr |
Spearman corr |
2 anomaly + NN + XGBoost |
13.42±0.4 |
7.78±0.2 |
0.92±0.01 |
0.92±0.01 |
Классификация:
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
2 anomaly + NN + XGBoost |
0.956±0.01 |
0.72±0.1 |
0.973±0.004 |
0.82±0.03 |
0.954±0.01 |
Вау и ах! Вот это действительно хорошие результаты. Да, они не идеальны, но получить такой результат с помощью классических моделей и интуитивных эвристик, это великолепно!
Вам мало? нам тоже
Рассказав о наших результатах коллегам и врачам, мы услышали много интересных комментариев, идей и замечаний, некоторые из них: feature selection, свёрточные сети, интерпретируемость для врачей.
Действительно, заняться отбором признаков не помешало бы, так как 9150 — звучит многовато.
Попробовать свёрточные сети — отличная идея. В идеале, мы научим сетку находить те визуальные паттерны, которые находят врачи.
А вот с интерпретируемостью возникает проблема, возможно получится решить её с помощью сверток и их карт активации.
Свёрточные сети
Я попробовал обучить классическую свёрточную сеть, а также использовать transfer learning подход с модели ConvNextV2 (huggingface).
В качестве картинок были использованы 78 попарных точечных диаграмм, которые обычно строят врачи. Далее я использовал классическую пирамидальную схему с двумя свёрточными слоями, батч нормализацией (batch normalization), SiLU и MaxPooling, на выходе было 2 полносвязных слоя. Решалась только задача классификации. Таблица с результатами:
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
CNN |
0.93 |
0.76 |
0.95 |
0.7 |
0.96 |
Как ни странно, используя ConvNextV2 модель дико переобучалась и определяла всех больными(
Модель |
roc_auc |
Recall_0 |
Recall_1 |
Precision_0 |
Precision_1 |
CNN |
0.29 |
0.0 |
1.0 |
0.0 |
0.86 |
А что с картами активации в CNN? Давайте посмотрим вместе.
Карты активации (feature map) для одного из больных и здоровых:
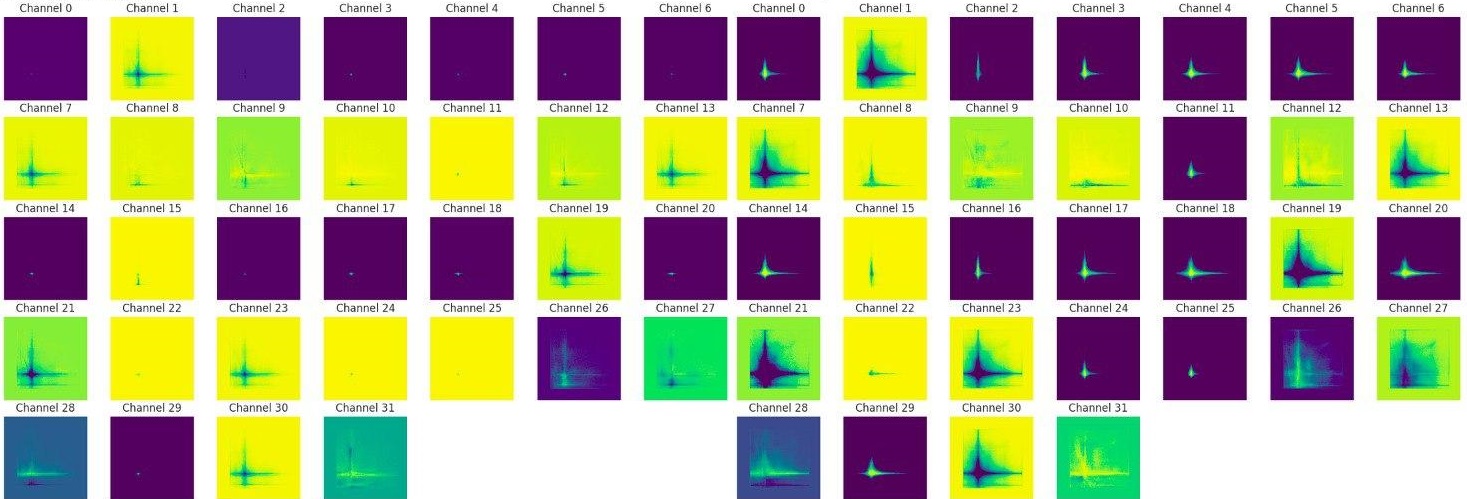
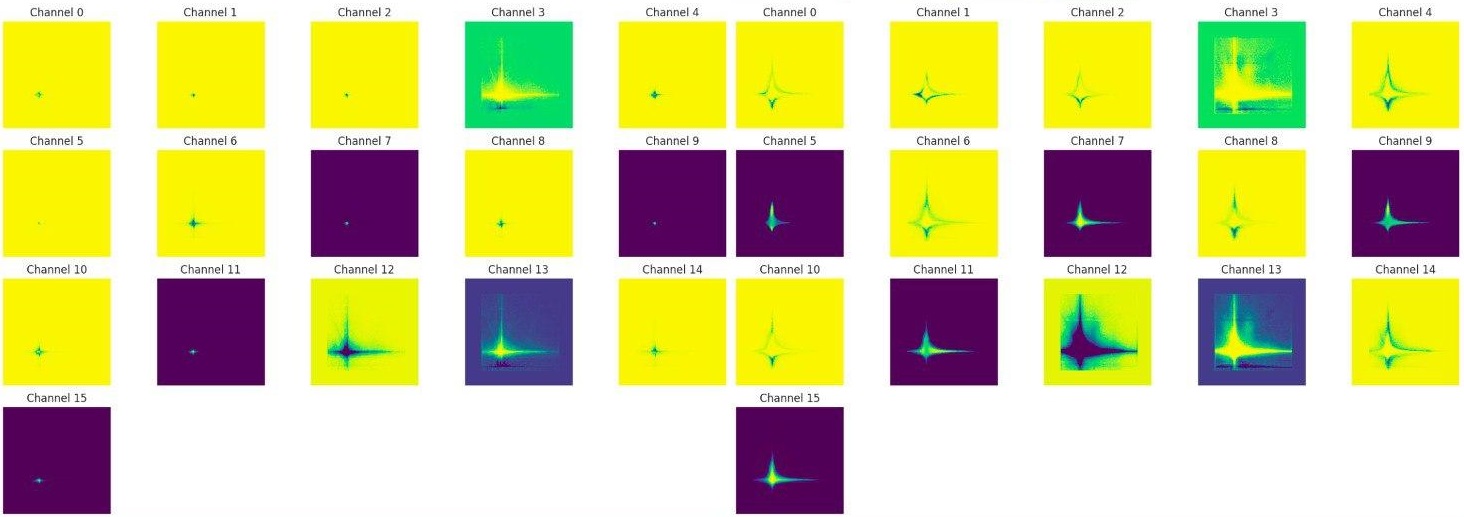
Можно заметить разные карты активации для здоровых и больных. И в целом по результатам на тесте видно, что CNN неплохо справляется как на больных, так и на здоровых. Но как выяснилось из разговоров с врачами, к сожалению, это не является той интерпретацией, которую они ожидали увидеть. Врачам было бы интересно получить картинку, с которой им привычно работать, например график FSC vs SSC, на котором выделялись бы отдельными цветами бластные клетки. В целом они хотят смотреть на более приближенную к их работе картинку, но с прорисованными кластерами бластных клеток.
Итоговые результаты
Как ни странно, добавление CNN в мета классификатор ухудшило метрики на тесте, но улучшило метрики на валидации. Для регрессора результат почти не изменился. Поэтому мы пока придерживаемся моделей без него.
А что делали другие?
Уже в процессе финализации результатов я наткнулся на статью 2018 года (ссылка). Там они делают акцент на скорость, а также говорят про неконсистентность мнений врачей, которую мы также упомянули. В их статье они учились в том числе отделять различные типы лейкозов друг от друга, чем мы не занимались. В их работе использовались две метрики: точность и ROC AUC. Также интересно, что использовали z‑score нормализацию. Их пайплайн состоял из трёх основных частей:
Использовали Gaussian Mixture Model (GMM) и метод максимального правдоподобия (подробнее) для поиска параметров эмпирического распределения для каждого из красителей.
Fisher gradient vectorization (подробнее уже у них в статье), чтобы более точно рассчитать параметры распределения каждого из красителей с помощью GMM.
Составили вектор из параметров распределения для каждого из пациентов и затем подавали в SVM (ссылка).
Подход кажется достаточно простым и понятным и концептуально похож на наш базовый подход. Справедливости ради, ребята решали даже более сложную задачу — выявление минимальной остаточной болезни. Избежать проблем с визуализацией им также не удалось. Ещё одно интересно наблюдение, что они использовали меньше каналов и метрика не изменилась сильно.
Финальный аккорд
На данный момент у нас имеется пожелание придумать интерпретацию модели (врачи хотят видеть конкретные кластеры клеток, на которые модель смотрит, при принятии решения), а также запрос на повышение чувствительности до 99–100%! Мы не собираемся останавливаться и будем пробовать улучшить наш результат, возможно проверим более современные ML‑алгоритмы, отобрать признаки. Также для продолжения работы необходимо увеличение выборки данных и выравнивание дисбаланса классов.
Если у вас есть идеи ждём ваши предложения в комментариях.
Работы по проекту ещё очень и очень много, но мы надеемся помочь врачам всего мира в этом нелёгком деле.
Всем спасибо!
Комментарии (8)

desperadoo95
30.11.2023 11:36+1Понравилась статья, очень большая работа была проделана, видео сразу ! Пишите чаще, будем наблюдать

Viroslav_Venskii Автор
30.11.2023 11:36+1Спасибо за прочтение! Ну как только так сразу) Исследования требуют достаточно продолжительного времени)

CrazyElf
30.11.2023 11:36+2PCAне пробовали? Возможно, послеPCAкластеры лучше будут разделяться. Или чем вы 13 колонок к 2D виду приводили, было же какое-то преобразование 13 колонок в 2 координаты? ) Сходу вообще не очень понимаю, где там координаты у вас получаются )
Viroslav_Venskii Автор
30.11.2023 11:36+1PCA пробовали, но тут дело в другом. Мы брали 13 мерное пространство и там делали кластеризацию. После этого мы запоминали положения центроидов => можно осуществить классификацию каждой клетки, а уже потом рисовать в двумерном пространстве. PCA интересных и вменяемых результатов не дал(

CrazyElf
30.11.2023 11:36+1Понятно. Да, возможно в многомерном пространстве кластеризовать действительно лучше. Но тогда я бы попробовал разные методы кластеризации, их же много: https://scikit-learn.org/stable/modules/clustering.html Или там уже в скорость/количество данных упиралось и не всякий метод бы справился? Просто разные методы могут давать довольно разные результаты.

Viroslav_Venskii Автор
30.11.2023 11:36+1Да, прям в точку, самые быстрые методы и те работают достаточно долго (около часа для пациентов). Была ещё интересная идея использовать количество кластеров (для unsupervised методов) как ФИЧУ!!! И это работает тоже неплохо. То есть мы делали несколько раз DBSCAN, но с разными параметрами и получали табличку: типо DBSCAN(30, 300) - 20 кластеров. И на удивление такой подход для большого количества кластеров дал хорошие метрики, но хуже чем наши более умные подходы, да и объяснить это достаточно непросто для врачей)


Denisdrus
Спасибо, Николай, очень давно наблюдаю и пользуюсь вашими статьями, добра вам
Viroslav_Venskii Автор
Спасибо и Вам! А как Вы ими пользуетесь?)