Простейшая линейная регрессия
Пусть нам даны исходные данные в виде таблицы со столбцами x1, x2, x3 и y. И мы будем строить линейную модель зависимости y от факторов x, то есть модель будет иметь следующий вид:
y = b0 + b1 x1 + b2 x2 + b3 x3 + ?
где x, y — исходные данные, b — параметры модели, ? — случайная величина.
Поскольку х и y у нас уже есть, то наша задача заключается в расчете параметров b.
Обратите внимание, что мы ввели параметр b0, который также называется intercept, так как наша модельная линия совсем не обязательно будет проходить через начало координат. Если исходные данные отцентрированы, то этот параметр не требуется.
Чтобы построить модель нам также нужно ввести параметрическое предположение относительно ? — без этого мы не сможем выбрать метод решения. Какой попало метод мы не имеем права применить, потому что рискуем получить сколько угодно ошибочный результат, в то время как нам требуется «достаточно хорошая» оценка параметров модели. Так что для простоты и удобства предположим, что ошибки распределены нормально, поэтому мы можем использовать метод обычных наименьших квадратов (ОНК).
# загружаем исходные данные
df <- read.csv("http://roman-kh.github.io/files/linear-models/simple1.csv")
# запускаем расчет модели
# модель glm по умолчанию включает intercept, явно его указывать в данном случае не требуется
g <- glm("y ~ x1 + x2 + x3", data=df)
# выводим коэффициенты модели
coef(g)
import numpy as np
import pandas as pd
import statsmodels.api as sm
import patsy as pt
import sklearn.linear_model as lm
# загружаем файл с данными
df = pd.DataFrame.from_csv("http://roman-kh.github.io/files/linear-models/simple1.csv")
# x - таблица с исходными данными факторов (x1, x2, x3)
x = df.iloc[:,:-1]
# y - таблица с исходными данными зависимой переменной
y = df.iloc[:,-1]
# создаем пустую модель
skm = lm.LinearRegression()
# запускаем расчет параметров для указанных данных
skm.fit(x, y)
# и выведем параметры рассчитанной модели
print skm.intercept_, skm.coef_
# добавим фиктивную переменную для расчета intercept'а
x_ = sm.add_constant(x)
# создаем модель для метода обычных наименьших квадратов (Ordinary Least Squares)
smm = sm.OLS(y, x_)
# запускаем расчет модели
res = smm.fit()
# теперь выведем параметры рассчитанной модели
print res.params
# создаем модель на основе формулы
smm = sm.OLS.from_formula("y ~ x1 + x2 + x3", data=df)
# запускаем расчет модели
res = smm.fit()
# теперь выведем параметры рассчитанной модели
print res.params
# создаем матрицу факторов и результатов из формулы и исходного датафрейма
pt_y, pt_x = pt.dmatrices("y ~ x1 + x2 + x3", df)
# вызываем стандартную numpy'вскую процедуру оптимизации по МНК
# кстати, именно ее и вызывает LinearRegression из scikit-learn для неразреженной матрицы с исходными данными
# а для разреженной вызывается scipy.sparse.linalg.lsqr
res = np.linalg.lstsq(pt_x, pt_y)
# достаем коэффициенты модели
b = res[0].ravel()
print b
Обратите внимание, что все способы расчета коэффициентов дают один и тот же результат. И это совершенно не случайно. Вследствие нормальности ошибок оптимизируемый функционал (ОНК, OLS) оказывается выпуклым, а значит имеет единственный минимум, и искомый набор коэффициентов модели также будет единственным.
Однако даже незначительное изменение исходных данных, несмотря на сохранение формы распределений, приведет к изменению коэффициентов. Проще говоря, хотя реальная жизнь не меняется, и природа данных также остается неизменной, в результате неизменной методики расчета вы будете получать разные модели, а ведь по идее модель должна быть единственной. Подробнее об этом мы поговорим в статье про регуляризацию.
Изучаем данные
Итак, мы уже построили модель… Однако скорее всего мы допустили ошибку, возможно даже и не одну. Поскольку мы не изучили данные, прежде чем браться за модель, то получили модель непонятно чего. Поэтому давайте все же заглянем в данные и посмотрим что там есть.
Как вы помните, в исходной таблице было 4 столбца: x1, x2, x3 и y. И мы построили регрессионную зависимость y от всех x. Поскольку мы не можем одним взглядом сразу охватить весь 4-мерный гиперкуб, то посмотрим на отдельные графики x-y.
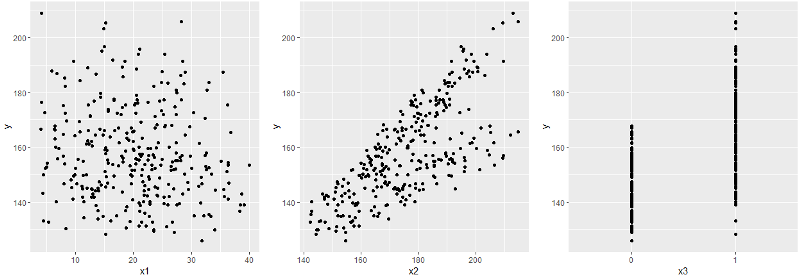
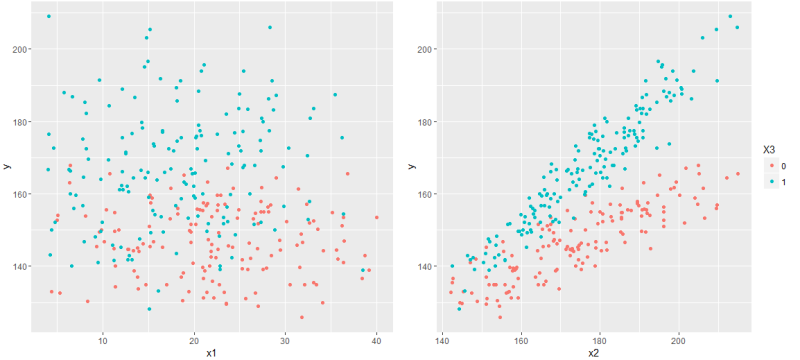
Сразу бросается в глаза x3 — бинарный признак — а с ними стоит быть повнимательнее. В частности, имеет смысл взглянуть на совместное распределение остальных x вместе с x3.
Теперь пришло время отобразить график, соответствующий построенной модели. И тут стоит обратить внимание, что наша линейная модель — это вовсе не линия, а гиперплоскость, поэтому на двумерном графике мы сможем отобразить только отдельный срез этой модельной гиперплоскости. Проще говоря, чтобы показать график модели в координатах x1-y надо зафиксировать значения x2 и x3, а, меняя их, получим бесконечное множество графиков — и все они будут графиками модели (а точнее проекцией на плоскость x1-y).
Поскольку x3 — бинарный признак — можно показать отдельную линию для каждого значения признака, а x1 и x2 зафиксировать на уровне среднего арифметического, то есть нарисуем графики y=f(x1 | x2=E(x2)) и y=f(x2 | x1=E(x1)), где E — среднее арифметическое по всем значениям x1 и x2 соответственно.
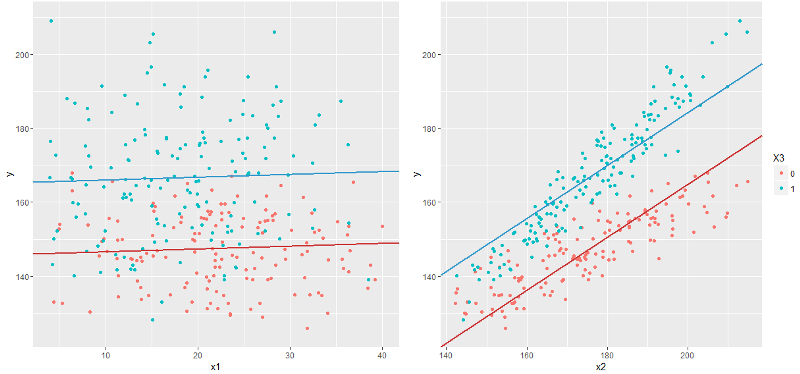
И сразу бросается в глаза, что график y~x2 выглядит как-то неправильно. Помня, что построенная модель должна предсказывать математическое ожидание y, тут мы видим, что модельная линия как раз математическому ожиданию совсем не сооветствует: например в начале графика голубые точки реальных значений находятся ниже голубой линии, а в конце — выше, причем у красной линии все наоборот, хотя обе линии должны проходить примерно посредине облака точек.
Приглядевшись внимательнее, можно догадаться, что голубая и красная линии должны быть даже непараллельны. Как же это сделать в линейной модели? Очевидно, что построив линейную модель у=f(x1,x2,x3) мы можем получить бесконечное количество линий вида y=f(x2 | x1,x3), то есть зафиксировав две из трех переменных. Так, в частности, получены красная линия у=f(x2 | x1=E(x), x3=0) и голубая у=f(x2 | x1=E(x), x3=1) на правом графике. Однако, все подобные линии будут параллельны.
Непараллельная линейная модель
Чтобы внести в модель непараллельность, немного усложним ее, добавив всего одно слагаемое:
y = b0 + b1 x1 + b2 x2 + b3 x3 + b4 x2 x3 + ?
К чему это приведет? Раньше любая линия на графике y~x2 имела один и тот же наклон, задаваемый коэффициентом b2. Теперь же в зависимости от значения x3 линия будет иметь наклон b2 (для x3=0) или b2+b4 (для x3=1).
# загружаем исходные данные
df <- read.csv("http://roman-kh.github.io/files/linear-models/simple1.csv")
# запускаем расчет модели
# модель glm по умолчанию включает intercept, явно его указывать в данном случае не требуется
g <- glm("y ~ x1 + x2 + x3 + x2*x3", data=df)
# выводим коэффициенты модели
coef(g)
import numpy as np
import pandas as pd
import statsmodels.api as sm
import patsy as pt
import sklearn.linear_model as lm
# загружаем файл с данными
df = pd.DataFrame.from_csv("http://roman-kh.github.io/files/linear-models/simple1.csv")
# x - таблица с исходными данными факторов (x1, x2, x3)
x = df.iloc[:,:-1]
# y - таблица с исходными данными зависимой переменной
y = df.iloc[:,-1]
# создаем новый фактор
x["x4"] = x["x2"] * x["x3"]
# создаем пустую модель
skm = lm.LinearRegression()
# запускаем расчет параметров для указанных данных
skm.fit(x, y)
# и выведем параметры рассчитанной модели
print skm.intercept_, skm.coef_
# создаем новый фактор
x["x4"] = x["x2"] * x["x3"]
# добавим фиктивную переменную для расчета intercept'а
x_ = sm.add_constant(x)
# создаем модель для метода обычных наименьших квадратов (Ordinary Least Squares)
smm = sm.OLS(y, x_)
# запускаем расчет модели
res = smm.fit()
# теперь выведем параметры рассчитанной модели
print res.params
# создаем модель на основе формулы
smm = sm.OLS.from_formula("y ~ x1 + x2 + x3 + x2*x3", data=df)
# запускаем расчет модели
res = smm.fit()
# теперь выведем параметры рассчитанной модели
print res.params
# создаем матрицу факторов и результатов из формулы и исходного датафрейма
pt_y, pt_x = pt.dmatrices("y ~ x1 + x2 + x3 + x2*x3", df)
# вызываем стандартную numpy'вскую процедуру оптимизации по МНК
# кстати, именно ее и вызывает LinearRegression из scikit-learn для неразреженной матрицы с исходными данными
# а для разреженной вызывается scipy.sparse.linalg.lsqr
res = np.linalg.lstsq(pt_x, pt_y)
# достаем коэффициенты модели
b = res[0].ravel()
print b
А теперь взглянем на обновленный график.
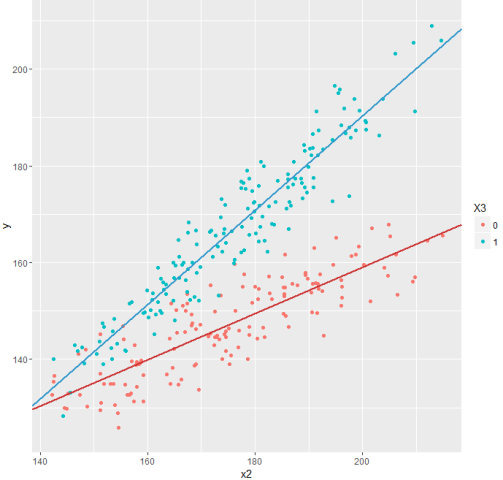
Гораздо лучше!
Этот прием — перемножение переменных — чрезвычайно полезен для бинарных и категорийный факторов и позволяет в рамках одной модели по сути построить сразу несколько моделей, отражающих особенности разных групп исследуемых объектов (мужчин и женщин, рядовых сотрудников и менеджеров, любителей классической, рок или поп-музыки). Особенно интересные модели можно получить, когда в исходных данных есть несколько бинарных и категорийных факторов.
По просьбам желающих я также создал небольшой ipython notebook.
Подводим итог: мы построили пусть простую, но все же весьма адекватную модель, которая, судя по графикам, неплохо отражает реальные данные. Однако, все эти «весьма» и «неплохо» лучше представить в измеримых величинах. А также пока остается непонятным, насколько построенная модель устойчива к небольшим изменениям в исходных данных или к структуре этих данных (в частности, к взаимозависимости между факторами). К этим вопросам мы обязательно вернемся.
Комментарии (14)

OlegUV
17.03.2016 07:29+1Вел дан! Для полноты картины можно было бы добавить код отрисовки графиков, а для того графика с параллельными линиями ещё пририсовать внизу график с остатками, для усиления видимости эффекта.

rinaskela
17.03.2016 11:10Отличная статья! Еще несколько таких и, уверен, многие перейдут с MathLab/MathCad/Mathematica на подобное окончательно. Тем более, что отрисовка графиков с помощью numphy становится чуть ли не единым стандартом для научных статей.

Arastas
17.03.2016 12:58+1Простите, но в какой области Вы пишите/читаете научные статьи?
Нет, в среднесрочной перспективе не перейдут, это разные инструменты. Да и незачем.
PS: MatLab — от Matrix, а не от Mathematics, там нет h в названии.
rinaskela
17.03.2016 13:06В основном статьи, касающиеся физики частиц, данные об экспериментах. Даже слышал от коллеги, что его редакция просила по возможности рисовать графики там.
PS: Действительно досадная опечатка с моей стороны. Два других наименования с "th" были :)

miptgirl
17.03.2016 11:12+2Спасибо за статью, очень познавательно и интересно.
В статье рассмотрено несколько Python библиотек: scikit-learn, statmodels и Patsy. Есть ли между ними какая-то принципиальная разница кроме синтаксиса (может, в функциональности или быстродействии)? С какой библиотеки лучше начинать свое изучение Machine Learning?
Roman_Kh
17.03.2016 13:25+2Patsy — это библиотека для подготовки данных и описания моделей, а все расчеты придется делать самостоятельно. В данном примере, с помощью patsy я лишь описал модель в виде удобной формулы, получив на выходе массивы все с теми же исходными данными, но немного в другом формате, которые затем передал для расчета модели в numpy.
Scikit-learn — очень богатая библиотека статистических методов и алгоритмов машинного обучения. Многие люди пользуются только ей, поскольку в ней есть все, что (им) нужно.
Statsmodels немного удобнее людям, переходящим на Python с R. В ней есть только стат.методы, причем по набору методов она сильно пересекается со scikit-learn. Хотя и в той и другой есть что-то уникальное, например, модель для таймсерий ARIMA есть только в statsmodels.
Так что без patsy можно обойтись. Основной анализ данных я обычно провожу с помощью scikit-learn, но и statsmodels бывает полезен и удобен.

381222
17.03.2016 11:59+1Спасибо за статью.
Лучше все таки сразу использовать метрику, оценка на глаз может промахнуться :)
В R после добавления переменной x2*x3 переменные x2 и x3 можно в модели не указывать, т.е. будет y~x1+x2*x3.
Переменная X имеет какой то физический смысл? Из за неравномерного распределения x3 от X, модель с X будет лучше, т.е. y~X+x1+x2*x3
x1 больше похож на шум, надо чистить/преобразовывать или убирать. Т.е. для glm будет лучше y~x2*x3
atikhonov
18.03.2016 10:12В R после добавления переменной x2x3 переменные x2 и x3 можно в модели не указывать, т.е. будет y~x1+x2x3.
верно, а если оставлять в исходном виде (перемножение (взаимодействие) переменных, g <- glm(y ~ x1 + x2 + x3 + x2x3, data=df)), то использовать надо :, а не , так как * это комбинация факторов


ariel32
В этом разрезе стоит обратить на пакет glmulti для R — он осуществляет автоматический перебор взаимодействий между переменными и дает на выход top-N моделей. Читаю с удовольствием, пишите! Очень интересна тема валидации моделей, и модели со смешанными эффектами. Особенно последнее дурманит мой разум и не дает спать по ночам...