На написание этой заметки меня вдохновила недавняя статья про кишочки обезьян. Поскольку чукча не читатель, чукча — писатель, то решил пробовать сделать подобное самому. Тем более задача не кажется сложной и много кода не потребуется.
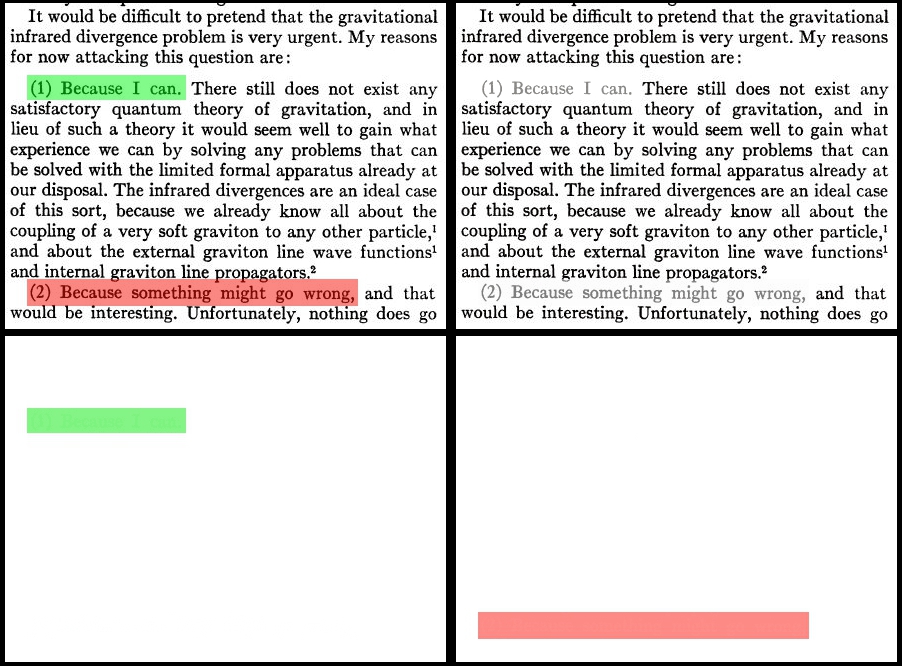
Простейший алгоритм, который приходит в голову, выглядит так:
Далее, более подробно по каждому пункту.
Базовые цвета это просто средние цвета сегментов изображения. Алгоритмов сегментации — море разливанное, выбирай на вкус, некоторые реализованы в Mathematica. Сразу скажу, что от выбора алгоритма сегментации мало что зависит, главное, чтобы число кластеров совпадало с желаемым числом компонент:
Ещё одна простенькая функция, которая нам понадобиться,
Теперь функция, которая возвращает базисные вектора.
Запускаем…
А не, погодите, не вуаля. Разлагать картинку по таким базисным векторам нет никакого смысла: полученные компоненты будут более менее повторять кластеры. Вот тут момент истины, т.е. единственный неизбежный эвристический шаг. Для того, чтобы компоненты картинки получились более полными, надо базисные вектора чуть-чуть раздвинуть.
Если мы хотим, из вектора а вычесть вектор b, при этом хотим сохранить компоненты вектора положительными, то максимум, что мы можем сделать c?=?а???min(аi/bi)?b. При этом одна из компонент c обратится в нуль. Естественно, так сильно раздвигать мы не хотим. Просто вычтем чуть-чуть первый из второго:
Можно, к примеру, вычитать средний вектор из всех, это не важно, главное как-нибудь увеличить угол между векторами. Свобода в этом шаге есть неизбежная плата за неопределённость задачи. Коэффициент 0?<?
Матрица
Разложение по базису это стандартная процедура. Единственная тонкость: из постановки задачи ясно, что коэффициенты разложения должны быть положительны. Линейная оболочка набора векторов с положительными коэффициентами это бесконечный симплекс (пирамида, вершина которой упирается в начало координат, а основание унесено на бесконечность). Грани этого симплекса суть симплексы меньшей размерности. Всё что нам надо — это проецировать заданный вектор на симплекс. Если вектор попадает внутрь симплекса (все коэффициенты разложения по базису положительны), то задача решена, если нет, то проецировать надо на грани.
Вот, собственно, вся функция:
Проверяем, что работает корректно:
Ниже комментарии для тех, кто хочет понять код. Коэффициенты разложения вектора v по базису e(i) называются контравариантными координатами v?=?vi?e(i). Прежде всего, вычисляем ковариантные координаты vi?=?(e(i),?v). Если метрика gij?=?(e(i),?e(j)) обратима, то контравариантные координаты вычисляем по обратной метрике gij=(g?1)ij, т.е. vi?=?gij?vj. Вот и всё. Флаг
Хотя все функции откомпилированы в C, Mathematica упорно не хочет запускать
Первая компонента:
Вторая компонента:

Можно. Пихайте в
Скачать файл Mathematica можно тут.

Простейший алгоритм, который приходит в голову, выглядит так:
- Определяем несколько базовых цветов картинки. RGB компоненты этих цветов будем использовать как базисные вектора.
- Цвет каждого пикселя разлагаем в линейную комбинацию базисных.
- Выводим изображение для каждого базисного цвета.
- Самооценка автоматически повышается.
Далее, более подробно по каждому пункту.
Базовые цвета
Базовые цвета это просто средние цвета сегментов изображения. Алгоритмов сегментации — море разливанное, выбирай на вкус, некоторые реализованы в Mathematica. Сразу скажу, что от выбора алгоритма сегментации мало что зависит, главное, чтобы число кластеров совпадало с желаемым числом компонент:
original =
Import["https://hsto.org/files/f42/962/1c5/f429621c55cd46a1beb4d4eb5aefed53.jpg"];
sizeX = First@ImageDimensions[original];
clusters =
ClusteringComponents[
original,
3,
Method -> "KMeans",
DistanceFunction -> CosineDistance
];
Ещё одна простенькая функция, которая нам понадобиться,
minIndex — возвращает позицию минимального элемента в списке, т.е., откомпилированная замена Composition[First, Ordering]:не ради правды, но ради истины
minIndex =
Compile[
{
{list, _Real, 1}
},
Module[
{
i = 0,
min = list[[1]],
minPos = 1
},
Do[
If[list[[i]] < min, minPos = i; min = list[[i]]],
{i, 1, Length@list}
];
minPos
],
CompilationTarget -> "C"
];
Теперь функция, которая возвращает базисные вектора.
removeDarkest удаляет самый тёмный цвет — это фон, он нам не нужен:removeDarkest[list_] := Delete[list, minIndex[Norm /@ list]]
getBasisColors[image_, clusters_] :=
Module[
{
data = ImageData[image],
components = Union[Flatten @ clusters]
},
Table[
Median[Join @@ Pick[data, clusters, component]],
{component, components}
]
]
Запускаем…
B1 = removeDarkest@getBasisColors[ColorNegate@original, clusters]
и, вуаля: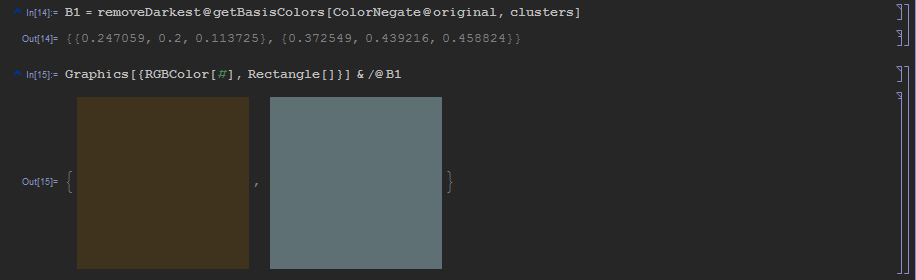
А не, погодите, не вуаля. Разлагать картинку по таким базисным векторам нет никакого смысла: полученные компоненты будут более менее повторять кластеры. Вот тут момент истины, т.е. единственный неизбежный эвристический шаг. Для того, чтобы компоненты картинки получились более полными, надо базисные вектора чуть-чуть раздвинуть.
Если мы хотим, из вектора а вычесть вектор b, при этом хотим сохранить компоненты вектора положительными, то максимум, что мы можем сделать c?=?а???min(аi/bi)?b. При этом одна из компонент c обратится в нуль. Естественно, так сильно раздвигать мы не хотим. Просто вычтем чуть-чуть первый из второго:
alpha = 0.5;
basis = {B1[[1]], B1[[2]] - alpha Min[B1[[2]]/B1[[1]]] B1[[1]]};
metric = Outer[Dot, basis, basis, 1];
Можно, к примеру, вычитать средний вектор из всех, это не важно, главное как-нибудь увеличить угол между векторами. Свобода в этом шаге есть неизбежная плата за неопределённость задачи. Коэффициент 0?<?
alpha?<?1 есть единственный подгоночный параметр алгоритма. Например, для того, чтобы получить картинку в начале поста, мне пришлось положить alpha?=?0.95.Матрица
metric это метрика линейной оболочки базисных векторов, она же матрица Грама, которая понадобиться нам в следующем разделе.Разложение по базису
Разложение по базису это стандартная процедура. Единственная тонкость: из постановки задачи ясно, что коэффициенты разложения должны быть положительны. Линейная оболочка набора векторов с положительными коэффициентами это бесконечный симплекс (пирамида, вершина которой упирается в начало координат, а основание унесено на бесконечность). Грани этого симплекса суть симплексы меньшей размерности. Всё что нам надо — это проецировать заданный вектор на симплекс. Если вектор попадает внутрь симплекса (все коэффициенты разложения по базису положительны), то задача решена, если нет, то проецировать надо на грани.
Вот, собственно, вся функция:
Выгладит длинно только из-за моего стиля писать код.
reduceMetric =
Compile[
{
{metric, _Real, 2},
{index, _Integer}
},
Transpose@Delete[Transpose@Delete[metric, index], index],
CompilationTarget -> "C"
];
getComponents =
Compile[
{
{vec, _Real, 1},
{basis, _Real, 2},
{metric, _Real, 2}
},
Module[
{
covariant,
contravariant = Table[0., {Length[basis]}],
flag = True,
subspace
},
covariant = basis.vec;
flag =
If[
Det[metric] != 0,
contravariant = Inverse[metric].covariant;
FreeQ[Sign[contravariant], -1],
False
];
Chop@If[
flag,
contravariant,
subspace =
Table[
Insert[
getComponents[vec, Delete[basis, i], reduceMetric[metric, i]], 0., i],
{i, 1, Length@basis}
];
subspace[[minIndex[-(subspace.covariant)]]]
]
],
{
{_minIndex, _Integer},
{_reduceMetric, _Real, 2},
{_getComponents, _Real, 1}
},
CompilationTarget -> "C",
RuntimeAttributes -> {Listable},
Parallelization -> True
]
Проверяем, что работает корректно:
getComponents[{0.1, 0.9}.basis, basis, metric]
{0.1, 0.9}
Ниже комментарии для тех, кто хочет понять код. Коэффициенты разложения вектора v по базису e(i) называются контравариантными координатами v?=?vi?e(i). Прежде всего, вычисляем ковариантные координаты vi?=?(e(i),?v). Если метрика gij?=?(e(i),?e(j)) обратима, то контравариантные координаты вычисляем по обратной метрике gij=(g?1)ij, т.е. vi?=?gij?vj. Вот и всё. Флаг
flag?=?False генерится в двух случаях: метрика необратима (симплекс меньшей размерности, чем мы думали) или хотя бы одна из контравариантных координат отрицательна (не попали внутрь симплекса). В этих случаях тупо рекурсивно перебираем все грани, и выбираем ту проекция на которую максимальна. Проекция на подпространство базиса e(i) это свёртка ковариантных и контравариантных координат vi?vi. Теперь точно всё.Результаты
Хотя все функции откомпилированы в C, Mathematica упорно не хочет запускать
getComponents на нескольких ядрах. Разбираться лень, пусть это останется на совести разработчиков. Поэтому просто перемалываем все 2736000 пикселей:pixels = Join @@ ImageData[ColorNegate@original];
components =
Table[
getComponents[pixel, basis, metric],
{pixel, pixels}
];
Первая компонента:
data1 = (({1, 0} #).basis) & /@ components;
ColorNegate[Image@Partition[data1, sizeX]]

Вторая компонента:
data2 = (({0, 1} #).basis) & /@ components;
ColorNegate@Image@Partition[data2, sizeX]

А можно больше?
Можно. Пихайте в
getComponents любой, даже вырожденный/избыточный базис, и будет вам счастье:Module[
{
basis = {{1, 0}, {0, 1}, {1, 1}},
metric
},
metric = Outer[Dot, basis, basis, 1];
getComponents[{0.1, 0., 0.9}.basis, basis, metric]
]
{0.1, 0., 0.9}
Скачать файл Mathematica можно тут.
Комментарии (7)

Grisha_Kirilin
04.09.2015 14:40Не, ну если цвета даны, то делать абсолютно нечего. Вот программа , вот результат. Просто нажимайте Evaluation>Evaluate notebook и смотрите результат в конце. А без цветов эту картинку разложить трудно — на ней почти нет голубого цвета.

Meklon
04.09.2015 14:50Я ж о том и говорю. Без опорных цветов невозможно (вроде бы) выделить исходные красители, так как нет точек изображения, где они изолированно присутствуют. Если очень надо, то можно попытаться найти максимально моноокрашенный кусочек и иногда даже почти полностью раскладывается. Но это не совсем правильный подход. Эталонные образцы все-таки нужны. Тот же эозин тупо диффузно все пропитывает. Да, результат лучше здесь выложи. У тебя заблокирован доступ.



Meklon
Прекрасно, спасибо) Буду изучать. Вообще крайне востребованный функционал для гистологии, на самом деле. Основная проблема — получение чистых векторов красителя. Нужно заморочиться и сделать отдельные срезы. Получить чистый краситель пипеткой на изображении в отдельном варианте почти невозможно. Почти сто процентов, что будет примесь второго.
Meklon
Вот еще тестовые образцы — поиграться. Гематоксилин/эозин. В качестве векторов можете взять уже разложенные образцы.



Grisha_Kirilin
Блин, комментарий не туда поместил. Программа и результат уровнем ниже.