Второе десятилетие 21 века отлично показало, как многие трудно формализуемые в рамках классических методов задачи могут быть решены с использованием искусственных нейронных сетей. Важно и то, что для таких решений уже давно было найдено свое применение в различных прикладных областях жизни современного общества, связанных с безопасностью, автоматизацией производственных и бизнес процессов, и просто привнесением большого количества удобств. Значительная часть таких решений располагается в области Computer Vision, где в качестве основного источника данных выступают визуальные образы (изображения, видео, карты глубины). ИИ-исследователи хорошо знакомы с проблемой острой необходимости достаточного количества качественных обучающих данных (ground truth examples) для тренировки нейронных сетей, ведь фактически, чем больше таких данных использовано при обучении, тем точнее предсказания модели. Мы в компании Intel используем разные источники таких данных. Один из основных — подготовка собственных данных силами внутренней команды разметки. Для облегчения этого процесса в нашей команде был разработан инструмент Computer Vision Annotation Tool (CVAT), исходный код которого был опубликован на GitHub. Продукт является частью экосистемы OpenVINO — набора инструментов для быстрой разработки эффективных приложений, использующих различные модели глубокого обучения. К настоящему времени CVAT успел набрать достаточно высокую популярность среди обычных и коммерческих пользователей по всему миру.
Немного истории
Однажды, после новогодних праздников один из таких пользователей обратился к нам с запросом: может ли CVAT работать с данными, представленными в формате медицинских изображений? Один из наиболее популярных форматов для хранения медицинских данных — это .dcm файлы, внутренний формат которых специфицирован стандартом Digital Imaging and COmmunication in Medicine (DICOM). CVAT, к сожалению, никогда не разрабатывался с целью поддержки такого формата данных, хотя в прошлом и применялся для разметки разных медицинских данных, например при разработке продукта NerveTrack. Были и попытки самостоятельно изменить исходный код CVAT для поддержки данных DICOM, одна из которых описана здесь. В последнем источнике отмечаются преимущества использования CVAT для этих целей: открытый исходный код, с возможностью доработки под конечного пользователя, хранение конфиденциальных данных локально на внутренних серверах и развертка в локальной или корпоративной сети с возможностью работы через браузер, поскольку конечным пользователям, которые не имеют продвинутых знаний в системном администрировании, сложно осуществлять установку и настройку самостоятельно.
С тех пор мы начали задумываться о том, что стоит научить CVAT работать с этим форматом и предоставить удобный интерфейс для разметки такого рода данных. Ведь, с одной стороны, доля медицины в ИИ, достаточно высока, а значит есть и спрос на подобного рода функциональность (особенно после начавшейся пандемии COVID 19). С другой стороны, на существующем рынке нами не было найдено многофункциональных (в сравнении c CVAT) и доступных решений, в особенности с открытым исходным кодом, для разметки DICOM датасетов. Есть достаточно хорошие решения, которые в основном предоставляют функциональность для обработки, хранения, управления и просмотра DICOM данных (pydicom, cornerstonejs, orthanc-server), простые аннотационные инструменты (md.ai), и комплексные решения с хорошим функционалом для разметки данных, но они являются коммерческими, с ограниченным функционалом в бесплатных версиях (medseg.ai).
Почему все не так просто
Наша команда провела исследование, в ходе которого мы накидали возможные высокоуровневые дизайн-решения (т.е. того, как поддержка DICOM форматов может быть реализована в существующей экосистеме CVAT) и столкнулись с двумя трудностями, которые в совокупности являются существенным препятствием для дальнейшей работы. Первая проблема заключается в том, что DICOM стандарт подразумевает огромную вариативность .dcm файлов. Например, стандарт подразумевает существование 79 DICOM модальностей, которые определяют контент DICOM файла (CT – компьютерная томография, CR – компьютерная рентгенография, LEN – линзометрия, MR – магнитно-резонансная томография и т.д.). Кроме того, DICOM стандарт определяет огромное количество разных атрибутов или тегов, некоторые универсальные, другие зависящие от модальности. Файлы также могут включать одно изображение или несколько (слайс), или несколько слайсов. Данные в этих изображениях, часто не интерпретируются как пиксели, а могут определять, например, физические значения измерений, произведенных тем или иным оборудованием. В конце концов, DICOM это не единственный формат файлов, используемых в медицине. Поддержка всех этих сценариев была бы чрезмерно объемной задачей для нашей команды. Да и нужно ли это?
Здесь мы плавно подходим ко второй проблеме. Обычно процесс разработки новых функций в CVAT основывается на том, что необходимо нашим пользователям в первую очередь. Например, значительная часть разработанных функций была добавлена с целью удовлетворения требований внутренней команды разметки, которая занималась подготовкой данных для обучения многих моделей в OpenVINO Model Zoo – зоопарке точных и высоко-оптимизированных моделей глубокого обучения. Такой подход подразумевает, что сперва какое-либо заинтересованное лицо обращается к нам с определенным запросом, разметить тот или иной набор данных тем или иным образом, чтобы на выходе получить разметку в определенном формате. После этого мы решаем конкретную задачу, а не пытаемся предугадать, что именно нужно пользователям. Однако, поскольку CVAT никогда и нигде не позиционировался как инструмент разметки медицинских данных, настоящее CVAT сообщество, глобально говоря, не слишком заинтересовано в реализации подобного функционала Таким образом у нас нет достаточно хорошего представления о направлении дальнейшей разработки. И одна из целей настоящего материала – собрать больше сведений о задачах, с которыми работает сообщество и востребованных функциях. Так, если вы заинтересованы в разработке, вы можете обратиться к нам через GitHub или прямым письмом (все ссылки и адреса предоставлены в конце статьи).
Тем не менее, это возможно
Статья не оправдала бы свое название, если бы закончилась на предыдущем абзаце. Несмотря на указанные проблемы, мы подготовили небольшое, «быстрое» решение проблемы разметки DICOM файлов в CVAT. Решение, конечно, не является самым удобным, но по крайней мере для самых простых случаев использования оно может оказаться применимым. Как было отмечено, DICOM файлы очень вариативны, но значительную долю проблем позволяют решить существующие решения, в том числе и с открытым исходным кодом. Так, мы использовали модуль языка программирования Python для работы с DICOM (pydicom) для подготовки скрипта, который осуществляет конвертацию DICOM файлов в обычные изображения и далее покажем подробный пример использования. Интерфейс командной строки для конвертации располагается в репозитории CVAT по ссылке. Скрипт доступен начиная с релиза 1.4. Набор команд ниже был протестирован на ОС Ubuntu 20.04, но действия по большей части простые и легко выполнимы в ОС Windows или других с помощью графического пользовательского интерфейса.
Для корректной работы команд, описанных ниже подразумевается, что в системе установлен ряд инструментов. Чтобы установить их в Ubuntu, вы можете воспользоваться следующей командой:
sudo apt install curl zip unzip python3 python3-pip python3-venv gitПервый шаг – склонировать репозиторий, если вы этого еще не сделали:
git clone --branch v1.4.0 https://github.com/openvinotoolkit/cvat.git cvat && cd cvatСледующим шагом необходимо перейти в папку с утилитой и рекомендуется создать виртуальное окружение Python, чтобы избежать установки лишних пакетов в систему, затем активировать его и установить необходимые зависимости:
cd utils/dicom_converter/
python3 -m venv .env
. .env/bin/activate
pip install -r requirements.txtТеперь можно запустить скрипт для того, чтобы преобразовать набор данных. Для примера возьмем набор данных CHAOS (Combined (CT-MR) Healthy Abdominal Organ Segmentation). Вы можете скачать его по предоставленной ссылке вручную, либо с помощью утилиты curl (Ubuntu):
curl -L https://zenodo.org/record/3431873/files/CHAOS_Test_Sets.zip?download=1 --output CHAOS_Test.zip
curl -L https://zenodo.org/record/3431873/files/CHAOS_Train_Sets.zip?download=1 --output CHAOS_Train.zipРазархивируем полученные данные с использованием графического интерфейса или с помощью командной строки Ubuntu:
unzip CHAOS_Test.zip -d CHAOS_Test
unzip CHAOS_Train.zip -d CHAOS_TrainИ сконвертируем их, запустив скрипт и передав нужные аргументы. Первый аргумент – корневой каталог с исходными файлами. Второй – корневой каталог для преобразованных файлов:
python3 script.py CHAOS_Train CHAOS_Train_converted
python3 script.py CHAOS_Test CHAOS_Test_convertedПримечание: Скрипт осуществляет рекурсивный поиск DICOM файлов. Структура каталогов в результате сохраняется.
Примечание: Если DICOM файл содержит несколько изображений, полученные изображения будут содержать постфикс с номером изображения. Например, многофреймовый DICOM файл с именем 055829-00000000.dcm будет преобразован в набор файлов 055829-00000000_000.png, 055829-00000000_000.png, ...:
В результате работы скрипта в каталогах CHAOS_Train_converted и CHAOS_Test_converted находятся обычные изображения, с которыми CVAT умеет работать. Далее в зависимости от ваших потребностей вы можете выбрать часть данных или все данные целиком и создать аннотационную CVAT задачу. Для удобства запакуем преобразованные файлы в архив с использованием графического интерфейса или с помощью командной строки в Ubuntu:
zip -r CHAOS_Train_converted.zip CHAOS_Train_converted/
zip -r CHAOS_Test_converted.zip CHAOS_Test_converted/Кроме того, каталог можно просто переместить без дополнительной архивации в CVAT хранилище (если оно подключено) и использовать соответствующую вкладку файлов в CVAT при создании задачи, либо загрузить изображения стандартными средствами браузера из любого места на компьютере, если работаете с линейным списком изображений (загрузка каталогов пока не поддерживается).
Теперь необходимо установить CVAT, если вы этого еще не сделали. Подробный гайд по установке и настройке, в зависимости от операционной системы, может быть найден в документации.
В установленном CVAT создадим проект DICOM, который включает пару абстрактных классов разметки (мы будем размечать нечто абстрактное, поскольку не являемся медицинскими экспертами и не ясно, что именно представляет область интереса в наборе данных), откроем его и затем создадим две задачи для двух архивов:


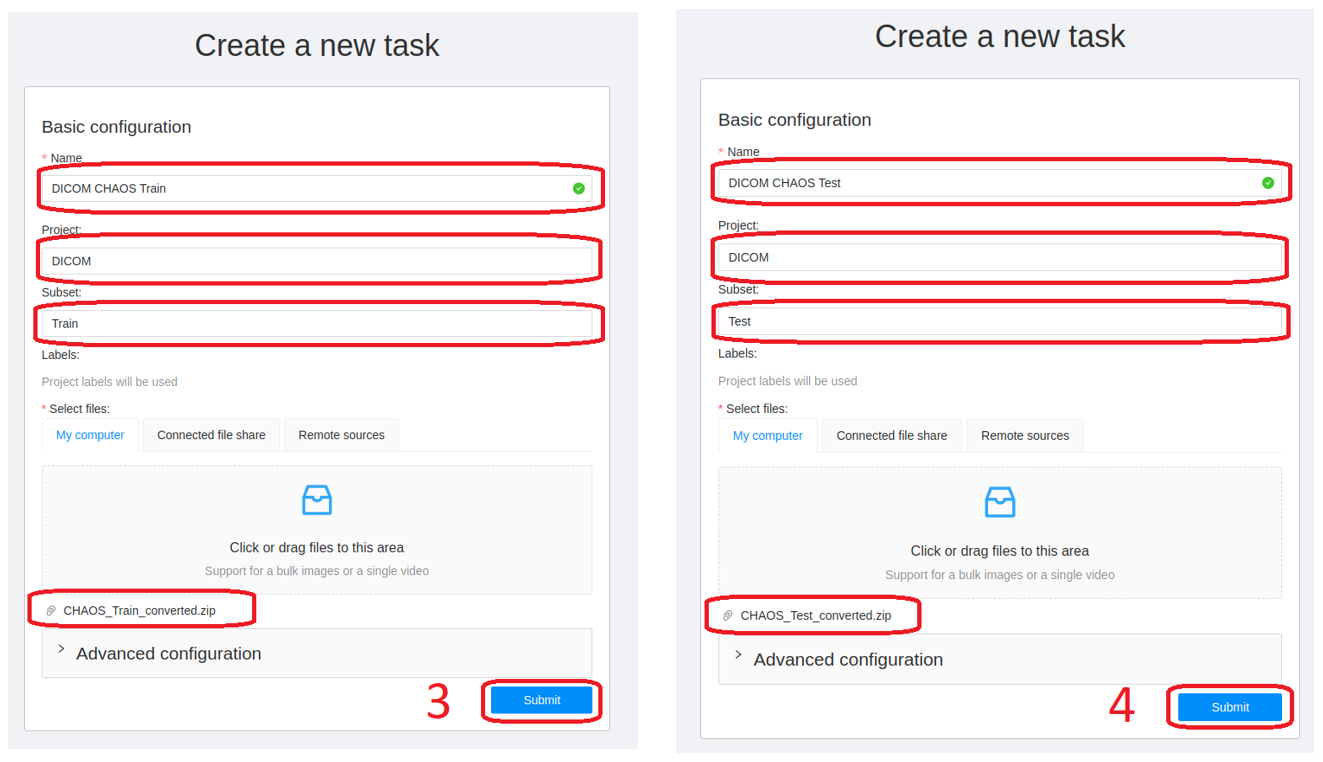
Теперь разметим абстрактные сущности на изображениях:

Кроме ручной разметки, можно использовать полуавтоматические методы:

Полуавтоматическая сегментация с использованием OpenCV JS выглядит эффективно в данном сценарии, поскольку у размечаемых объектов высокий контраст, а значит алгоритм работает довольно точно. Еще одно размеченное изображение:

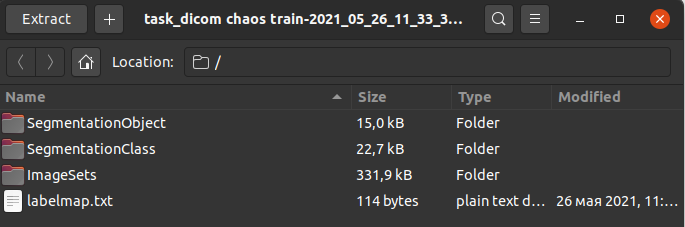
После того, как все данные размечены, можно получить результат в виде, например, PNG маски. CVAT поддерживает большое количество разных форматов, но, как показывает практика, маски довольно популярны при работе с DICOM данными.

Конечный файл содержит маски и другую полезную информацию:

В этом небольшом руководстве мы рассмотрели возможный сценарий разметки DICOM данных с использованием инструмента Computer Vision Annotation Tool. Кроме того, мы постарались объяснить как именно наша команда приходит к разработке новой функциональности, какое важное влияние в этом оказывают наши пользователи, их отзывы и описания задач, которые необходимо решить с помощью инструмента. Если вы желаете оставить отзыв по инструменту или данному материалу, либо предоставить ваши идеи по охваченной теме, пожалуйста, свяжитесь с нами, используя контакты ниже.
Computer Vision Annotation Tool: универсальный подход к разметке данных
Author: Борис Секачев boris.sekachev@intel.com
Team Lead: Никита Манович nikita.manovich@intel.com


aarner
Финальные маски очень очень далеки от сколь-нибудь точного результата. Вы уверены, что есть смысл размечать руками (полуавтоматический результат мягко говоря не далеко ушел)? Быстрый тест используя color-based сегментацию (потратил 5 минут, игнорируйте «выход за границы»):




Кстати, внутренние детали легко скрыть, если действительно нужно «упростить» результат:
Ну и уж точно руками вы не получите достаточно качественную семантику, например:
Уровень доступных деталей (маски сгенерировал как в векторе так и в растре):
Использовал вот этот исходник: www.youtube.com/watch?v=EHdyoYXo3bw