
Однако уже сейчас есть элементарный носитель, близкий нам в прямом смысле — простые молекулы ДНК. Исследователи уже несколько десятилетий работают над записью информации в ДНК, здесь ничего нового. Стандартный способ записи — синтез нуклеотидных цепочек (A, T, C, G), а чтение производится путём секвенирования. Недавно разработан альтернативный подход: чтение оптическим способом. Прогресс в этой области нешуточный.
Преимущества ДНК
 Молекулы ДНК (дезоксирибонуклеиновой кислоты) — великолепный материал для цифровой памяти. Физически это длинная макромолекула, которая состоит из четырёх повторяющихся блоков (нуклеотидов) — именно их мы используем для кодирования данных.
Молекулы ДНК (дезоксирибонуклеиновой кислоты) — великолепный материал для цифровой памяти. Физически это длинная макромолекула, которая состоит из четырёх повторяющихся блоков (нуклеотидов) — именно их мы используем для кодирования данных.Макромолекула сворачивается в форму двойной спирали и выглядит довольно красиво, особенно с учётом её значения как носителя генетической информации, то есть эссенции жизни.
Фантастическая информационная плотность (215 петабайт на грамм), длительный срок хранения информации и низкое энергопотребление — преимущества таких «накопителей» на практике. Стоимость синтеза и секвенирования ДНК снижается быстрыми темпами. Рост производительности синтеза и секвенирования опережает закон Мура. Это стимулирует инновации в разработке технологий так называемой нуклеотидной памяти. Также стоит отметить и стабильность такого варианта хранения информации, учитывая плотность, так как отсутствует потеря данных при записи.
Перспективная технология
Над технологиями синтеза и секвенирования, улучшением алгоритмов чтения/записи и коррекции ошибок работают десятки научных коллективов по всему миру.
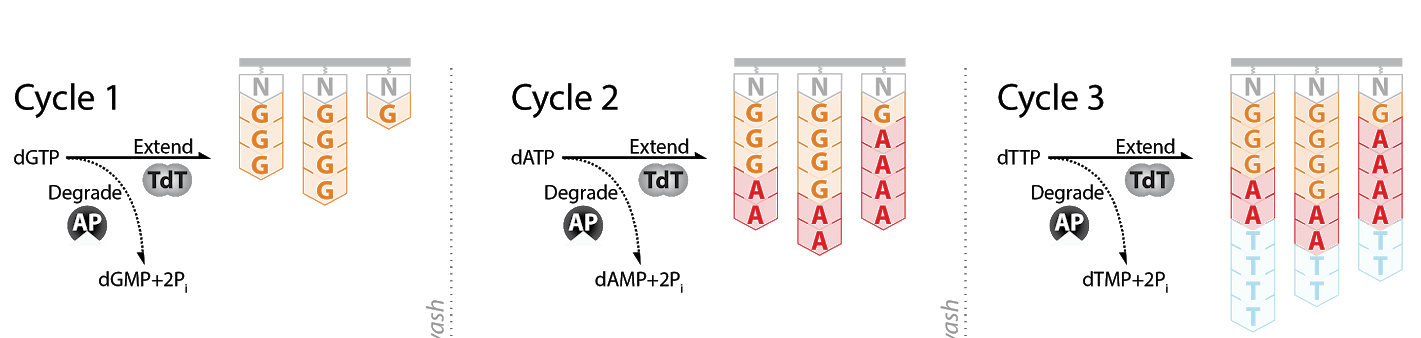
Например, оригинальная технология ферментного синтеза предполагает использование ферментов TdT и AP для добавления и удаления нуклеотидов тремя способами. Здесь трёхбитная система, потому что нуклеотид C не появляется.
Другие исследователи специализируются на оптимизации алгоритмов коррекции ошибок. Авторы этой работы закодировали и записали в ДНК 35 файлов общим размером 200,2 МБ (в сжатом виде), а затем прочитали их без ошибок.
| Данные | Размер файла | Количество последовательностей ДНК |
| HD-видео | 44,2 Мб | 3,2 миллиона |
| Классическая музыка |
13,9 Мб | 890 000 |
| Текстовая информация | 11,1 Мб | 708 000 |
| Все 35 файлов | 200,2 Мб | 13,4 миллиона |

Давно разработана технология для внедрения синтезированной ДНК в геном живой клетки (CRISPR).
Таким образом, запись файлов в ДНК — вполне реальная возможность. По сути, эту технологию уже можно использовать в продакшене. Скорее всего, первые автоматические коммерческие установки для записи/чтения больших объёмов информации в ДНК появятся в ближайшие годы.
Альтернативный подход
Отдельно стоит упомянуть самую последнюю инновацию в этой области — технологию так называемой «цифровой нуклеотидной памяти» (digital Nucleic Acid Memory, dNAM). Научная статья «Альтернативный подход к нуклеотидной памяти» опубликована в журнале Nature Communications совсем недавно — 22 апреля 2021 года.
Идея состоит в том, что запись информации производится своеобразными пегами из ДНК на пегборде, а чтение — с помощью микроскопа. То есть не требуется секвенирование. Созданный учёными прототип сохраняет информацию в виде паттернов, используя нити ДНК, расположенные на расстоянии около 10 нанометров друг от друга.

Тут аналогия с детской игрой Lite-Brite. Пегборд в игре представляет собой световой короб с маленькими цветными пластиковыми «прищепками» (пегами), которые вставляются в панель и светятся, создавая освещённую картинку, либо используя один из прилагаемых шаблонов, либо создавая изображение «свободной формы» на чистом листе чёрной бумаги.
Поэтому в научной работе и упоминается термин «пегборд».
Предыдущие методы получения данных из ДНК требовали секвенирования. Секвенирование — это процесс считывания нуклеотидной последовательности нитей ДНК. Довольно трудоёмкая процедура. Генетический материал распределяют по пробиркам, туда добавляют формамид, проводят электрофорез, а на последнем этапе — радиоавтографию, которая и определяет последовательность нуклеотидов.
Хотя это мощный инструмент в медицине и биологии, он не подходит для высокопроизводительной и скоростной работы. То есть не оптимален для считывания цифровой информации из ДНК.
Здесь же информация считывается с помощью микроскопа оптическим способом. Поскольку пеги ДНК расположены ближе, чем половина длины волны видимого света, учёные использовали микроскопию сверхвысокого разрешения, которая обходит дифракционный предел света. В данном случае используется микроскопия сверхвысокого разрешения типа DNA-PAINT.

Паттерны из нитей ДНК (пеги) светятся, когда с ними связывается флуоресцентно меченая ДНК. Поскольку флуоресцентные нити короткие, они быстро связываются и расцепляются. Это заставляет их мигать, так что их проще различить — и считать информацию. Светящиеся паттерны каждого пегборда — это код для хранения фрагментов данных.

В результате эксперимента сообщение было успешно прочитано. Алгоритм декодирования выполнил коррекцию ошибок и успешно восстановил сообщение (синие квадратики на иллюстрации (g) указывают на исправленные ошибки).
Микроскоп может зафиксировать сотни тысяч ДНК-пегов с одного изображения. Данный прототип смог считать данные с плотностью 330 гигабит на квадратный сантиметр (для сравнения, на самой продвинутой магнитной плёнке плотность записи составляет около 31 гигабита на см²). А вообще, теоретически расстояние между точками в 10 нанометров соответствует плотности примерно 1 тыс. Гбит на см², просто две трети точек выделено под индексы и коррекцию ошибок (см. ниже).
Но даже 330 гигабит на квадратный сантиметр — это огромная цифра. Если мы не ошибаемся в расчётах, то с заявленной информационной плотностью на странице А4 размером 21×30 см поместится 00,33*21*30/8 = 25,9875, то есть почти 26 терабайт! Под микроскопом закодированная информация выглядит примерно так:

Учёные надеются в будущем увеличить объём данных, которые можно хранить в dNAM, уменьшить время, необходимое для записи и чтения, а также улучшить кодирование данных. «Можно увеличить ареальную плотность dNAM, разместив домен данных на каждом витке спирали ДНК (расстояние между витками ~3,5 нм)», — сказано в научной работе.
Применение
ДНК отлично подходит для долговременного хранения большого количества файлов. Вся информация в мире (порядка 100 зеттабайт, то есть примерно 100×10²¹ байт) поместится в маленькой шкатулке на вашем столе. При этом информация в ДНК может храниться веками: период распада молекулы ДНК превышает 500 лет. Правда, время доступа к файлам при стандартных технологиях секвенирования — десятки часов.
Примечание. Геном человека занимает 3,3 ГБ, домовой мыши — 2,7 ГБ, гриба — 30 МБ, вирусы обычно укладываются в несколько килобайт.Альтернативный вариант dNAM имеет немного другую сферу применения. Эта технология скорее нацелена на варианты с ограниченным количеством информации, но высокой информационной плотностью. То есть «напечатать» на листе 1-2 петабайта данных, а потом относительно быстро их прочитать. Понятно, что это уже иная область использования.
Плюсы:
- Крайне малое место для хранения данных (помним про шкатулку).
- Сохранение данных в течение долгого срока.
Минусы:
- Дороговизна процесса записи.
- Долгий процесс считывания информации.
Общий вывод: Технология только-только начинает развиваться. Скорее всего, такой формат записи данных будет использоваться только в научных исследованиях и каких-нибудь специфических приложениях, связанных с биотехнологиями, ну или какими-нибудь крайне богатыми, но эксцентричными людьми. Если вам когда-нибудь захочется сделать себе ДНК-флешку, знайте, чтобы заплатить за 1 МБ данных, вам придётся выложить около 3,5 тысяч $. Не очень приятная стоимость, если вы хотите организовать семейный фотоархив :) Но, несмотря на это, плотность записи позволяет решить задачу поистине мировых масштабов, при помощи такой технологии можно сделать архив всей информации, которую накопил человек за время своего длительного существования.
Ранее мы рассказывали, как сделать свой архив интернета, а также поднимали тему веб-сервера на старом смартфоне Android. А какие альтернативные способы хранения информации вы знаете? Пишите в комментариях.
НЛО прилетело и оставило здесь промокоды для читателей нашего блога:
— 15% на все тарифы VDS (кроме тарифа Прогрев) — HABRFIRSTVDS.
— 20% на выделенные серверы AMD Ryzen и Intel Core — HABRFIRSTDEDIC.
Доступно до 31 декабря 2021 г.
Комментарии (14)

GritsanY
22.11.2021 11:35+1В книге Эхопраксия Уоттса упоминаются довольно печальные последствия такого способа хранения информации, если он получит большое распространение.

shadrap
22.11.2021 12:55+1Хочется ответить словами тов. Саахова - "...Все эт конечно правильно, ноо..."
Хорошая тема - изнасиловать каждое открытие до конца , авось что-то и появится дельное. но пока профит в использовании ДНК в качестве носителя информации не ясен. Система Днк-РНК вообще хороша не только сама по себе , а прежде всего ее способностью быть регулированной и компактной. Я не знаю , какие способы синтеза изобрели товарищи , которые 200mb загнали в ДНК, но на сегодняшний день олигонуклеотидный синтез это очень дорогое и очень сложное мероприятие. Коммерческий синтез цепочек длиннее 200бп стоит довольно дорого и занимает двольно много времени... Для общего представления синтез кластера или генома размером до 10кбп займет 3-5 недель и стоить будет под десятку тысяч долларов, да скорее всего для использования как data storage не нужна будет 99% точность, но за неточность придется то же платить длиной( CRC code и тп). К тому же вся эта фосфорамидная химия сильно подвержена влиянию окружающей среды, особенно для очень длинных цепочек.
Вопрос считывания это то же себе вопрос- нанопроное секвенирование конечно подойдет в этом случае , но у него пока в целом совсем беда с точностью, а NGS как таковой - хорош только для одноразового чтения -кодирования, потому как радио или просто фосфоресцирующий квенчер улетел с нуклеотида и все... - второго квенчера там нет, следовательно более мы эту закодированную в ДНК цепочку не прочитаем без повторного "тэгирования" с флуоресцирующим DNTP .
хранение же DNA на базе dNTP (это нуклеотид с фосфоресцирующей меткой) довольно сильно отличается от хранения DNA в обычном формате , в каком оно хранится в клетке - накрученом на гистоны и свернутом в нуклеосомы .., там совсем другая химия.
Итого - в целом конечно очень круто , но в деталях - очень сложно дорого и долго писать - острожно и бережно хранить - следовательно то ж не дешево и дорого, медленно и нудно читать . Возможно я просто чего то недопонимаю.

Ad_Infinitum
22.11.2021 13:31+2Итого - в целом конечно очень круто , но в деталях - очень сложно дорого
Со временем технологии дешевеют. Не успеете опомнится, как молодёжь будет ходить с плеерами читающими музыку на прямую из котиков, пёсиков, а может и самих себя. (Утрирую. А может и нет... :))
P.S.: Представил рынок где торговцы продают из под полы запрещёнку записанную на улитках.

5Мб диск 50-х годов. Круто, дорого, очень сложно. 
shadrap
22.11.2021 15:03там были очевидные неоспоримые плюсы, да было дорого и "много" , но быстрее всех существующих аналогов и след в будущее явно прослеживался. Здесь то какие плюсы?
Скорее уж белковый сторадж будет эффективнее.

Ad_Infinitum
22.11.2021 13:38+1Кстати прививочный паспорт при такой технологии можно вживлять вместе с вакциной в ближайшие ткани возле укола. При таком подходе ни один противник прививок не захочет паспорт подделывать. И не благодарите :)

Sergdesign
22.11.2021 13:52+1Плюсы технологии — золотую коллекцию СоюзМультфильма можно записать сразу в сына.
Минусы технологии — случайный документ, записанный в домашнее ДНК хранилище, может спровоцировать у него отрастание ног и хвоста.

resk0
22.11.2021 15:40+1Наверное проще было бы использовать для таких целей полимерные молекулы - намного выше плотность. Не знаю как цена технологий создания-чтения, но сомнительно что дороже.
Какой-то там С(СН3)-С(СН2ОН)-С(СН3)...

mattroskin
22.11.2021 18:49А что с ошибками считівания? Насколько я понимаю, после нескольких копирований записанная информация будет существенно отличаться от оригинала.

humanzer0
23.11.2021 12:32Такой вид памяти вполне может использоваться, допустим, в медицинских нанороботах, лечащих человека от различных болезней при их введении в кровь.



Ad_Infinitum
100 лет спустя...
Тот момент, когда ты узнал, что родители в тебя (ещё на этапе яйцеклетки) зашили свою любимую песню, но ты ненавидишь попсу :)