
Всем привет! Рассмотрю два популярных алгоритма уменьшения размерности, а именно T-distributed Stochastic Neighbor Embedding (t-SNE) и Uniform Manifold Approximation and Projection (UMAP). Их удобно использовать, когда необходимо визуализировать данные с большим количеством параметром (также будем называть это размерностью данных).
Оба алгоритма осуществляют преобразование данных большой размерности в меньшую. На выходе, обычно, получают два измерения, оси которых или расстояния между полученным объектами не поддаются прямой интерпретации, в отличии, например, от метода главных компонент (Principal component analysis, PCA).
Указанное выше не значит, что PCA лучше рассматриваемых нами алгоритмов. По своей сути они различны. PCA является линейным алгоритмом, присваивающим равные веса всем попарным расстояниям. Что касается t-SNE и UMAP, то они не линейны, и умеют определять приоритеты расстояний между соседями, что дает возможность выявить внутреннюю двумерность данных. При этом отличие t-SNE от UMAP заключается в том, что последний лучше сохраняет глобальную структуру при выводе итоговых результатов.
Сравним оба алгоритма на примере базы изображений COIL-20, созданной сотрудниками лаборатории CAVE Колумбийского университета, США. База содержит изображения 20 предметов по 76 штук изображений на каждый из 20 предметов Рис 1.

Для того, чтобы иметь возможность применить рассматриваемые алгоритмы необходимо изображения предобработать, например, привести их в пиксельные значения. Используем для этого код ниже.
data = []
path = 'datasets/coil-20-proc/'
for file in os.listdir(path):
im = Image.open('datasets/coil-20-proc/'+file)
pixels = list(im.getdata())
cl = int(file.split('__')[0][3:])
data.append([cl] + pixels)
df = pd.DataFrame(data)
df.columns = ['label'] + ['a{}'.format(i) for i in range(df.shape[1]-1)]Мы получили матрицу с параметрами рассматриваемых изображений и теперь можем применять t-SNE и UMAP.
tsne = TSNE()
embedding_tsne = tsne.fit_transform(df.drop('label', axis = 1))
umap = UMAP()
embedding_umap = umap.fit_transform(df.drop('label', axis = 1))
Заметно, что UMAP уплотнил данные и развел большинство из них сильнее, чем t-SNE (рис 2). При этом UMAP затратил на работу 29 секунд, а t-SNE около 103 секунд. С этой точки зрения UMAP выглядит предпочтительнее. Однако, существует мнение, что такое сравнение не корректно, из-за стохастического характера t-SNE. Для нивелирования данного момента необходимо сделать t-SNE детерминированным, инициализировав через PCA. Давайте попробуем это сделать, применив PCA также к UMAP.

Хотя данные изображения визуально выглядят приятнее, но получились они менее плотные (рис.3). Попытки поработать с параметрами алгоритмов t-SNE и UMAP в контексте инициализации через PCA существенного изменения изображений выше не дали. Таким образом можно сделать вывод, что для нашего случая применение PCA не оправдано. А значит вывод о предпочтительности UMAP, остается прежним.
Давайте рассмотрим основные параметры алгоритма UMAP:
A) n_components – это размерность итоговых данных. Для двухмерного представления необходимо установить значение 2, для трехмерного, соответственно, 3.
B) n-neighbours – определяет работу алгоритма с глобальной структурой данных. Значение по умолчанию установлено 15. Чем большая величина задается, тем большее количество соседних значений рассматривает UMAP, то есть смещает свое внимание с локальной структуры данных в пользу глобальной.
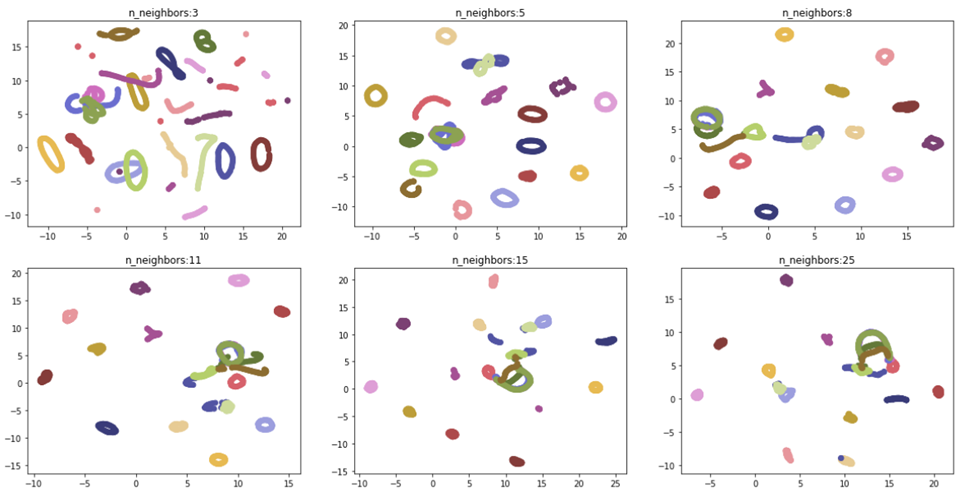
На рисунке 4 видно, что данные лучше всего разделились при значениях n_neighbors равным пяти и восьми. Это обусловлено небольшим размером рассматриваемого нами датасета.
C) min_dist – параметр задает минимальное расстояние для точек в итоговом представлении меньшей размерности. По умолчанию величина 0.1.

При устремлении параметра к нулю алгоритм начинает искать различия внутри полученных кластеров. При увеличении – внутренние различия стираются и структура данных представляется в виде единого целого (рис.5).
D) spread – задает максимальное расстояние между точками в группе. По умолчанию значение равно 1. В сочетании с min_dist определяет на сколько сгруппированы и разгруппированы точки.

В сочетании с min_dist, позволяет более тонко балансировать между отражением внутренней структуры и глобальной картины. (рис.6)
В завершении хотелось бы добавить, что UMAP успешно справляется не только с картинками, но и векторизованными текстами, социологическими опросами и т.п., позволяя сразу сформировать общее представление о данных, не вчитываясь в них. Данный инструмент значительно превосходит конкурентов по скорости и лучше отражает внутреннюю структуру данных.

