
Компьютерное зрение и машинное обучение находят своё применение во многих сферах деятельности человека. Исключением не стала и медицина. В этой статье рассматриваются наиболее интересные, на взгляд автора, задачи компьютерного зрения в медицине.
Автоматическое детектирование циркулирующих опухолевых клеток
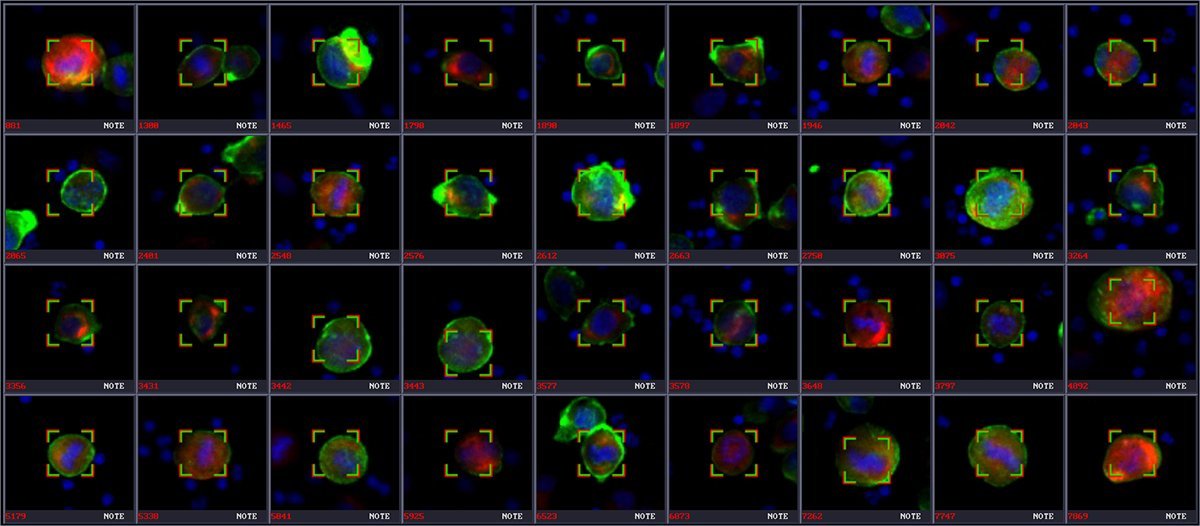
Циркулирующие опухолевые клетки скрестившиеся с с несколькими флуоресцентными антителами
Циркулирующие опухолевые клетки — это клетки, которые отделяются от места основной опухоли и распространяются через кровоток, образуя вторичные опухоли в других органах.
Раннее обнаружение таких клеток и оценивание прогрессирования болезни очень важно для эффективного лечения, поэтому сейчас активно разрабатываются системы для автоматического обнаружения опухолевых клеток. Например, исследователи из Германии [2] получили accuracy > 99%, recall = 88% и precision = 86% на экспериментальном датасете небольшого размера.
Источники:
- Automated Detection of Circulating Cells Using Low Level Features
- Automated Detection of Circulating Tumor Cells with Naive Bayesian Classifiers
- bioview.com/applications/circulating-tumor-cells
Автоматическое обнаружение диабетической ретинопатии
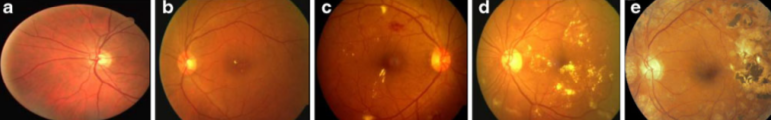
Классификация изображений в зависимости от степени заболевания: a) normal; b) mild; c) moderate; d) severe; e) prolific
Авторы соревнований на kaggle по обнаружению диабетической ретинопатии утверждают, что 40-45% американцев, страдающих диабетом, также подвержены и диабетической ретинопатии (думаю, что в России показатели примерно такие же). Прогрессирование ухудшения зрения может быть замедленно или предотвращено, если вовремя обнаружить заболевание. Таким образом, разработка систем по обнаружению диабетической ретинопатии также является актуальной.
Самый лучший результат quadratic weighted kappa на соревнованиях составил 0.84958 (Private Leaderboard). Некоторые исследователи разрабатывают адаптеры для смартфонов для получения снимков сетчатки. Делают они это при помощи 28D или 40D линз (стоят такие линзы около 300$) и насадки для смартфона, которую печатают на 3d принтере.
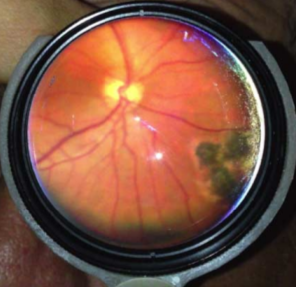
Адаптер для получения снимков сетчатки
Как видно, возможность проверки состояния своей сетчатки без похода в больницу уже не за горами.
Источники:
- www.kaggle.com/c/diabetic-retinopathy-detection
- Algorithms for the Automated Detection of Diabetic Retinopathy Using Digital Fundus Images: A Review
- 3D Printed Smartphone Indirect Lens Adapter for Rapid, High Quality Retinal Imaging
- Smartphone fundoscopy. Ophthalmology
Сегментация МРТ изображений
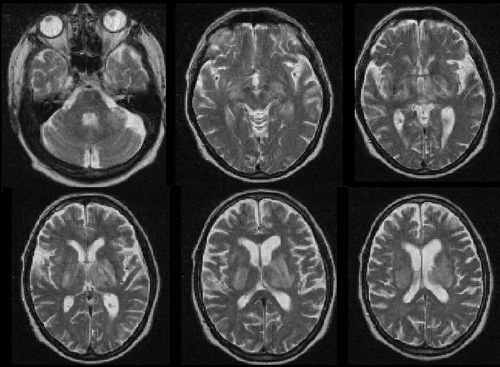
Снимки МРТ головного мозга
Метод магнитно-резонансной томографии (МРТ) широко применяется для диагностирования и отслеживания динамики заболеваний мозга, а также для исследования его функционирования. Метод позволяет получать трехмерные изображения высокого качества и разрешения, которые строятся по набору последовательных двумерных «срезов». Разметка изображений МРТ мозга на анатомические структуры является важным шагом для дальнейшего анализа во многих задачах в этой области.
Полная разметка трехмерного изображения предполагает разбиение (сегментацию) объема мозга на несколько десятков регионов, соответствующих основным анатомическим структурам. Каждой точке (вокселю) ставится в соответствие метка анатомической структуры. Таким образом, ручная разметка в данном случае становится длительным и трудоемким процессом. Поэтому необходимы алгоритмы, автоматизирующие процесс анатомической разметки. [1]
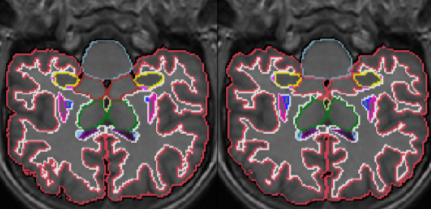
Пример работы алгоритма сегментации. Слева — истинная разметка; справа — полученная разметка
Авторам из [1] удалось получить следующую точность сегментации для анатомических структур(показатель DSC — мера сходства Дайса):
- Мозжечок — 0,885 ± 0,05
- Паллидум — 0,7442 ± 0,009
- Желудочек — 0,9 ± 0,02
- Кровеносные сосуды — 0,2 ± 0,001
- Средний мозг — 0,8474 ± 0,0073
Источники:
- А. Ю. Зубов, О. В. Сенюкова. GraphiCon 2015. Сегментация изображений магнитно-резонансной томографии головного мозга с помощью сопоставления с несколькими атласами
- Sequence-independent segmentation of magnetic resonance images
Анализ ультразвуковых изображений
Для примера возьмём задачу по нахождению нервной структуры шеи с недавних соревнований на kaggle.
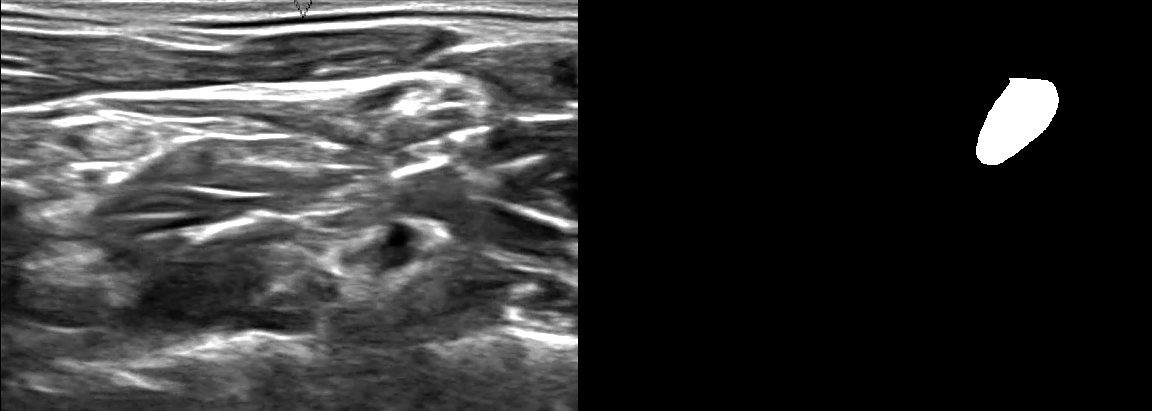
Слева — ультразвуковое изображение шеи, справа — нервная структура; на изображениях видно, что нахождение нервной структуры является нетривиальной задачей даже для человека (не являющимся специалистом в этой области)
Точное нахождение нервной структуры на ультразвуковых изображениях является важных этапом в эффективной вставке катетера для блокирования или уменьшения боли. Такие катетеры, в частности, помогают пациентам с наркотической зависимостью быстрее восстанавливаться.
Наилучший результат по мере сходства Дайса DSC на kaggle составил — 0.73226.
Источники:
Анализ лазерных спекл-картин
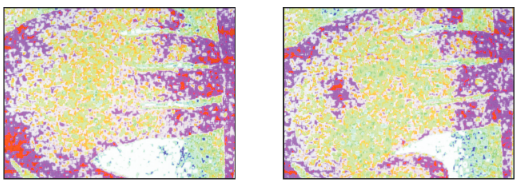
Лазерная спекл-картина руки, показывающая перфузию до и после растирания небольшой области руки
Автоматический анализ лазерных спекл-картины применяют для измерения кровотока. В частности, это помогает при лазеротерапии повреждённых тканей.
Источники:
- Laser speckle contrast imaging for measuring blood flow
- Laser speckle contrast imaging in biomedical optics
Заключение
Рассмотренные задачи лишь капля воды в море. В области анализа медицинских изображений большой простор для исследований.
Бурное развитие deep learning помогает постепенно улучшать точность разрабатываемых систем для анализа медицинских изображений, которые возможно вскоре будут применяться повсеместно. А это, несомненно, повысит уровень здоровья населения.
Комментарии (13)

ternaus
04.09.2016 22:09+3Предсказание эпилептических припадков?
https://www.kaggle.com/c/melbourne-university-seizure-prediction
JamaGava
04.09.2016 22:28+2Да, кстати, ещё одна большая группа задач — анализ биомедицинских сигналов, в том числе многоканальных (ЭЭГ, ЭКГ).
Туда же анализ ЭКГ и предсказание фибрилляций сердца.
А ещё знаю группу, которая занималась распознаванием заболеваний речевого тракта по анализу записи голоса. Они распознавали не просто простуды, а серьёзные болячки, вплоть до опухолей, причём дифференцировали их друг от друга. Планировали в будущем сделать такой анализатор в смартфоне для персонального пользования (для контроля здоровья). И даже планировали в голосе прослушивать модулированные тоны сердца тоже для контроля состояния, как-то так… я тогда ЭКГ занимался, в акустику сильно не углублялся.
К сожалению, не стало руководителя группы Бовбеля Евгения Ивановича, светлая память. Не знаю, продолжил ли кто-то из научного коллектива эти разработки по анализу биосигналов…
elingur
05.09.2016 11:40+2добавлю сюда ссылочку на небезызвестного в data mining Воронцова К.В. Применение машинного обучения и вычислительной лингвистики для диагностики заболеваний по электрокардиограмме

AllanStark
05.09.2016 09:21+3Аналогичными задачами озаботились в игре EVE Online — скрестили приятное с полезным, онлайновую игрушку с реальной наукой…
Project Discovery

max_fruit
05.09.2016 11:15+1Меня, как практикующего патологоанатома, интересует возможность распознавания гистологических микропрепаратов. Самых обычных, окрашенных гематоксилином и эозином, тех, которые составляют основную часть работы рядового патологоанатома. Пока не встречались упоминания работ в этом направлении, хотя, как мне кажется, сделать это очень и очень сложно, т.к. для каждого диагноза выборка, которой будут обучать нейросеть, должна быть просто колоссальных размеров. Была наивная мысль что-то попробовать самому сделать в этом направлении, поэтому начал понемногу изучать программирование, но, по мере изучения материалов, связанных с deep learning, пришел к выводу, что такие задачи под силу только каким-нибудь НИИ. Хотя, может я и ошибаюсь.

sergeypid
05.09.2016 13:39Задача собрать выборку образцов для каждого диагноза — непосильная для тех, кто умеет обучать нейросети.

max_fruit
05.09.2016 20:28Т.е. Вы имеете в виду, что необходима кооперация тех, кто может создать такую выборку, и тех, кто может обучать нейросети?

sergeypid
06.09.2016 08:06Конечно. Машинное обучение — это инструмент. Вы ведь не хотите изучить оптику и ремесло шлифовщика стекла, чтобы использовать микроскоп. Зато если вы соберете годный датасет и опубликуете его, весь мир машинного обучения набросится на него и будет сражаться за сотые доли процента точности распознавания, вам не надо будет ни копейки за это платить )))
Для того, чтобы нейросеть надежно распознавала образы, нужны огромные обучающие выборки. Весь нынешний прогресс нейросетей начался с того, что был собран ImageNet — 1.2 млн. изображений. Не уверен, что вы сможете собрать по 1000 РАЗЛИЧНЫХ образцов изображений клетки каждого диагноза (извините, я не специалист, формулирую медицинские понятия неправильно). Тут очень простые среднепотолочные соотношения: собрали по 100 образцов — получили точность 75%, по 1000 — 95%.
Gryphon88
05.09.2016 14:43Можете пояснить, что имеете в виду под «распознаванием», выделение границ клеток, отсев мусора, бинаризация+типирование клеток+поиск атипичных клеток, или вообще постановку диагноза? Для малого увеличения и апертуры в проходящем свете (>x20, >NA0.5) в области бинаризации сделано довольно много, по типированию я тоже читал статьи с адекватными картинками…
Основной геморрой — это выбор критерия для типирования. Например, есть у нас клетка Пирогова-Лангганса, она вполне себе узнаваемая «глазом», но критерий под неё писать не совсем очевидно как. Ну, большая, ну многоядерная, но она такая не одна так выглядит, особенно если препарат не очень или свет косо выставлен. Конечно, мы можем использовать нейросети, но, чисто иррационально, я им не доверяю. Мы можем получить сильно разные результаты в зависимости от окраски и условий съёмки/просмотра, и это не считая случаев, когда биоптат неинформативен.
max_fruit
05.09.2016 19:45Честно сказать, про критерии для отдельных клеток я даже и не думал, т.к. сам совершенно не представляю как можно прийти здесь к какой-то чёткой формулировке, которая будет понятна машине. И если у клетки Лангханса хотя бы есть более-менее строгий, так сказать, визуальный признак — периферическое расположение ядер (опять же не совсем строгое), то у, например, эпителиоидной клетки таких четких визуальных признаков нет, кроме многоядерности. Тут я с Вами полностью согласен.
Как это виделось мне (заранее прошу прощения за поток фантазии, который, скорее всего, далек от реальности):
Отличным материалом для первых шагов в этом направлении мне виделись либо соскобы из полости матки, т.е. эндометрий, либо биоптаты с влагалищной части шейки матки, т.е. экзоцервикс. Это один из «рутинных» материалов, его много, так же много и шаблонных заключений по нему. О распознавании (не уверен, что это удачный термин, но буду пользоваться им) отдельных клеток, повторюсь, я даже и не думал. А вот подсчет плотности расположения, например, эндометриальных желез на заданном количестве полей зрения и при определенной степени увеличения и, соответственно, определение наличия/отсутствия гиперплазии эндометрия, мне изначально казался вполне реальным. Или же определение наличия эндоцервикального эпителия в экзоцервиксе, что соответствует эндоцервикозу, который, в свою очередь, является одним из самых распространенных диагнозов в работе рядового патологоанатома. Т.е. работа с довольно крупными структурами — железами или же целыми пластами эпителия.
Но тут я сам себя завел в дебри рассуждений: для полноценной постановки диагноза необходимо не только установить наличие/отсутствие гиперплазии, но также важно определить простая она или сложная, есть атипия или нет и т.д, а это всё — дополнительные признаки, которые всё более усложняют процесс обучения нейронной сети (если мы используем нейронную сеть). Но даже если оставить в качестве задачи только определение наличия/отсутствия гиперплазии, то тут же возникают сложности в дифференциальной диагностике с тем же эндометриальным полипом, одна из форм которого очень и очень похожа на ЖГЭ. Т.е. со временем мне начало казаться, что огромное количество нюансов, которые патологоанатом улавливает, основываясь на собственном опыте и опыте коллег, делает работу по созданию системы, ставящей даже элементарные, с точки зрения врача, диагнозы, невероятно трудоемкой. Я не говорю, что это нереально. IT-сфера всегда доказывала, что фантастика может легко становиться реальностью. Но мне сложно представить необходимые ресурсы (человеческие и технические), которые потребуются для воплощения этой задумки. И это даже при работе с такими крупными структурами как, например, железы.
Что говорить тогда о диагностике опухолевого роста, где порой приходится искать отдельные атипичные клетки на больших участках, или же где тангенциальный срез может дать ложную картину инвазии?
Прошу прощения, что получилась целая «простыня» текста, причем сумбурного. Подытожу так: я прихожу в замешательство, начиная думать о критериях «распознавания», выше описал почему.Gryphon88
06.09.2016 00:57Сразу оговорюсь, я по образованию гистолог (2 года анатомии+патанатомии, потом временами пил с патанатомами), занимался онкологией, гематологией и немного туберкулёзом, соскоб матки при личной встрече не узнаю :) Если скинете пару картинок (не образцовых, их учебника, а как их приносят Вам в укладках), то можно покумекать. В принципе, задача не выглядит нерешаемой, если картинка нормальная.
Про нейросети и классификаторы. Насколько мне известно (последний обзор делал в 2014), нет полностью автоматического фреймворка для сегментации и постановки диагноза. Есть полуавтоматические методы (человек ставит базовый маркер, а потом поправляет ошибки) и методы, где человек — ментор, проверяет работу сегментации и, если надо, правит ошибки. Даже в Human Brain Project до сих пор полную автоматику ввести не смогли. Заключение программы тоже писать не умеют. А сбор классификаторов… берём справочники по цитогии, гистологии и стопку ВОЗовских классификаторов и долго и грустно пишем для каждого типа клеток, каждой архитектуры ткани и патогистологической картины заболевания :) Нужен или программист-медик, или пара из прогера и медика, для верификации. Затем, как вишенка на торте, QA на как минимум двух независимых коллективах врачей. По объёму работ — как минимум хорошая кандидатская.
Главный затык не в том, чтобы написать программу, а чтобы доказать её аналогичность классическим методам и сертифицировать. Диагностика серповидноклеточной анемии по фото мазков сделана много раз за последние 20 лет (у меня ушло 4 часа на мой вариант, это уже стандартная задача для курсовиков), но автоматического анализатора, внедрённую в клиническую практику, до сих пор не существует.

wAngel
05.09.2016 12:27+2Коллеги занимаются диагностированием заболеваний легких на основе снимков DiCom.
Если у кого-то есть интерес, пишите в личку.


JamaGava
Отличный обзор. Позвольте ещё от себя добавить пару интересных задач:
1) Поиск фрагментов изображения по образцу.
К этой задаче может сводиться, например, автоматический поиск шарообразных образований на компьютерной томограмме лёгких. Образования могут быть любого размера, и плотности, что усложняет задачу, но их форма должна быть близкой к шаровидной. Применяется в диагностике рака лёгких и отдельных форм туберкулёза.
2) Поиск скрытых границ.
Текстура изображения компьютерной томографии может быть однородна по яркости (матожидание) и степени «шумности» (дисперсия), а значит однородна для зрительного восприятия человеком. Но при этом, разные участки могут обладать различными значения статистик более высокого порядка (асимметрия, эксцесс). А значит, программно можно построить границу между областями, которая не видна человеку. Применяется в диагностике рака лёгких для поиска границы между опухолью и здоровой но сжатой тканью лёгких (ателектаз).
Примеры, конечно, жуткие… Но когда-то занимался такими задачами, поэтому не смог не поделиться.