На эксклюзивных условиях представляем для вас полный вариант статьи из журнала Хакер, посвященной разработке на R. Под катом вы узнаете, как выжать максимум скорости при работе с табличными данными в языке R.

Примечание: орфография и пунктуация автора сохранены.
К чему лишние слова? Ты же читаешь статью про скорость, поэтому давай сразу к сути! Если у тебя в проекте идет работа с большим объемом данных и на трансформацию таблиц тратится больше времени, чем хотелось, то data.table поможет решить эту проблему. Статья будет интересна тем, кто уже немного знаком с языком R, а также разработчикам, которые его активно используют, но еще не открыли для себя пакет data.table.
Устанавливаем пакеты
Все необходимое для нашей сегодняшней статьи можно проинсталлить с помощью соответствующих функций:
install.packages("data.table")
install.packages("dplyr")
install.packages("readr")R атакует
В последние годы язык R заслуженно набирает популярность в среде машинного обучения. Как правило, для работы с этим подразделом искусственного интеллекта необходимо загрузить данные из нескольких источников, провести с ними преобразования для получения обучающей выборки, на ее основе создать модель, а затем использовать эту модель для предсказаний.
На словах все просто, но в реальной жизни для формирования «хорошей» и устойчивой модели требуется множество попыток, большинство из которых могут быть абсолютно тупиковыми. Язык R помогает упростить процесс создания такой модели, так как это эффективный инструмент анализа табличных данных. Для работы с ними в R существует встроенный тип данных data.frame и огромное количество алгоритмов и моделей, которые его активно используют. К тому же вся мощь R заключается в возможности расширять базовую функциональность с помощью сторонних пакетов. В момент написания материала их количество в официальном репозитории достигло 8914.
Но, как говорится, нет предела совершенству. Большое количество пакетов позволяют облегчить работу с самим типом данных data.frame. Обычно их цель — упростить синтаксис для выполнения наиболее распространенных задач. Здесь нельзя не вспомнить пакет dplyr, который уже стал стандартом де-факто для работы с data.frame, так как за счет него читаемость и удобство работы с таблицами выросли в разы.
Перейдем от теории к практике и создадим data.frame DF со столбцами a, b и с.
DF <- data.frame(a=sample(1:10, 100, replace = TRUE), # Случайные числа от 1 до 10
b=sample(1:5, 100, replace = TRUE), # Случайные числа от 1 до 5
c=100:1) # Числа от 100 до 1Если мы хотим:
- выбрать только столбцы
aис, - отфильтровать строчки, где
a= 2 ис> 10, - создать новую колонку
aс, равную суммеaис, - записать результат в переменную
DF2,
базовый синтаксис на чистом data.frame будет такой:
DF2 <- DF[DF$a == 2 & DF$c > 10, c("a", "c")] # Фильтруем строки и столбцы
DF2$ac <- DF2$a + DF2$c # Создаем новую колонкуС помощью dplyr все гораздо нагляднее:
library(dplyr) # Загрузим пакет dplyr
DF2 <- DF %>% select(a, c) %>% filter(a == 2, c > 10) %>% mutate(ac = a + c) Эти же шаги, но с комментариями:
DF2 <- # результат всего, что справа записать в DF2
DF %>% # взять DF и передать дальше (%>%)
select(a, c) %>% # выбрать колонки «a» и «с» и передать дальше (%>%)
filter(a == 2, c > 10) %>% # отфильтровать строки и передать дальше (%>%)
mutate(ac = a + c) # добавить колонку «ac», равную сумме «а» и «с»Есть и альтернативный подход для работы с таблицами — data.table. Формально data.table — это тоже data.frame, и его можно использовать с существующими функциями и пакетами, которые зачастую ничего не знают о data.table и работают исключительно с data.frame. Этот «улучшенный» data.frame может выполнять многие типовые задачи в несколько раз быстрее своего прародителя. Возникает законный вопрос: где подвох? Этой самой «засадой» в data.table оказывается его синтаксис, который сильно отличается от оригинального. При этом если dplyr с первых же секунд использования делает код легче для понимания, то data.table превращает код в черную магию, и только годы изучения колдовских книг несколько дней практики с data.tableпозволят полностью понять идею нового синтаксиса и принцип упрощения кода.
Пробуем data.table
Для работы с data.table необходимо подключить его пакет.
library(data.table) # Подключение пакетаВ дальнейших примерах эти вызовы будут опущены и будет считаться, что пакет уже загружен.
Так как данные очень часто загружаются из файлов CSV, то уже на этом этапе data.table может удивлять. Для того чтобы показать более измеримые оценки, возьмем какой-нибудь достаточно большой файл CSV. В качестве примера можно привести данные с одного из последних соревнований на Kaggle. Там ты найдешь тренировочный файл CSV размером в 1,27 Гбайт. Структура файла очень простая:
row_id— идентификатор события;x,y— координаты;accuracy— точность;time— время;place_id— идентификатор организации.
Попробуем воспользоваться базовой функцией R — read.csv и измерим время, которое понадобится для загрузки этого файла (для этого обратимся к функции system.time):
system.time(
train_DF <- read.csv("train.csv")
)Время выполнения — 461,349 секунды. Достаточно, чтобы сходить за кофе… Даже если в будущем ты не захочешь пользоваться data.table, все равно старайся реже применять встроенные функции чтения CSV. Есть хорошая библиотека readr, где все реализовано гораздо эффективнее, чем в базовых функциях. Посмотрим ее работу на примере и подключим пакет.
library(readr) Дальше воспользуемся функцией загрузки данных из CSV:
system.time(
train_DF <- read_csv("train.csv")
)Время выполнения — 38,067 секунды — значительно быстрее предыдущего результата! Посмотрим, на что способен data.table:
system.time(
train_DT <- fread("train.csv")
)Время выполнения — 20,906 секунды, что почти в два раза быстрее, чем в readr, и в двадцать раз быстрее, чем в базовом методе.
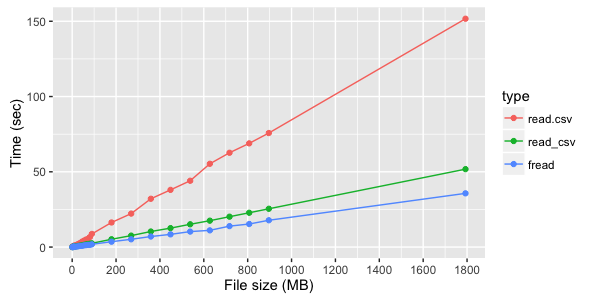
В нашем примере разница в скорости загрузки для разных методов получилась достаточно большая. Внутри каждого из используемых методов время линейно зависит от объема файла, но разница в скорости между этими методами сильно зависит от структуры файла (количества и типов столбцов). Ниже указаны тестовые замеры времени загрузки файлов.
Для файла из трех текстовых колонок видно явное преимущество fread:
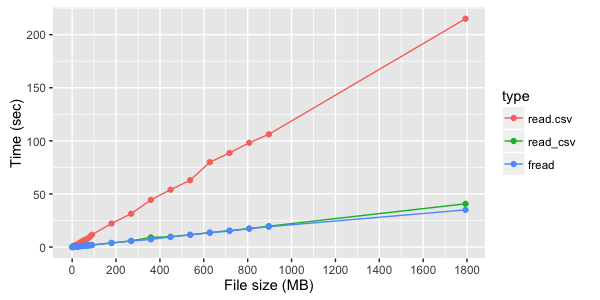
Если же считываются не текстовые, а цифровые колонки, то разница между fread и read_csv менее заметна:
Если после загрузки данных из файла ты собираешься дальше работать с data.table, то fread сразу его возвращает. При других способах загрузки данных будет необходимо сделать data.table из data.frame, хотя это просто:
train_DF # Загруженный data.frame
train_DT <- data.table(train_DF) # data.table созданный из `data.frame`Большинство оптимизаций по скорости в data.table достигается за счет работы с объектами по ссылке, дополнительные копии объектов в памяти не создаются, а значит, экономится время и ресурсы.
Например, ту же задачу создать data.table из data.frame можно было бы решить одной командой на «прокачку», но надо помнить, что первоначальное значение переменной будет потеряно.
train_DF # Загруженный data.frame
setDT(train_DF) # Теперь в переменной DF у нас уже содержится data.tableИтак, данные мы загрузили, пора с ними поработать. Будем считать, что в переменной DT уже есть загруженный data.table. Авторы пакета используют следующее обозначение основных блоков DT[i, j, by]:
- i — фильтр строк;
- j — выбор колонок или выполнение выражения над содержимым
DT; - by — блок для группировки данных.
Вспомним самый первый пример, где мы использовали data.frame DF, и на нем протестируем различные блоки. Начнем с создания data.table из data.frame:
DT <- data.table(DF) # Сделаем экземпляр data.table из существующего data.frameБлок i — фильтр строк
Это самый понятный из блоков. Он служит для фильтра строк data.table, и, если больше ничего дополнительно не требуется, остальные блоки можно не указывать.
DT[a == 2] # Фильтр строк где a == 2
DT[a == 2 & c > 10] # Фильтр строк где a == 2 и c > 10Блок j — выбор колонок или выполнение выражения над содержимым data.table
В данном блоке выполняется обработка содержимого data.table с отфильтрованными строками. Ты можешь просто попросить вернуть нужные столбцы, указав их в списке list. Для удобства введен синоним list в виде точки (то есть list (a, b) эквивалентно .(a, b)). Все существующие в data.table столбцы доступны как «переменные» — тебе не надо работать с ними как со строками, и можно пользоваться intellisense.
DT[, list(a, c)] # Возвращает столбцы «а» и «с» для всех строк
DT[, .(a, c)] # Аналогично предыдущемуМожно также указать дополнительные колонки, которые хочешь создать, и присвоить им необходимые значения:
DT[, .(a, c, ac = a+c)] Если все это объединить, можно выполнить первую задачу, которую мы пробовали решать разными способами:
DT2 <- DT[a == 2 & c > 10, .(a, c, ac = a + c),]Выбор колонок — всего лишь часть возможностей блока j. Также там можно менять существующий data.table. Например, если мы хотим добавить новую колонку в существующем data.table, а не в новой копии (как в прошлом примере), это можно сделать с помощью специального синтаксиса :=.
DT2[, ac_mult2 := ac * 2] # Создание внутри DT2 нового столбца ac_mult2 = ac * 2С помощью этого же оператора можно удалять колонки, присваивая им значение NULL.
DT2[, ac_mult2 := NULL] # Удалим внутри DT2 столбец ac_mult2 Работа с ресурсами по ссылке здорово экономит мощности, и она гораздо быстрее, так как мы избегаем создания копии одних и тех же таблиц с разными колонками. Но надо понимать, что изменение по ссылке меняет сам объект. Если тебе нужна копия этих данных в другой переменной, то надо явно указать, что это отдельная копия, а не ссылка на тот же объект.
Рассмотрим пример:
DT3 <- DT2
DT3[, ac_mult2 := ac * 2] # Создаем новую колонкуМожет показаться, что мы поменяли только DT3, но DT2 и DT3 — это один объект, и, обратившись к DT2, мы увидим там новую колонку. Это касается не только удаления и создания столбцов, так как data.table использует ссылки в том числе и для сортировки. Так что вызов setorder(DT3, "a") повлияет и на DT2.
Для создания копии можно воспользоваться функцией:
DT3 <- copy(DT2)
DT3[, ac_mult2 := NULL] Теперь DT2 и DT3 — это разные объекты, и мы удалили столбец именно у DT3.
by — блок для группировки данных
Этот блок группирует данные наподобие group_by из пакета dplyr или GROUP BY в языке запросов SQL. Логика обращения к data.table с группировкой следующая:
- Блок i фильтрует строки из полного
data.table. - Блок by группирует данные, отфильтрованные в блоке i, по требуемым полям.
- Для каждой группы выполняется блок j, который может либо выбирать, либо обновлять данные.
Блок заполняется следующим способом: by=list(переменные для группировки), но, как и в блоке j, list может быть заменен на точку, то есть by=list(a, b) эквивалентно by=.(a, b). Если необходимо группировать только по одному полю, можно опустить использование списка и написать напрямую by=a:
DT[,.(max = max(c)), by=.(a,b)]
# Для каждого значения «a» и «b» вывести в столбце «max» максимальный «с»
DT[,.(max = max(c)), by=a]
# Для каждого значения «a» вывести в столбце «max» максимальный «с»Самая частая ошибка тех, кто учится работать с data.table, — это применение привычных по data.frame конструкций к data.table. Это очень больное место, и на поиск ошибки можно потратить очень много времени. Если у нас в переменных DF2 (data.frame) и DT2 (data.table) находятся абсолютно одинаковые данные, то указанные вызовы вернут абсолютно разные значения:
DF2[1:5,1:2]
## a c
## 1 2 95
## 2 2 94
## 3 2 92
## 4 2 80
## 5 2 65
DT2[1:5,1:2]
## [1] 1 2Причина этого очень проста:
- логика
data.frameследующая —DF2[1:5,1:2]означает, что надо взять первые пять строк и вернуть для них значения первых двух колонок; - логика
data.tableотличается —DT2[1:5,1:2]означает, что надо взять первые пять строк и передать их в блок j. Блок j просто вернет1и2.
Если надо обратиться к data.table в формате data.frame, необходимо явно указать это с помощью дополнительного параметра:
DT2[1:5,1:2, with = FALSE]
## a c
## 1: 2 95
## 2: 2 94
## 3: 2 92
## 4: 2 80
## 5: 2 65Скорость выполнения
Давай убедимся, что изучение этого синтаксиса имеет смысл. Вернемся к примеру с большим файлом CSV. В train_DF загружен data.frame, а в train_DT, соответственно, data.table.
В используемом примере place_id является целым числом большой длины (integer64), но об этом «догадался» только fread. Остальные методы загрузили это поле как число с плавающей запятой, и нам надо будет явно провести преобразование поля place_id внутри train_DF, чтобы сравнить скорости.
install.packages("bit64") # Пакет для поддержки типа integer64
library(bit64)
train_DF$place_id <- as.integer64(train_DF$place_id)Допустим, перед нами поставлена задача посчитать количество упоминаний каждого place_id в данных.
В dplyr с обычным data.frame это заняло 13,751 секунды:
count <-
train_DF %>% # Выбираем содержимое train_DF
group_by(place_id) %>% # Группируем по place_id
summarise(length(place_id)) # Считаем количество элементов в группе При этом data.table делает то же самое за 2,578 секунды:
system.time(
count2 <- train_DT[,.(.N), by = place_id]
# .N - встроенная функция, показывает количество элементов в группе
)Усложним задачу — для всех place_id посчитаем количество, медиану по x и y, а затем отсортируем по количеству в обратном порядке. data.frame c dplyr справляются с этим за 27,386 секунды:
system.time(
count <-
train_DF %>% # Выбираем train_DF
group_by(place_id) %>% # Группируем по place_id
summarise(count = length(place_id), # Считаем количество элементов в группе
mx = median(x), # Считаем медиану x в группе
my = median(y)) %>% # Считаем медиану y в группе
arrange(-count) # Сортируем в обратную сторону по count
)data.table же справился намного быстрее — 12,414 секунды:
system.time(
count2 <- train_DT[,.(count=.N,
mx = median(x), my = median(y)),
by = place_id][order(-count)]
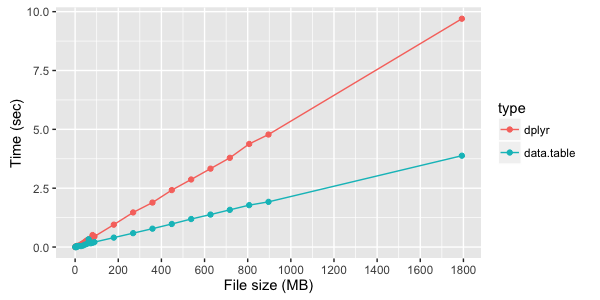
)Тестовые замеры времени выполнения простой группировки данных с помощью dplyr и data.table:
Вместо выводов
Это все лишь поверхностное описание функциональности data.table, но его достаточно, чтобы начать пользоваться этим пакетом. Сейчас развивается пакет dtplyr, который позиционируется как реализация dplyr для data.table, но пока он еще очень молод (версия 0.0.1). В любом случае понимание особенностей работы data.table необходимо до того, чтобы пользоваться дополнительными «обертками».
Об авторе
Станислав Чистяков — эксперт по облачным технологиям и машинному обучению.
WWW от автора
Очень советую почитать статьи, входящие в состав пакета:
WWW от журнала Хакер
Тема языка R не впервые поднимается в нашем журнале. Подкинем тебе еще пару линков на статьи по теме:


ikashnitsky
Для сравнения скорости исполнения операций в
Rстандартом давно стал пакетmicrobenchmark. Суть в том, что время исполнения никогда не бывает строго одинаковым для итераций одной и той же команды. Поэтому для корректного сравнения надо запускать команду много раз подряд (стандарное число итераций — 100) и потом сравнивать распределния потраченного времени. Чаще всего сравнивают медианы. Пример: