 Привет, Хаброжители! «Предиктивное моделирование на практике» охватывает все аспекты прогнозирования, начиная с ключевых этапов предварительной обработки данных, разбиения данных и основных принципов настройки модели. Все этапы моделирования рассматриваются на практических примерах из реальной жизни, в каждой главе дается подробный код на языке R.
Привет, Хаброжители! «Предиктивное моделирование на практике» охватывает все аспекты прогнозирования, начиная с ключевых этапов предварительной обработки данных, разбиения данных и основных принципов настройки модели. Все этапы моделирования рассматриваются на практических примерах из реальной жизни, в каждой главе дается подробный код на языке R.Эта книга может использоваться как введение в предиктивные модели и руководство по их применению. Читатели, не обладающие математической подготовкой, оценят интуитивно понятные объяснения конкретных методов, а внимание, уделяемое решению актуальных задач с реальными данными, поможет специалистам, желающим повысить свою квалификацию.
Авторы постарались избежать сложных формул, для усвоения основного материала достаточно понимания основных статистических концепций, таких как корреляция и линейный регрессионный анализ, но для изучения углубленных тем понадобится математическая подготовка.
Отрывок. 7.5. Вычисления
В этом разделе будут использоваться функции из пакетов R caret, earth, kernlab и nnet.
В R имеется немало пакетов и функций для создания нейросетей. К их числу относятся и пакеты nnet, neural и RSNNS. Основное внимание уделяется пакету nnet, поддерживающему базовые модели нейросетей с одним уровнем скрытых переменных, снижение весов, и характеризующемуся сравнительно простым синтаксисом. RSNNS поддерживает широкий спектр нейросетей. Отметим, что у Бергмейра и Бенитеса (Bergmeir and Benitez, 2012) есть краткое описание различных пакетов нейросетей в R. Там же приводится учебное руководство по RSNNS.
Нейросети
Для апрроксимации регрессионной модели функция nnet может получать как формулу модели, так и матричный интерфейс. Для регрессии линейная связь между скрытыми переменными и прогнозом используется с параметром linout = TRUE. Простейший вызов функции нейросети будет иметь вид:
> nnetFit <- nnet(predictors, outcome,
+ size = 5,
+ decay = 0.01,
+ linout = TRUE,
+ ## Сокращенный вывод
+ trace = FALSE,
+ ## Расширение количества итераций для нахождения
+ ## оценок параметров..
+ maxit = 500,
+ ## и количества параметров, используемых моделью
+ MaxNWts = 5 * (ncol(predictors) + 1) + 5 + 1)Этот вызов создает одну модель с пятью скрытыми переменными. Предполагается, что данные в предикторах были стандартизированы по одной шкале.
Для усреднения моделей используется функция avNNet из пакета caret, имеющая тот же синтаксис:
> nnetAvg <- avNNet(predictors, outcome,
+ size = 5,
+ decay = 0.01,
+ ## Количество усредняемых моделей
+ repeats = 5,
+ linout = TRUE,
+ ## Сокращенный вывод
+ trace = FALSE,
+ ## Расширение количества итераций
+ ## для нахождения оценок параметров..
+ maxit = 500,
+ ## и количества параметров, используемых моделью
+ MaxNWts = 5 * (ncol(predictors) + 1) + 5 + 1)Новые точки данных обрабатываются командой
> predict(nnetFit, newData)
> ## или
> predict(nnetAvg, newData)Чтобы воспроизвести представленный ранее метод выбора количества скрытых переменных и величины снижения весов посредством повторной выборки, применим функцию train с параметром method = «nnet» или method = «avNNet», сначала удалив предикторы (с тем, чтобы максимальная абсолютная парная корреляция между предикторами не превышала 0,75):
> ## findCorrelation получает матрицу корреляции и определяет
> ## номера столбцов, которые должны быть удалены для поддержания
> ## всех парных корреляций ниже заданного порога
> tooHigh <- findCorrelation(cor(solTrainXtrans), cutoff = .75)
> trainXnnet <- solTrainXtrans[, -tooHigh]
> testXnnet <- solTestXtrans[, -tooHigh]
> ## Создание набора моделей-кандидатов для оценки:
> nnetGrid <- expand.grid(.decay = c(0, 0.01, .1),
+ .size = c(1:10),
+ ## Другой вариант — использование бэггинга
+ ## (см. следующую главу) вместо случайной
+ ## инициализации генератора.
+ .bag = FALSE)
> set.seed(100)
> nnetTune <- train(solTrainXtrans, solTrainY,
+ method = "avNNet",
+ tuneGrid = nnetGrid,
+ trControl = ctrl,
+ ## Автоматическая стандартизация данных
+ ## перед моделированием и прогнозированием
+ preProc = c("center", "scale"),
+ linout = TRUE,
+ trace = FALSE,
+ MaxNWts = 10 * (ncol(trainXnnet) + 1) + 10 + 1,
+ maxit = 500)Многомерные адаптивные регрессионные сплайны
Модели MARS содержатся в нескольких пакетах, но самая обширная реализация находится в пакете earth. Модель MARS, использующая номинальную фазу прямого прохода и усечения, может вызываться следующим образом:
> marsFit <- earth(solTrainXtrans, solTrainY)
> marsFit
Selected 38 of 47 terms, and 30 of 228 predictors
Importance: NumNonHAtoms, MolWeight, SurfaceArea2, SurfaceArea1, FP142, ...
Number of terms at each degree of interaction: 1 37 (additive model)
GCV 0.3877448 RSS 312.877 GRSq 0.907529 RSq 0.9213739Поскольку эта модель во внутренней реализации использует метод GCV для выбора модели, ее устройство несколько отличается от модели, описанной ранее в этой главе. Метод summary генерирует более обширный вывод:
> summary(marsFit)
Call: earth(x=solTrainXtrans, y=solTrainY)
coefficients
(Intercept) -3.223749
FP002 0.517848
FP003 -0.228759
FP059 -0.582140
FP065 -0.273844
FP075 0.285520
FP083 -0.629746
FP085 -0.235622
FP099 0.325018
FP111 -0.403920
FP135 0.394901
FP142 0.407264
FP154 -0.620757
FP172 -0.514016
FP176 0.308482
FP188 0.425123
FP202 0.302688
FP204 -0.311739
FP207 0.457080
h(MolWeight-5.77508) -1.801853
h(5.94516-MolWeight) 0.813322
h(NumNonHAtoms-2.99573) -3.247622
h(2.99573-NumNonHAtoms) 2.520305
h(2.57858-NumNonHBonds) -0.564690
h(NumMultBonds-1.85275) -0.370480
h(NumRotBonds-2.19722) -2.753687
h(2.19722-NumRotBonds) 0.123978
h(NumAromaticBonds-2.48491) -1.453716
h(NumNitrogen-0.584815) 8.239716
h(0.584815-NumNitrogen) -1.542868
h(NumOxygen-1.38629) 3.304643
h(1.38629-NumOxygen) -0.620413
h(NumChlorine-0.46875) -50.431489
h(HydrophilicFactor- -0.816625) 0.237565
h(-0.816625-HydrophilicFactor) -0.370998
h(SurfaceArea1-1.9554) 0.149166
h(SurfaceArea2-4.66178) -0.169960
h(4.66178-SurfaceArea2) -0.157970
Selected 38 of 47 terms, and 30 of 228 predictors
Importance: NumNonHAtoms, MolWeight, SurfaceArea2, SurfaceArea1, FP142, ...
Number of terms at each degree of interaction: 1 37 (additive model)
GCV 0.3877448 RSS 312.877 GRSq 0.907529 RSq 0.9213739В этом выводе h(·) — шарнирная функция. В приведенных результатах составляющая h(MolWeight-5.77508) равна нулю при значении молекулярной массы меньше 5,77508 (как в верхней части рис. 7.3). Отраженная шарнирная функция имеет вид h(5.77508 — MolWeight).
Функция plotmo из пакета earth может использоваться для построения диаграмм, сходных с изображенными на рис. 7.5. Для настройки модели с использованием внешней повторной выборки можно воспользоваться train. В следующем коде воспроизведены результаты, отображенные на рис. 7.4:
> # Определение моделей-кандидатов для тестирования
> marsGrid <- expand.grid(.degree = 1:2, .nprune = 2:38)
> # Инициализация генератора для воспроизведения результатов
> set.seed(100)
> marsTuned <- train(solTrainXtrans, solTrainY,
+ method = "earth",
+ # Явное объявление моделей-кандидатов для тестирования
+ tuneGrid = marsGrid,
+ trControl = trainControl(method = "cv"))
> marsTuned
951 samples
228 predictors
No pre-processing
Resampling: Cross-Validation (10-fold)
Summary of sample sizes: 856, 857, 855, 856, 856, 855, ...
Resampling results across tuning parameters:
degree nprune RMSE Rsquared RMSE SD Rsquared SD
1 2 1.54 0.438 0.128 0.0802
1 3 1.12 0.7 0.0968 0.0647
1 4 1.06 0.73 0.0849 0.0594
1 5 1.02 0.75 0.102 0.0551
1 6 0.984 0.768 0.0733 0.042
1 7 0.919 0.796 0.0657 0.0432
1 8 0.862 0.821 0.0418 0.0237
: : : : : :
2 33 0.701 0.883 0.068 0.0307
2 34 0.702 0.883 0.0699 0.0307
2 35 0.696 0.885 0.0746 0.0315
2 36 0.687 0.887 0.0604 0.0281
2 37 0.696 0.885 0.0689 0.0291
2 38 0.686 0.887 0.0626 0.029
RMSE was used to select the optimal model using the smallest value.
The final values used for the model were degree = 1 and nprune = 38.
> head(predict(marsTuned, solTestXtrans))
[1] 0.3677522 -0.1503220 -0.5051844 0.5398116 -0.4792718 0.7377222Для оценки важности каждого предиктора в модели MARS используются две функции: evimp из пакета earth и varImp из пакета caret (при этом вторая вызывает первую):
> varImp(marsTuned)
earth variable importance
only 20 most important variables shown (out of 228)
Overall
MolWeight 100.00
NumNonHAtoms 89.96
SurfaceArea2 89.51
SurfaceArea1 57.34
FP142 44.31
FP002 39.23
NumMultBond s 39.23
FP204 37.10
FP172 34.96
NumOxygen 30.70
NumNitrogen 29.12
FP083 28.21
NumNonHBonds 26.58
FP059 24.76
FP135 23.51
FP154 21.20
FP207 19.05
FP202 17.92
NumRotBonds 16.94
FP085 16.02Эти результаты масштабируются в интервале от 0 до 100, отличаясь от приведенных в табл. 7.1 (представленная в табл. 7.1 модель не прошла полный процесс роста и усечения). Отметим, что переменные, следующие за несколькими первыми из них, менее значимы для модели.
SVM, метод опорных векторов
Реализации моделей SVM содержатся в нескольких пакетах R. У Чанга и Лина (Chang and Lin, 2011) функция svm из пакета e1071 использует интерфейс к библиотеке LIBSVM для регрессии. Более полная реализация моделей SVM для регрессии по Карацоглу (Karatzoglou et al., 2004) содержится в пакете kernlab, включающем и функцию ksvm для регрессионных моделей и большого количества ядерных функций. По умолчанию используется радиальная базисная функция. Если значения стоимости и ядерных параметров известны, то аппроксимация модели может быть выполнена следующим образом:
> svmFit <- ksvm(x = solTrainXtrans, y = solTrainY,
+ kernel ="rbfdot", kpar = "automatic",
+ C = 1, epsilon = 0.1)Для оценки ? применен автоматизированный анализ. Так как y является числовым вектором, функция заведомо аппроксимирует регрессионную модель (вместо классификационной модели). Можно использовать и другие ядерные функции, включая полиномиальные (kernel = «polydot») и линейные (kernel = «vanilladot»).
Если значения неизвестны, то их можно оценить посредством повторной выборки. В train значения «svmRadial», «svmLinear» или «svmPoly» параметра method выбирают разные ядерные функции:
> svmRTuned <- train(solTrainXtrans, solTrainY,
+ method = "svmRadial",
+ preProc = c("center", "scale"),
+ tuneLength = 14,
+ trControl = trainControl(method = "cv"))Аргумент tuneLength использует по умолчанию 14 значений стоимости

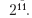 Оценка ? по умолчанию выполняется посредством автоматического анализа.
Оценка ? по умолчанию выполняется посредством автоматического анализа.> svmRTuned
951 samples
228 predictors
Pre-processing: centered, scaled
Resampling: Cross-Validation (10-fold)
Summary of sample sizes: 855, 858, 856, 855, 855, 856, ...
Resampling results across tuning parameters:
C RMSE Rsquared RMSE SD Rsquared SD
0.25 0.793 0.87 0.105 0.0396
0.5 0.708 0.889 0.0936 0.0345
1 0.664 0.898 0.0834 0.0306
2 0.642 0.903 0.0725 0.0277
4 0.629 0.906 0.067 0.0253
8 0.621 0.908 0.0634 0.0238
16 0.617 0.909 0.0602 0.0232
32 0.613 0.91 0.06 0.0234
64 0.611 0.911 0.0586 0.0231
128 0.609 0.911 0.0561 0.0223
256 0.609 0.911 0.056 0.0224
512 0.61 0.911 0.0563 0.0226
1020 0.613 0.91 0.0563 0.023
2050 0.618 0.909 0.0541 0.023
Tuning parameter 'sigma' was held constant at a value of 0.00387
RMSE was used to select the optimal model using the smallest value.
The final values used for the model were C = 256 and sigma = 0.00387.Подобъект finalModel содержит модель, созданную функцией ksvm:
> svmRTuned$finalModel
Support Vector Machine object of class "ksvm"
SV type: eps-svr (regression)
parameter : epsilon = 0.1 cost C = 256
Gaussian Radial Basis kernel function.
Hyperparameter : sigma = 0.00387037424967707
Number of Support Vectors : 625
Objective Function Value : -1020.558
Training error : 0.009163В качестве опорных векторов модель использует 625 точек данных тренировочного набора (то есть 66 % тренировочного набора).
Пакет kernlab содержит реализацию модели RVM для регрессии в функции rvm. Ее синтаксис очень похож на представленный в примере для ksvm.
Метод KNN
Функция knnreg из пакета caret аппроксимирует регрессионную модель KNN; функция train настраивает модель по K:
> # Сначала удаляются разреженные и несбалансированные предикторы.
> knnDescr <- solTrainXtrans[, -nearZeroVar(solTrainXtrans)]
> set.seed(100)
> knnTune <- train(knnDescr,
+ solTrainY,
+ method = "knn",
+ # Центрирование и масштабирование будет выполняться
+ # для новых прогнозов
+ preProc = c("center", "scale"),
+ tuneGrid = data.frame(.k = 1:20),
+ trControl = trainControl(method = "cv"))Об авторах:
Макс Кун — руководитель отдела статистических неклинических исследований и разработки Pfizer Global. Он работает с предиктивными моделями более 15 лет и является автором нескольких специализированных пакетов для языка R. Предиктивное моделирование на практике охватывает все аспекты прогнозирования, начиная с ключевых этапов предварительной обработки данных, разбиения данных и основных принципов
настройки модели. Все этапы моделирования рассматриваются на практических примерах
из реальной жизни, в каждой главе дается подробный код на языке R.
Кьелл Джонсон — более десяти лет работает в области статистики и предиктивного моделирования для фармацевтических исследований. Является соучредителем Arbor Analytics – компании, специализирующейся на предиктивном моделировании; ранее возглавлял отдел статистических исследований и разработки в Pfizer Global. Его научная работа посвящена применению и разработке статистической методологии и алгоритмов обучения.
» Более подробно с книгой можно ознакомиться на сайте издательства
» Оглавление
» Отрывок
Для Хаброжителей скидка 25% по купону — Applied Predictive Modeling
По факту оплаты бумажной версии книги на e-mail высылается электронная книга.

