Пару недель назад мы начали рассказывать о проектах, которые стали победителями Школы по практическому программированию и анализу данных НИУ ВШЭ — Санкт-Петербург и компании JetBrains.
Второе место заняла команда одиннадцатиклассников из СУНЦ МГУ. Ребята реализовали модель, которая предсказывает растворимость веществ, основываясь на SMILES представлении молекул. Что это такое, какие методы машинного обучения можно использовать в этой задаче, и согласуются ли полученные результаты с реальными химическими экспериментами, авторы проекта рассказали в этом посте.

Команда
В нашей команде было четыре участника: Андрей Шандыбин, Артём Власов, Владимир Свердлов и Захар Кравчук. Все мы в этом году окончили СУНЦ МГУ и уже имели опыт в машинном обучении на спецкурсе по ML в СУНЦ и на сторонних проектах. Нашим куратором была исследователь лаборатории Machine Learning Applications and Deep Learning JetBrains Research Алиса Аленичева.
Постановка проблемы
Растворимость молекул необходимо знать для расчета растворов в реакциях, а также при перекристаллизации. Но проводить эксперименты для определения растворимости очень затратно. В этом проекте мы предложили решение, позволяющее предсказывать растворимость, основываясь только на SMILES представлении (что это расскажем дальше) и некоторых других свойствах молекул. Подобные модели помогут в создании новых материалов и лекарственных средств.
Если формулировать проблему в терминах машинного обучения, то мы решали задачу регрессии, где целевая переменная — логарифм растворимости моль на литр. Для измерения качества модели мы выбрали метрику Root Mean Square Error (RMSE), которая считается по формуле:
План работы:
Предобработать данные и построить baseline модель;
Применить графовые сверточные нейросети (GCN);
Провести эксперименты;
Визуализировать результаты.
Данные
Мы использовали ESOL датасет, в котором представлены 1128 различных молекул. Для каждой молекулы было приведено десять параметров и описание ее структуры в формате SMILES. Надо заметить, что это достаточно маленький датасет, из-за этого при разных разбиениях на тренировочную и тестовую выборку результаты варьировались.
Разберемся, что такое SMILES. SMILES (Simplified Molecular Input Line Entry System) — система правил однозначного описания состава и структуры молекулы химического вещества с использованием строки символов ASCII. В дальнейшем с помощью open-source библиотеки RDKit мы сможем извлечь много полезных молекулярных свойств только из SMILES представления молекул.
Реализация класса для загрузки и предобработки датасета
class DatasetsHolder:
@staticmethod
def read_datasets(inp_folder_path):
df = pd.read_csv(inp_folder_path)
return df
# return pandas DataFrame
class StandardizeDatasets:
@staticmethod
def standardize_smiles(smi: str) -> Optional[str]:
mol = Chem.MolFromSmiles(smi)
mol = Chem.MolToSmiles(mol)
return mol
"crete typical standardization of one smiles"
@logger.catch()
def standardize(self, inp_path: Path, out_path: Path):
df_reader = DatasetsHolder()
df = df_reader.read_datasets(inp_path)
with Pool(10) as pool:
df['standardize_smiles'] = list(
tqdm(pool.imap(self.standardize_smiles, df.smiles), total=df.shape[0])
)
df.to_csv(out_path, index=False)
return df
"apply standardization to all smiles"
class StandardizeTautomers(StandardizeDatasets):
@staticmethod
def standardize_smiles(smi: str) -> Optional[str]:
Canonicalizer = TautomerCanonicalizer()
mol = Chem.MolFromSmiles(smi)
standorized = Canonicalizer.canonicalize(mol)
return Chem.MolToSmiles(standorized)
# "apply TautomerCanonicalizer() to standardization"Baseline
В качестве baseline-модели мы выбрали градиентный бустинг (XGBoost). Для начала мы обучили модель только на признаках, представленных в датасете, без учета SMILES.
Реализация функций для получений химических свойств молекул
from descriptastorus.descriptors import rdDescriptors
from rdkit import Chem
import logging
from descriptastorus.descriptors import rdNormalizedDescriptors
generator = rdNormalizedDescriptors.RDKit2DNormalized()
def rdkit_2d_features(smiles: str):
features = generator.process(smiles)
if features[0] == False:
print(f'{smiles} were not processed correctly')
return None
else:
return features[1:]
def create_feature_dataframe(df):
feature_names = [x[0] for x in generator.columns]
rdkit_feats = [ ]
for i in range(len(df)):
smiles = df.iloc[i][SMILES_COLUMN]
target_value = df.iloc[i]['measured log solubility in mols per litre']
features = generator.process(smiles)
dictionary = dict(zip(feature_names, features[1:]))
dictionary['target'] = target_value
rdkit_feats.append(dictionary)
return pd.DataFrame(rdkit_feats)Таким образом бейзлайн основывается только на отдельных свойствах молекул, в то время как следующий наш подход, графовые сверточные нейросети, учитывают еще и то, как атомы молекул соотносятся между собой (этот метод будет описан дальше).
Реализация обучения бейзлайна
from xgboost import XGBRegressor
X_train = train_data.drop(columns=['target'])
y_train = train_data['target']
X_test = test_data.drop(columns=['target'])
y_test = test_data['target']
model = XGBRegressor()
model.fit(X_train, y_train)Представление молекул в виде графа
Рассмотрим, как молекула представляется в виде графа. В SMILES содержится описание структуры молекулы. Из курса химии мы знаем, что молекулы можно представить в виде графа, где вершины — это атомы. Молекула состоит из атомов и каждому атому можно сопоставить вектор свойств: атомный номер, количество водорода, степень, заряд, хиральность, гибридизация, ароматичность и масса атома.
Реализация выделения свойств атомов и матрицу смежности с помощью библиотеки RDkit для представления молекул в виде графа
def get_atom_features(mol):
atomic_number = []
num_hs = []
degrees = []
charges = []
tags = []
hybridizations = []
aromatic = []
mass = []
for atom in mol.GetAtoms():
atomic_number.append(atom.GetAtomicNum()) # atomic number
num_hs.append(atom.GetTotalNumHs(includeNeighbors=True)) # number of H in atom
degrees.append(atom.GetTotalDegree()) # total Degree of atom
charges.append(atom.GetFormalCharge()) # Charge of atom
tags.append(int(atom.GetChiralTag())) # chiral tag
hybridizations.append(int(atom.GetHybridization())) # hybridization of atom
if atom.GetIsAromatic(): # Is aromatic of not
aromatic.append(1)
else:
aromatic.append(0)
mass.append(atom.GetMass() * 0.01) # mass
return torch.tensor([atomic_number, num_hs, degrees, charges, tags, hybridizations, aromatic, mass]).t()
def get_edge_index(mol):
row, col = [], []
for bond in mol.GetBonds():
start, end = bond.GetBeginAtomIdx(), bond.GetEndAtomIdx()
row += [start, end]
col += [end, start]
return torch.tensor([row, col], dtype=torch.long)Fingerprints
Что такое фингерпринт? Фингерпринт — вектор чисел, описывающий структурный состав молекулы, а с появлением машинного обучения — вектор абстрактных свойств молекулы, на основе которого можно делать предсказания молекулярных свойств.Дальше мы рассмотрим несколько видов фингерпринтов.
Morgan Fingerprints VS Neural Fingerprints
Моргановский фингерпринт — это универсальный способ представления молекул в виде бинарного вектора, где после конкатенации векторов из свойств атомов применяется хэш-функция.
У этого подхода есть несколько минусов. Во-первых, полученный фингерпринт очень разреженный, из-за чего дальнейшее обучение полносвязной нейросети становится очень затруднительным. Во-вторых, это общий подход, при котором фингерпринты не обучаются, а мы хотим оптимизировать фингерпринт для конкретной задачи. Такой подход называется Neural Fingerprint. Для этого хэширование и конкатенацию заменим на дифференцируемые операции, тогда мы сможем обучать фингерпринт одновременно с полносвязной нейросетью. На картинке ниже схематично изображены способы получения и Моргановских, и обучаемых фингерпринтов.
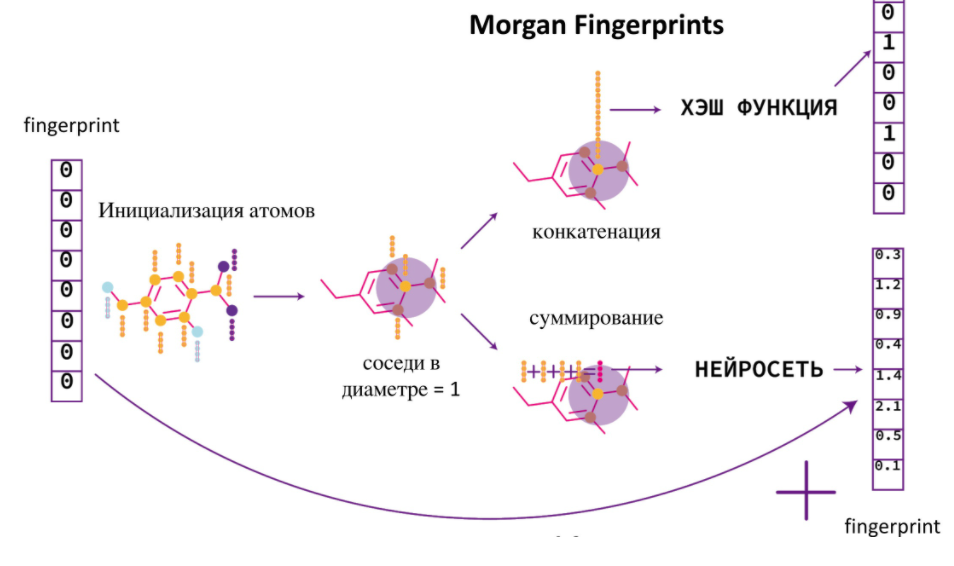
Message passing
Рассмотрим подробнее как обучаются фингерпринты. В основе их обучения лежит алгоритм Message passing.
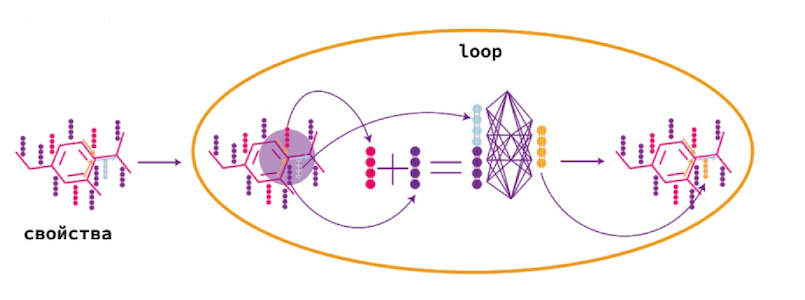
Message passing — это алгоритм обновления векторов, репрезентирующих атомы. Для каждого атома сначала складываются вектора данного атома и вектора его соседей.
После этого полученный вектор подается на вход в полносвязную нейросеть, тогда на выходе получается обновленный вектор, репрезентирующий атомы. И этот алгоритм повторяется для всех атомов молекулы.
Ниже представлены классы реализующие этот алгоритмы с использованием библиотеки PyTorch Geometric.
Message Passing
class GCNConv(MessagePassing):
def __init__(self, in_channels, out_channels):
super(GCNConv, self).__init__(aggr='add') # "Add" aggregation (Step 5).
self.lin = torch.nn.Linear(in_channels, out_channels)
def forward(self, x, edge_index):
# x has shape [N, in_channels]
# edge_index has shape [2, E]
# Step 1: Add self-loops to the adjacency matrix.
edge_index, _ = add_self_loops(edge_index, num_nodes=x.size(0))
# Step 2: Linearly transform node feature matrix.
x = self.lin(x)
# Step 3: Compute normalization.
row, col = edge_index
deg = degree(col, x.size(0), dtype=x.dtype)
deg_inv_sqrt = deg.pow(-0.5)
norm = deg_inv_sqrt[row] * deg_inv_sqrt[col]
# Step 4-5: Start propagating messages.
return self.propagate(edge_index, x=x, norm=norm)
def message(self, x_j, norm):
# x_j has shape [E, out_channels]
# Step 4: Normalize node features.
return norm.view(-1, 1) * x_jNeural loop
class NeuralFP(nn.Module):
def __init__(self, atom_features=52, fp_size=50):
super(NeuralFP, self).__init__()
self.atom_features = atom_features
self.fp_size = fp_size
self.loop1 = GCNConv(atom_features, fp_size)
self.loops = nn.ModuleList([self.loop1])
def forward(self, data):
fingerprint = torch.zeros((data.batch.shape[0], self.fp_size), dtype=torch.float).to(device)
out = data.x
for idx, loop in enumerate(self.loops):
updated_fingerprint = loop(out, data.edge_index)
fingerprint += updated_fingerprint
return scatter_add(fingerprint, data.batch, dim=0)Итоговая архитектура — GCN (Graph Convolutional Network)
После обновления векторов атомов мы также можем их сложить, после чего подать на вход в полносвязной нейросети. Тогда на выходе мы получим фингерпринт.
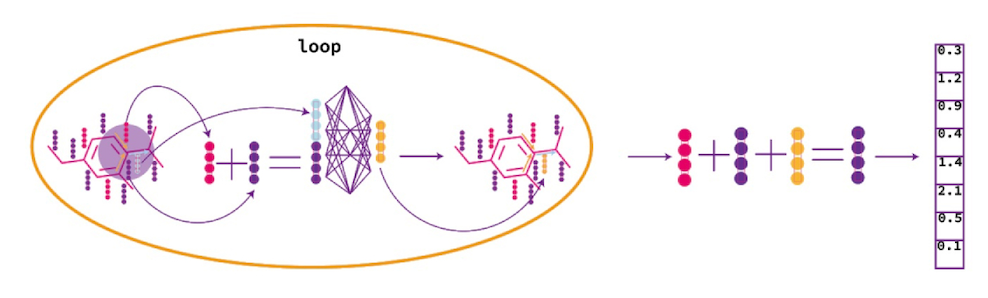
После полученный фингерпринт мы подадим на вход другой полносвязной нейросети, которая уже и будет делать финальное предсказание растворимости.
Код
import torch.nn.functional as F
class MLP_Regressor(nn.Module):
def __init__(self, neural_fp, atom_features=2, fp_size=50, hidden_size=100):
super(MLP_Regressor, self).__init__()
self.neural_fp = neural_fp
self.lin1 = nn.Linear(fp_size , hidden_size)
self.leakyrelu = nn.LeakyReLU(0.2)
self.lin2 = nn.Linear(hidden_size, 1)
self.dropout = nn.Dropout(0.2)
def forward(self, batch):
fp = self.neural_fp(batch)
hidden = self.dropout(self.lin1(fp))
out = self.leakyrelu(self.lin2(hidden))
return outЭксперименты
Рассмотрим эксперименты, поставленные в процессе работы.
После того, как мы обучили GCN только на SMILES, мы начали добавлять дополнительные фичи. То есть после получения фингерпринта к нему конкатенировали различные данные, после чего делали финальные предсказания. Сначала мы добавили 200 дополнительных параметров, которые были получены при построении бейзлайна. Затем попробовали к обучаемым фингерпринтам добавить Моргановские фингерпринты.
Реализация класса MLP regressor с добавлением различный свойств
class MLP_Regressor(nn.Module):
def __init__(self, neural_fp, atom_features=2, fp_size=100, hidden_size=300, num_additional_features = 207):
super(MLP_Regressor, self).__init__()
self.neural_fp = neural_fp
self.lin1 = nn.Linear(fp_size+num_additional_features, hidden_size)
self.leakyrelu = nn.LeakyReLU(0.2)
self.lin2 = nn.Linear(hidden_size, 1)
self.dropout = nn.Dropout(0.2)
def forward(self, batch, additional_features):
fp = self.neural_fp(batch)
fp = torch.cat((fp, additional_features), dim=1)
hidden = self.dropout(self.lin1(fp))
out = self.leakyrelu(self.lin2(hidden))
return outТак как мы хотели оптимизировать фингерпринты под нашу конкретную задачу, то подумали, что результаты градиентного бустинга могут улучшится, если добавить оптимизированные фингерпринты.
Результаты
Для измерения результатов моделей использовалось 10% всех данных, а для тренировки — 90%. На этой диаграмме представлены результаты наших экспериментов. По оси абсцисс представлены модели, по оси ординат — RMSE.
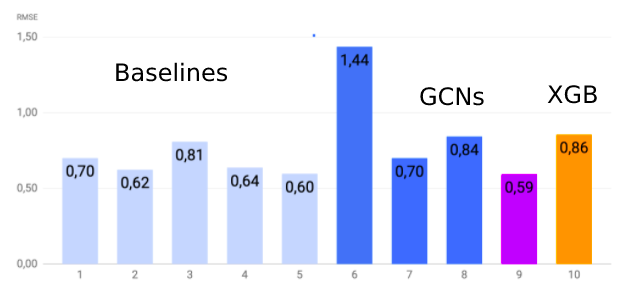
Как мы видим, градиентный бустинг показал достаточно хорошие результаты, но графовые нейросети все-таки оказались лучше. Надо заметить, что так как датасет маленький, результаты могут варьироваться при разных разбиениях на тренировочную и тестовую выборку.
Визуализация модели
В этом разделе мы попытаемся проинтерпретировать полученную нами модель. После обучения модели мы выделяем те подструктуры молекулы, которые сильнее всего активируют биты обученного фингерпринта, коррелирующие с предсказанной растворимостью (оригинальная статья). После этого мы посмотрим, соответствуют ли свойства, выделенные нашей моделью, свойствам, которые влияют на растворимость с точки зрения химии.

Как мы видим, наша модель выделила наличие гидроксильных и ароматических групп как наиболее важные признаки для предсказания растворимости. Это доказывает, что у нас получилась достаточно хорошая модель: из курса химии мы знаем, что гидроксильные группы создают водородные связи с водой, а ароматические группы довольно устойчивы из-за хорошо распределенной электронной плотности, и поэтому часто плохо растворимы. Таким образом, мы смогли проинтерпретировать и еще раз оценить качество полученной нами модели.
Улучшения
Данный проект можно улучшить, использовав не только свойства атомов, но и свойства связей в молекуле. А также алгоритм Directed Message Passing (DMPNN), который является усовершенствованной версией использованного алгоритма Message Passing. Так как на выполнение проекта у нас была всего неделя, мы не использовали алгоритм DMPNN, потому что на его реализацию ушло бы намного больше времени. Но в будущем мы хотим продолжить работать над этим проектом и попробовать подход DMPNN.
Еще одним существенным улучшением станет обучение модели на датасете большего размера. К сожалению, ESOL, который мы использовали, — один из самых больших датасетов, находящихся в открытом доступе. Другие датасеты принадлежат частным компаниям и открыто не публикуются.
Заключение
Нам очень понравилось заниматься этим проектом в рамках школы. Мы получили ценный опыт работы над реальным проектом, который поможет нам в дальнейшем продвигаться в области машинного обучения.
Также хотим выразить благодарность нашему куратору Алисе, которая помогала нам на протяжении всей смены, и без которой данный проект не был бы возможен.
Другие материалы из нашего блога о проектах старшеклассников и студентов первого курса:


andyudol
Растворимость молекул?
Loskechos
Тут важно понимать что они понимают под растворимостью. Способность к диссоциации или способность кристалла распадаться на молекулярные комплексы окруженные сольватной оболочкой.