
Краткий экскурс в генетику. Если вдруг вы помните, в 2003 г. было сделано сенсационное заявление: ученые, наконец, расшифровали геном человека. Геном построен из ДНК, а ДНК – это исходный код организма. ДНК представляет собой двойную цепочку, состоящую из 4-х видов нуклеотидов, которые повторяются в геноме человека порядка 3 млрд. раз. Так же, как в битах зашифрована вся информация на вашем компьютере, в нуклеотидах зашифрована инструкция о сборке всех белков человеческого тела. То есть зная, в какой последовательности расположены нуклеотиды в ДНК, мы теоретически можем собрать все необходимые белки и получить модель человека. Так вот в стандартном понимании ученые не расшифровали ДНК, а просто перевели химическую последовательность в набор нулей и единиц на компьютере. Что делать с этим дальше – отдельный разговор. Например, на данный момент нам ясна функция лишь 5% всего массива генома (это кодирование белков). Чем занимаются остальные 95%, можно только предполагать.
В 2003 году стоимость секвенирования ДНК человека составляла около 100 млн долларов. С течением времени эта цифра уменьшалась и сейчас она приближается к тысяче долларов. Вы платите, вашу ДНК секвенируют и отдают вам жесткий диск с 3 ГБ информации – вашим геномом в цифровом виде.
Сегодня на рынке представлено три основных секвенатора. Самый производительный, Hiseq, и его приемник NovaSeq, обеспечивает самое дешевое (флуоресцентное) секвенирование. Один его запуск длится несколько дней, и за это время обрабатываются геномы сразу нескольких человек. Однако сам запуск стоит около десятка тысяч долларов. К слову, и сам прибор стоит порядка $1 млн, а, поскольку устаревает он примерно за 3 года, для того, чтобы он окупился, он должен приносить вам $1000 в день.
Второй прибор появился на рынке буквально прошлым летом. Он называется Nanopore и базируется на очень интересной технологии, когда ДНК секвенируется путем пропускания через нанопору. Самый дешевый вариант Nanopore позиционируется как одноразовый домашний секвенатор и стоит $1000.
Третий прибор – PGM, полупроводниковый секвенатор, который стоит $50 000 у себя на родине и около 10 млн рублей (с доставкой, растаможиванием и т. д.) в России. Процесс секвенирования на нем занимает порядка нескольких часов.
Что ж, десяти миллионов у меня не было, а PGM захотелось. Пришлось сделать самому. Сначала вкратце о том, как происходит полупроводниковое секвенирование. Вся цепочка ДНК делится на фрагменты длиной по 300-400 нуклеотидов, называемые ридами. Затем риды прикрепляются к маленьким сферам и многократно копируются – в итоге на каждой сфере «висит» целый пучок одинаковых фрагментов ДНК. Копирование нужно для усиления сигнала от каждого конкретного рида. Набор разных сфер называется библиотекой ДНК.
Сердцем PGM является одноразовый чип – матрица, похожая на матрицу в фотокамере, только вместо пикселей, реагирующих на свет, здесь pH-транзисторы, реагирующие на изменение кислотно-щелочного баланса. Полученная библиотека ДНК загружается на чип, содержащий 10 млн лунок, на дне каждой из них находится pH-транзистор. В лунку умещается только одна сфера и, следовательно, риды только одного типа (с одной определенной последовательностью нуклеотидов). Далее на чип подаются реагенты таким образом, чтобы ДНК начала себя копировать. А копируется она линейно, то есть нуклеотиды прикрепляются к вновь создаваемой цепочке в том порядке, в котором они стоят в материнской цепочке. Поэтому на чип подается один тип нуклеотидов – и тут же фиксируется изменение pH в некоторых лунках (это значит, что в них произошло присоединение данного нуклеотида). Далее подается другой тип нуклеотидов и фиксируется изменение pH в лунках и т. д. Таким образом, подавая на чип все 4 типа нуклеотидов много раз, мы можем получить информацию о последовательности нуклеотидов в каждом риде. Затем математическими способами прочитанные короткие отрезки собираются на компьютере в единую цепочку. Чтобы собрать ее более-менее уверенно, каждый рид нужно прочесть примерно по 100 раз.
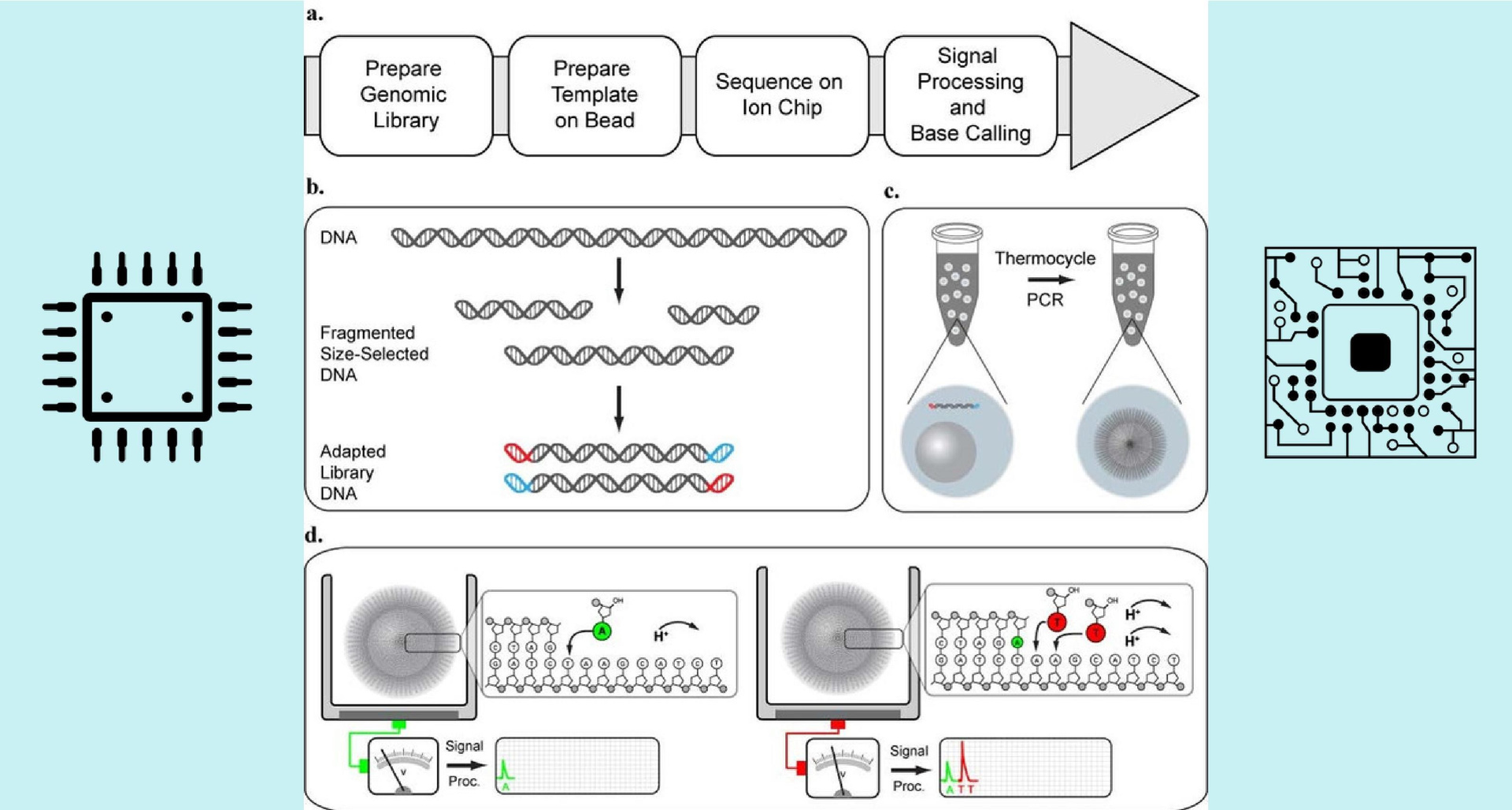
Рис.1. Полупроводниковое секвенирование
Теперь разберемся, из чего состоит сам прибор. Имеется, как мы уже знаем, чип, а также система подачи реагентов и материнская плата. Все секвенирование ведется именно на чипе – остальной аппарат только передает на него определенные сигналы, подает реагенты, считывает с него аналоговые сигналы, оцифровывает их и гонит полученный поток информации на компьютер, где данные накапливаются и обрабатываются.
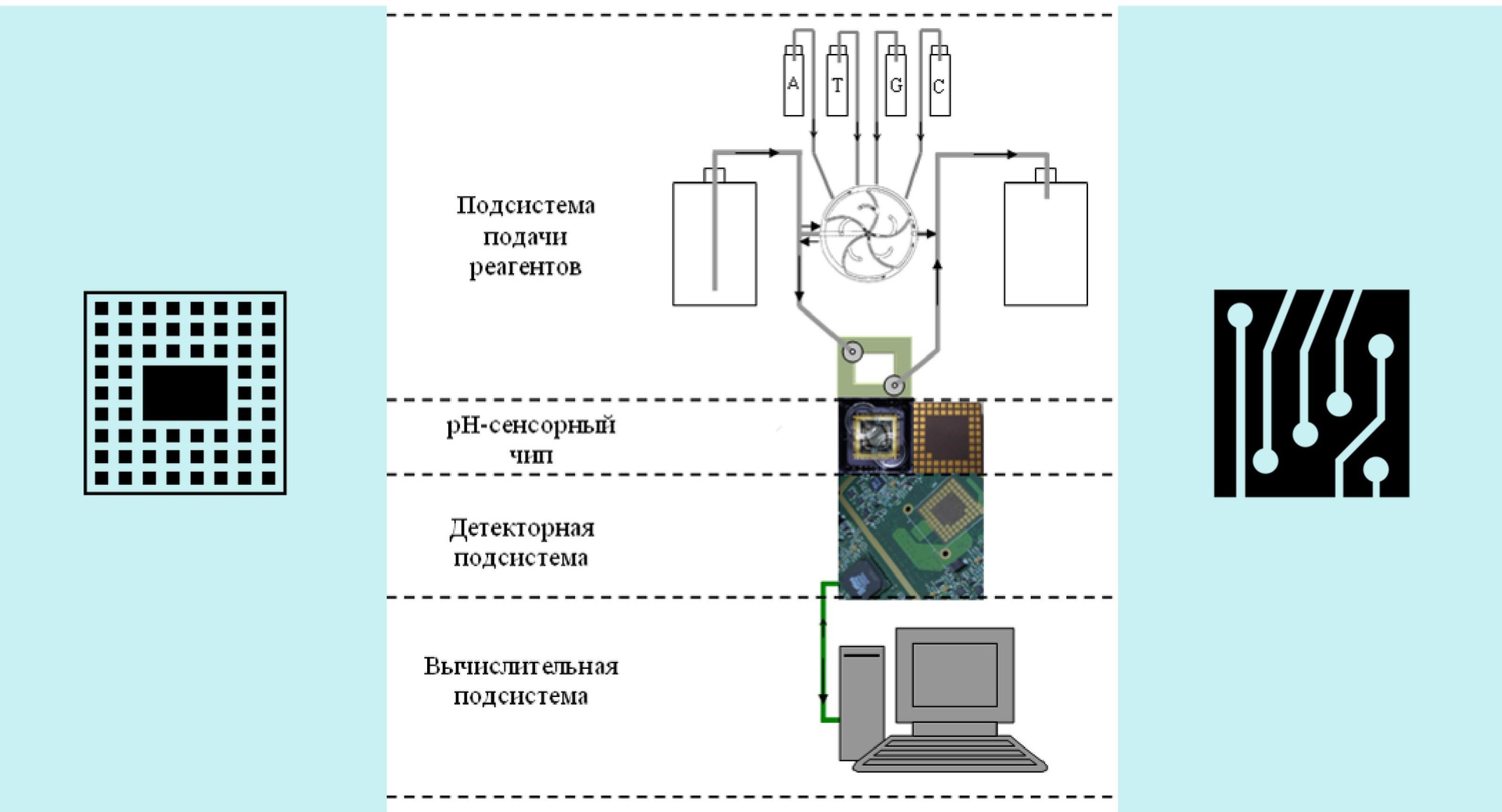
Рис. 2. Устройство секвенатора
Чип позиционируется как одноразовый и после использования выкидывается. Соответственно, там, где работает PGM, такие чипы можно достать бесплатно в любом количестве. Зачем их доставать, спросите вы? Дело в том, что чип мне уже удалось использовать многократно. По сути он вечен: достаточно хорошо промывать его – и можно применять вновь и вновь. По точности работы он ничем не будет отличаться от нового. Сама моя идея заключалась в том, чтобы сделать прибор под этот условно бесплатный чип.
Итак, передо мной встала задача реверс-инжиниринга чипа. Разумеется, никакой документации на заветную микросхему найти было нельзя – производитель не собирался делиться секретами производства, а хотел спокойно продавать свои приборы за $50 000. Для начала я сделал самое очевидное и простое: прозвонил контакты тестером. Стало ясно, где расположены цифровые и аналоговые входы-выходы, питание и прочее. Кое-какую информацию удалось почерпнуть из патентов на чип. Но всего этого, понятно, было недостаточно для создания полноценного продукта. Я еще повозился с чипом, проверял разные свои догадки, поэкспериментировал с подачей сигналов, но никуда принципиально не продвинулся. Пришлось поставить проект на паузу.

Рис. 3. Прозвонка чипа
А затем внезапно на Habrahabr мне попалась статья известного блоггера BarsMonster о том, как он делает реверс-инжиниринг чипов! Воодушевился, написал ему, написал другим энтузиастам, отправил запрос в Киев, где занимались фотографированием чипов. Из Киева ответили, что полировать по слоям они не умеют, могут только отснять верхний слой, а так как мой чип – многослойный, будет не понятно, куда идут дорожки от контактов. Потом познакомился с одним американцем, который тоже занимается реверс-инжинирингом чипов, послал ему свои микросхемы, но и тут дальше фотографирования верхнего слоя дело не пошло. Затем наткнулся в интернете на статью про тех, кто смог отреверсить чип Sony PlayStation и пр. («Слава героям!» и вот это все, если кто в курсе). Решил написать им с вопросами, нашел их ники – и тут же понял, что один из них мне знаком. Недавно товарищ свел меня со своим другом, который «тоже занимается генетикой на любительском уровне», мы пообщались с этим другом в Skype и на этом диалог закончили. И вот я понимаю, что мой новый приятель – мегакрутой мастер реверс-инжиниринга чипов. Тут же написал ему. Однако выяснилось, что, хоть помочь он и готов, у него нет микроскопа. Снова тупик.
А через несколько месяцев нужный микроскоп нашелся в соседней лаборатории! Правда, встроенная в него камера была ужасной, я фотографировал на мобильный телефон через окуляр и получал снимки вот такого качества:

Рис. 4. Чип под микроскопом
Затем на последний Новый год отличный микроскоп за 130 тыс. появился у меня на работе (я – специалист по квантовой криптографии). Мечты сбываются. Наконец, я смог нормально сфотографировать чип сверху.
Рис. 5. Мой рабочий микроскоп
А потом… Потом мне все-таки пришлось самому освоить технику его полировки. Трудность полировки заключается в том, чтобы снимать слои металла толщиной порядка 1 микрона – при этом ширина чипа составляет 1 сантиметр. Для сравнения скажу, что это примерно то же, что допустить на 1 км погрешность не более 10 см. Я очень старался. Результаты моих трудов представлены на следующем фото:

Рис. 6. Реверс-инжиниринг под оптическим микроскопом
Довольно хорошо видны нижний кремниевый слой, верхний слой с транзисторами, первый, второй, третий и четвертый слои металла.
Чип состоит из повторяющихся зон (типа сдвиговых регистров), и по таким картинкам было очень удобно его анализировать: сразу становилось ясно, что происходит на разных слоях. Я «отреверсил» самые «нафаршированные» участки с обилием логики, которые многократно повторялись. Но самым сложным оказалось отследить трассы, идущие по всему чипу, понять, какой внешний контакт к чему относится. С новогодних праздников до конца февраля, я, вооружившись новым прекрасным микроскопом, корпел над этой задачей – сидел на работе до десяти ночи, «реверсил», думал. И тут произошло новое чудо: товарищ смог организовать бесплатную фотосъемку чипа по слоям на электронном микроскопе в МИРЭА. «Фотосессия» крохи в 1 кв. см представляла собой 50 ГБ черно-белых фотографий.
Теперь все эти отдельные фотографии нужно было каким-то образом объединить в одну целую картинку. Чуть ли не в тот же день я написал на «питоне» программу, которая генерировала HTML-файл – при его открытии в браузере я получал требуемое. (Кстати, самая старая 10-я Opera справилась с этим лучше всего, рекомендую!) Затем на javascript написал еще одну программу, позволяющую сравнивать слои, плавно переходить между ними, выравнивать их, подбирать масштаб и т. д. Наконец, в моих руках были все инструменты для решения главных задач. Я отследил трассы, пронизывающие чип, и восстановил всю его структуру до последнего транзистора.
Еще одна фотография среза чипа, сделанная под рентгеном (в МИРЭА):
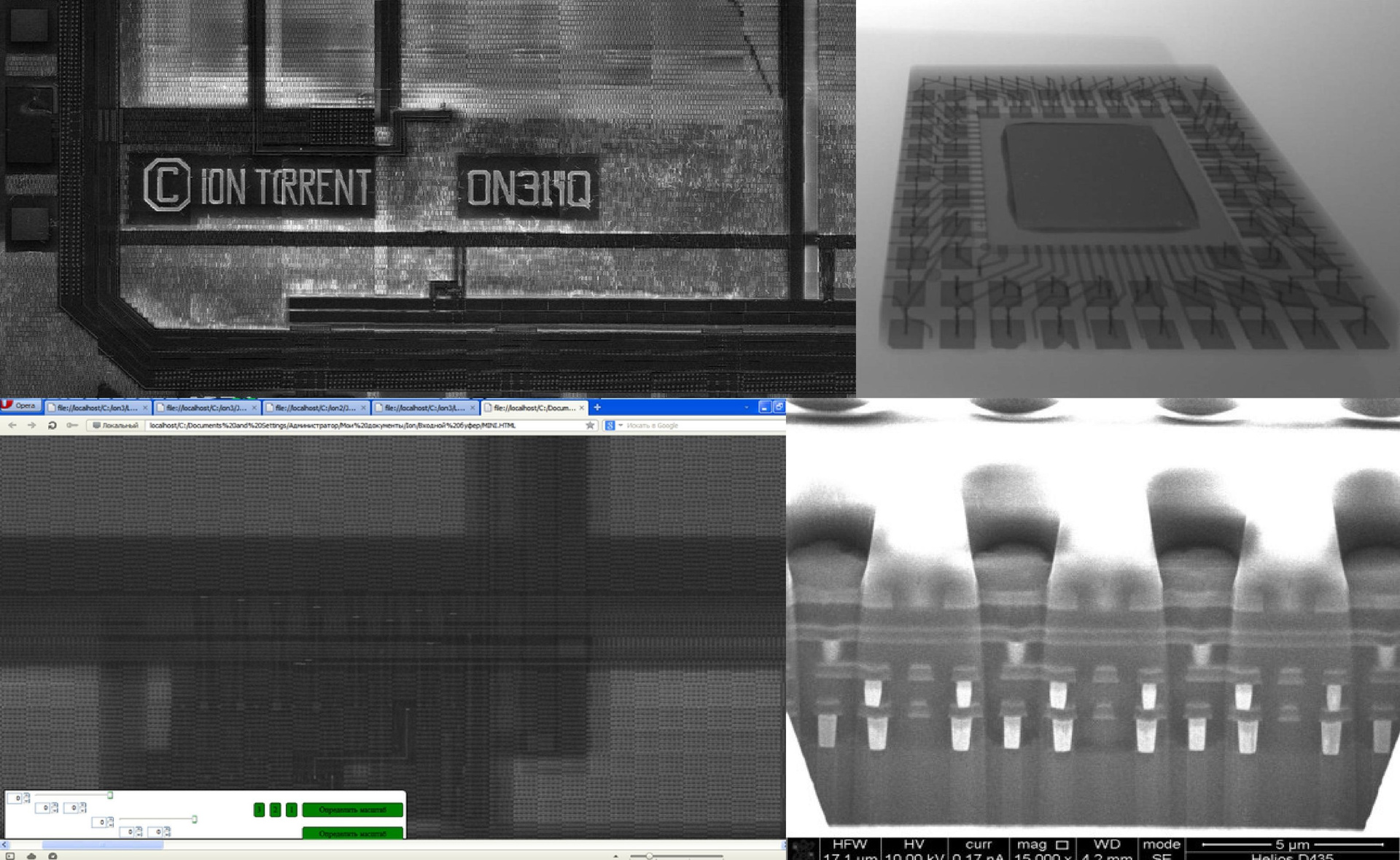
Рис. 7. Съемка под электронным микроскопом
Хорошо видны лунки, куда попадают сферы с ридами. Ниже располагаются три слоя металла, а еще ниже – слой с транзисторами.
Следующим этапом борьбы за светлое будущее стало создание под чип материнской платы. Спроектировал ее и отправил заказ на производство. А пока суд да дело использовал для работы с чипом плату «Марсоход-2» с FPGA. (FPGA – это, грубо говоря, массив из 10 000 универсальных логических элементов; программируя FPGA, мы можем получать любую логическую схему, легко обрабатывающую гигабитные потоки информации.) Прошивку для FPGA я написал сам, а кроме того, для динамического управления системой написал софт, который задает всю конфигурацию для FPGA. Потом вновь образовался полугодовой перерыв (я разводился, ездил в командировку на Байкал, готовил в лаборатории установку, которую демонстрировали Путину). Но в конце концов звезды сошлись: у меня появилось время, приехали готовые платы – и я собрал свою систему.
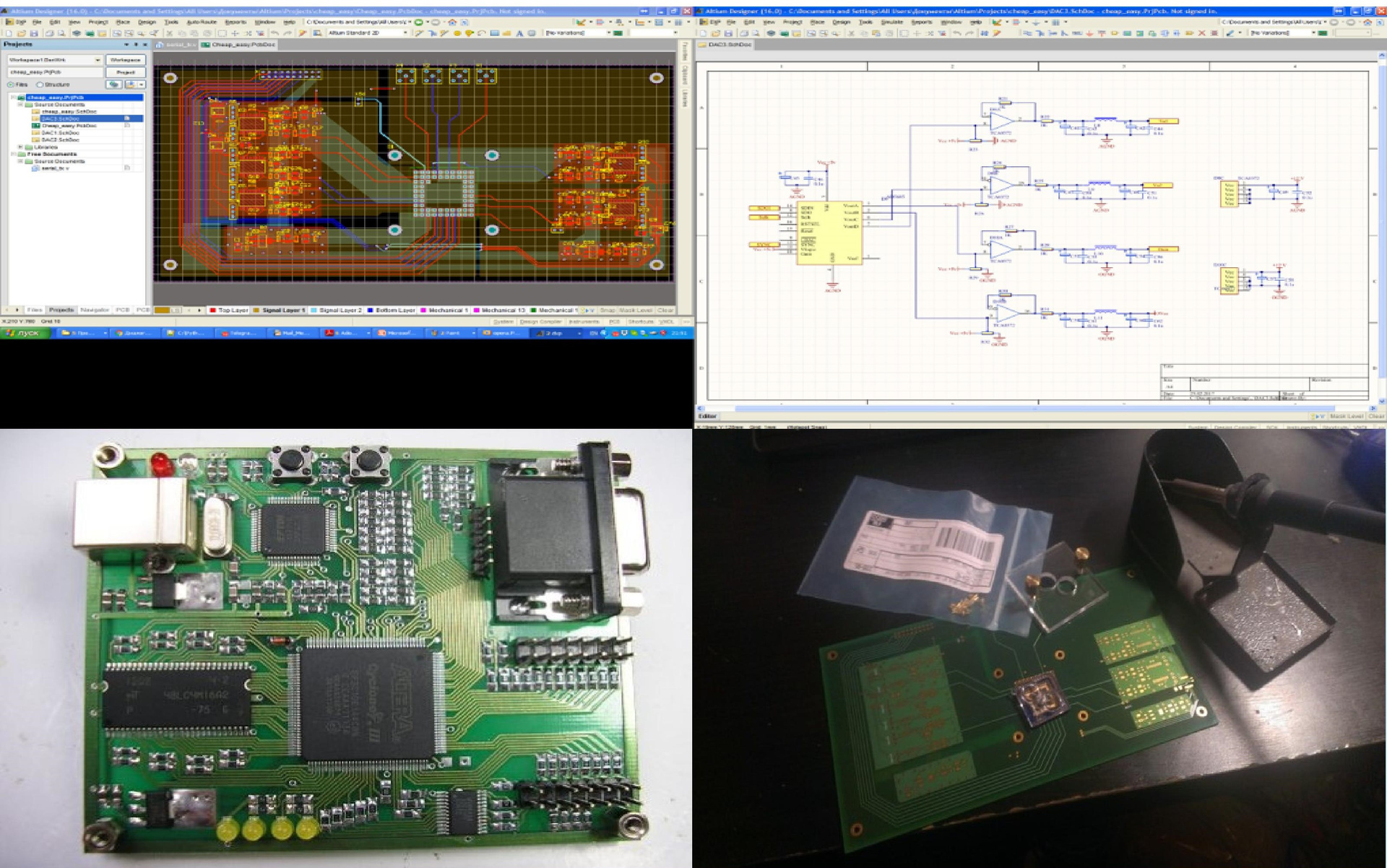
Рис. 8. Создание «железа»
Подал все необходимые сигналы и – о, чудо! – увидел на осциллографе сигнал с чипа. (Осциллограф я купил когда-то за 6 000 рублей на eBay, еще 1 000 стоила прошивка к нему.) На картинке хорошо видны пятна – капельки какого-то реагента.
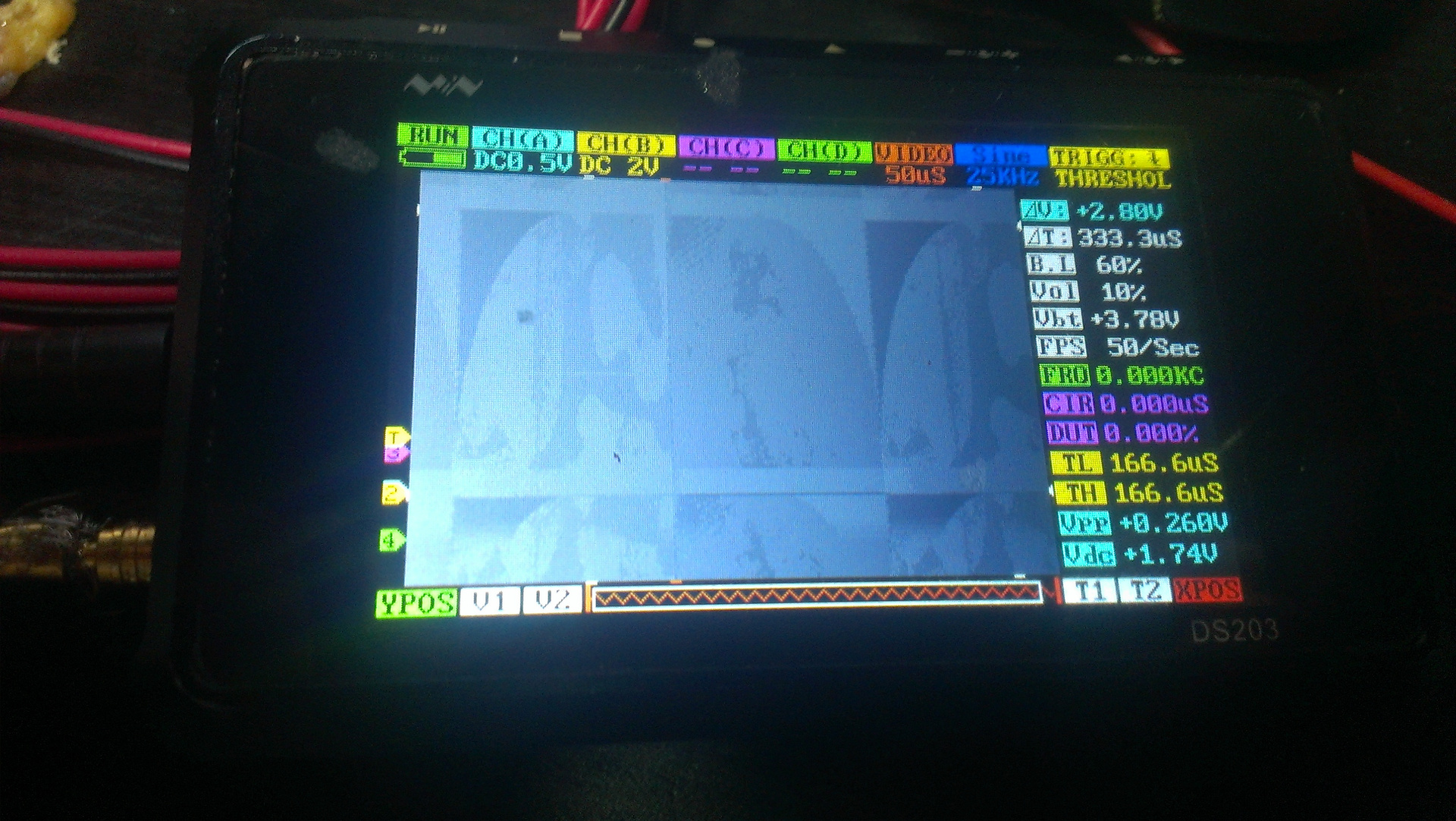
Рис. 9. Сигнал с чипа на осциллографе
Теперь мне нужно было придумать, как оцифровать эту картинку и передать ее на компьютер. Я собрал вот такую установку:
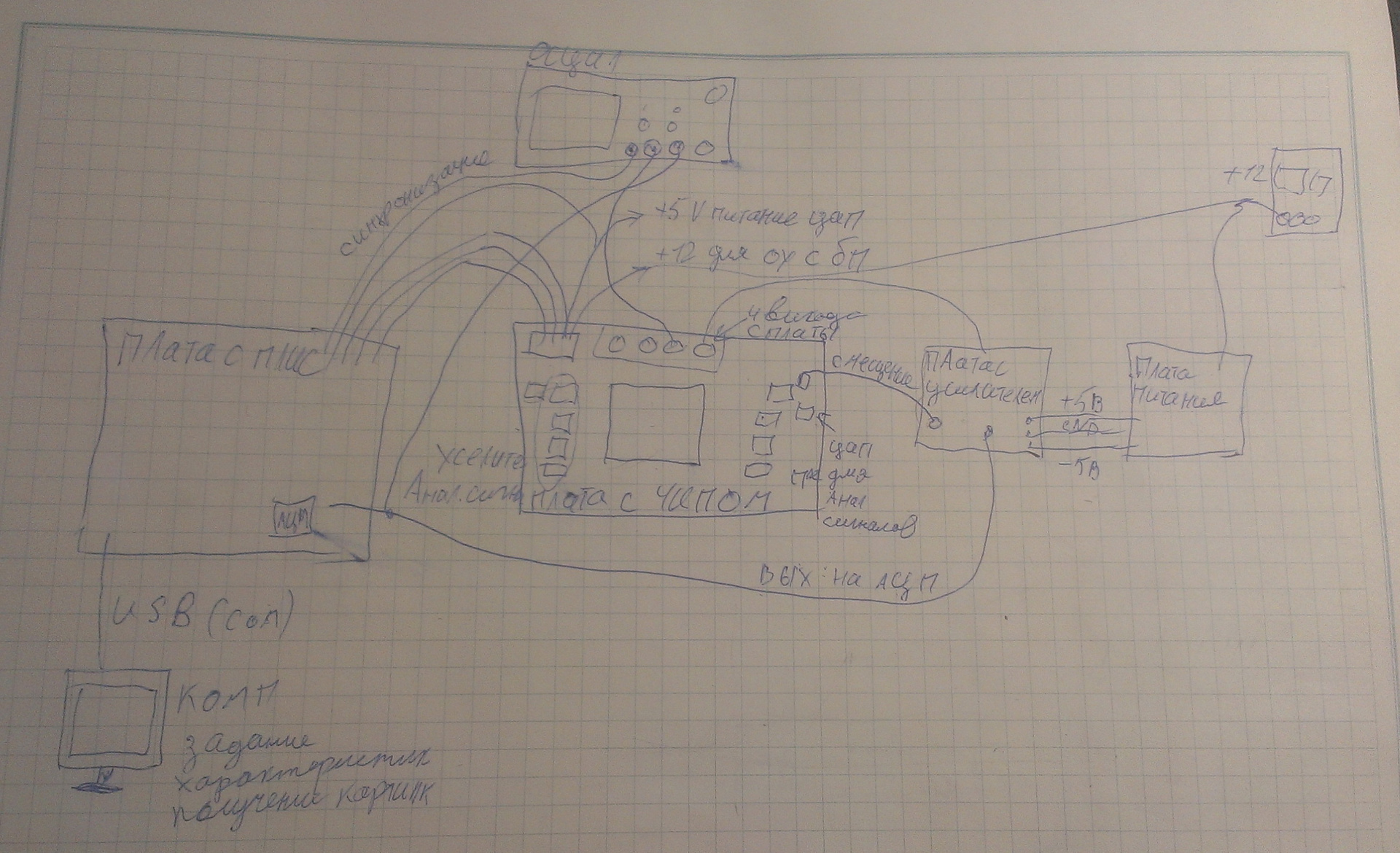
Рис.10. Схема прибора

Рис. 11. Готовая установка
Есть компьютер, который подает данные управления на плату с FPGA. Плата генерирует цифровые сигналы и отправляет их на чип. Сигнал с чипа идет на усилитель, далее – на АЦП на плате, оцифровывается и передается через COM-порт на компьютер. Вообще, пропускная способность COM-порта невелика: 15 килобит в секунду (т. к. в одном чипе находится от 1 млн до 10 млн «пикселей», а максимальная скорость передачи – 115200 бод). Тем не менее картинка на компьютер в итоге попадает.

Рис. 12. Обработанный сигнал на компьютере.
На фото выше видно, что, когда на использованный б/у-шный чип подается библиотека ДНК, чип заполняется неравномерно: по краям – в меньшей степени. Разные цвета обусловлены разным напряжением на pH-транзисторах. То есть мы можем ясно различить те лунки, куда попали сферы с ридами – впоследствии это поможет нам контролировать промывку чипа.
Соответственно, следующей задачей стала промывка чипа. Нужно было добиться, чтобы он стал, как новый. К счастью, у меня имелся совершенно новый чип в качестве референсного образца. На илл. А видно, что в активной области такой чип практически одного цвета (вертикальные повторяющиеся полосы – это просто шумы, наводки).

Рис. 13. Промывка чипа
На рис. 13 B неудачно промытый чип – он разноцветный. На рис.13 D – использованный, но хорошо промытый чип. Видно, что градиент по краям исчез. Тем не менее стоило бы еще доказать, что он действительно чистый и может использоваться повторно.
Поскольку библиотеки ДНК прикрепляются к танталовому покрытию чипа в кислой среде и открепляются – в щелочной (то есть при высоком pH), то чип промывается с помощью специальных полуавтоматических пипеток растворами с разными pH. На сегодняшний день мне удалось добиться практически полной очистки чипа.
У меня интересовались, почему, когда я полностью разобрался в структуре чипа, я не стал заказывать его изготовление, а предпочел по-прежнему искать и доставать б/у-шные, возиться с их промывкой и т. п. Да потому, что разработка микросхемы стоит огромных денег, миллионы долларов, и солидная часть этой суммы уходит на физическую отладку полученного продукта: подгонку, настройку всех параметров транзисторов и т. д. То есть просто скопировать логическую схему – недостаточно. Поэтому я беру условно бесплатную, уже готовую – спроектированную, изготовленную, отлаженную – микросхему и таким образом экономлю значительные средства, серьезно удешевляю проект.
Следующей моей задачей было собрать более продвинутый прибор, который позволял бы быстрее передавать информацию на компьютер и при этом не состоял бы из огромного количества отдельных плат.
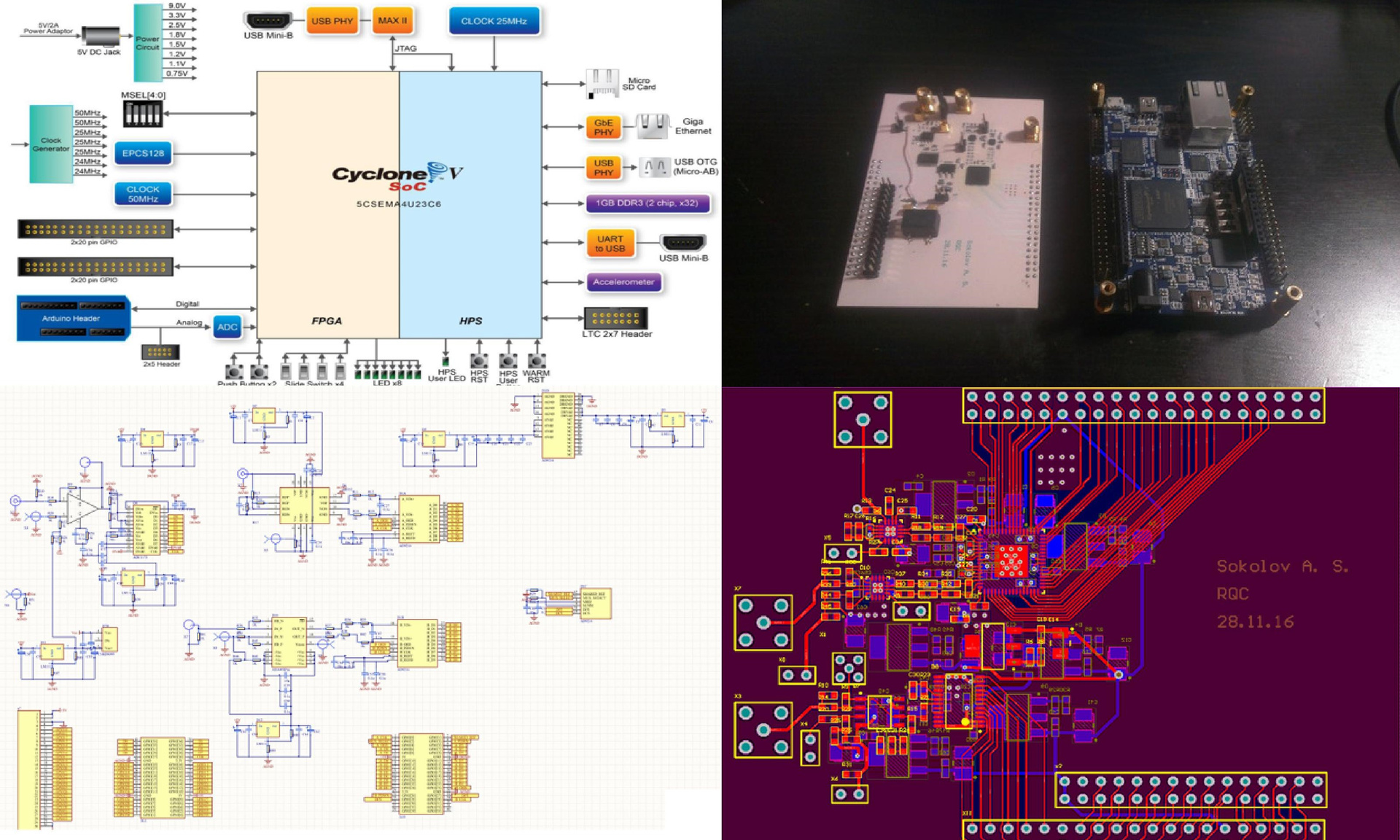
Рис.14 Разработка следующей версии прибора
Я взял новую плату с FPGA – на том же кристалле здесь было 2 ARM-ядра с Linux, имелся Gigabit Ethernet и прочие «плюшки», но зато, в отличие от предыдущего варианта, не было АЦП. Позже спроектировал еще одну плату, с высокоскоростными АЦП и всеми другими необходимыми элементами. Запустил – все заработало.
Что осталось сделать для появления финального прибора? Всего три вещи.
Первое. Нужен гигабитный интернет, быстрая передача данных на компьютер. Это я реализовал буквально вчера.
Второе. Система подачи реагентов. Проектирование специального клапана уже в процессе.
Третье. Софт для обработки информации с чипа. С ПО пока есть вопросы, поэтому приглашаю к сотрудничеству программистов.
Финальный прибор стоит 10 млн рублей. Себестоимость секвенирования составляет несколько тысяч долларов. Чипы обходятся от 100 до 1000 долларов – в зависимости от количества «пикселей» в них. (К слову, восстановление чипов само по себе может стать неплохим заработком, особенно учитывая, что для промывки нужно сделать лишь пару кликов.) Реагенты тоже покупаются, но в перспективе будут создаваться и они.
В общем все это очень интересно, но главное – за этим будущее. Сегодня биотехнологии занимают в мировом научно-техническом прогрессе то же место, что компьютерные технологии в 80-х гг. прошлого века. При этом секвенирование – одно из ключевых направлений для современной биологии и медицины. Ну и, конечно, биотехнологии – это очень прибыльно.
В последнее время на рынке появился полупроводниковый секвенатор S5, и в ближайшее будущие я планирую переключиться на него.
Буду рад пообщаться со всеми, кто захочет тем или иным образом поучаствовать в развитии этого проекта!
Проект был бы не возможен, без теоретической подготовки Владимира Зубова. Выражаю ему свою благодарность.
Спасибо за внимание!
Комментарии (171)

neochapay
31.07.2017 17:32+19Вы просто хардкорный маньяк в лучшем смысле этого слова… так на раз-два отреверсить спаять спроектировать… это ж какой объём знаний в голове держать то надо?

AlexanderS
01.08.2017 10:47+4Это при чтении так кажется, так как всё сжато. А вы внимательно посмотрите сколько у автора это заняло времени. Очень упорный труд, который достойно вознаградился. Уважаю.

neochapay
01.08.2017 11:21+3Да даже у людей знакомых с квантовой механикой и микробиологией с микроэволюцией это всё равно звучит как «Сейчас мы возьмём кусок адамантия потрём его криптонитом, смажем электролитом из грузинских конденсаторов...» и тыды…

frees2
31.07.2017 17:45-34Бизнес на этом, продают «ген мухаммеда» и «ген еврея», притом один и тот же. Лет пять назад стало модно «ген национальности» продавать. Расценки удивительны, прайслисты. В последнее время стихло или не так рекламируется.
Кроме этих фейков можно как то практически применять?

igorkozinov
31.07.2017 17:58+5А потом производители отреверсенной железки его засудят… Патаму чта их права нарушелесь!!!

UA3MQJ
01.08.2017 11:13+1Либо использовать б/у как-то запретят

amarao
01.08.2017 16:30Если бы я был мудаком-копирастом, то я бы просто выжигал чип в оригинальном устройстве. Как минимум, проблему б/у это решит.

Halt
04.08.2017 09:04С учетом того, что чип отреверсили, вполне могут появиться китайские аналоги.

vvzvlad
04.08.2017 21:02Эм. Реверс восстанавливает как бы только логическую схему. Для производства такого же чипа надо еще пилить и пилить — по затратам будет чуть меньше, чем с нуля разрабатывать. Если бы китайцы видели в этом смысл, они бы уже давно купили чип и скопировали его сами)

nikosias
31.07.2017 18:24-11Вы не пробовали через нейросеть пропускать данные?
Не могли бы поподробнее рассказать как формируются данные?AllexIn
31.07.2017 21:46+19Что значит «прогнать через нейросеть»?
Сеть на что должна быть обучена? И каким образом?
Часто вижу эту фразу «попробуй прогнать через нейросеть». Каждый раз ощущение, что написавшией её сичтает нейросеть некой крутой универсальной штукой, которая автоматически выдаст интересный результат на любых исходных данных.
Это не так. Сеть должна быть обучена. И только тогда она может что-то анализировать.vedenin1980
31.07.2017 22:01-5Это не так. Сеть должна быть обучена. И только тогда она может что-то анализировать.
Не совсем так, можно просто скормить сети данные и она построит какие-то группы (задача кластеризации). Например, разделит долгожителей и остальных или тех у кого талант к музыки и у кого нет. Анализируя полученные группы в теории можно тоже получить интересные результаты.
Можно например скармливать сети как геном, так болезни, увлечения, профессии и таланты, в теории она может сама найти интересные связи, даже если ее ничему реально не учить, просто отдавать сырые данные (любые вплоть до любви к огурцам).
Кстати, удивительно что никто не пытается сети скарвливать все данные о челевеке и геном, чтобы посмотреть результат.AllexIn
31.07.2017 22:05+9Необученная — это с коэфициентами от балды?
Честно говоря первый раз слышу, что случайные коэфициенты могут делать какую-то полезную работу.
Звучит как бред… Впрочем, не возьмусь утверждать. Пойду курить актуальные материалы.
Спасибо за правку.
Garbage in - garbage out
vedenin1980
31.07.2017 22:17+1Обучение без учителя (самообучение, спонтанное обучение) — один из способов машинного обучения, при котором испытуемая система спонтанно обучается выполнять поставленную задачу без вмешательства со стороны экспериментатора. С точки зрения кибернетики, это является одним из видов кибернетического эксперимента. Как правило, это пригодно только для задач, в которых известны описания множества объектов (обучающей выборки), и требуется обнаружить внутренние взаимосвязи, зависимости, закономерности, существующие между объектами.
случайные коэфициенты могут делать какую-то полезную работу.
Они настраиваются, через определенное время они уже не случайныAllexIn
31.07.2017 22:18+5А. Вот вы о чём. Ну так это просто один из способов обучения.
vedenin1980
31.07.2017 22:24-1Ну так это просто один из способов обучения
В данном случае есть просто получает некий набор сырых данных, и должна найти какие-то связи, при этом ей не говорят что она должна искать и не показывают примеры. В данном случае это именно «прогнать через сеть данные». Она, конечно, должна обучиться (то есть иметь какой-то достаточно большой набор данных), но это просто вопрос статистики, так как машинное обучение иметь смысл при достаточной выборке.

KiloLeo
31.07.2017 23:41+4Начнём с того, что этих данных у автора просто нет. Чтобы сделать то, о чём Вы пишете, надо иметь набор данных: Геном (3Гб), продолжительность жизни (т.е. брать геном надо у трупа), наличие таланта к музыке,… пр. И желательно на несколько сот тысяч человек. Ага. Автору пока есть чем заняться :-)
vedenin1980
01.08.2017 00:10-1В данном случае, я отвечал не на предложение автору использовать машинное обучение, а на то что сети должны быть только «обученные» (то есть, что не может быть сети, которая обучится совсем сама).
К тому же, конечно ставить глобальные цели вроде расшифровки всего генома при такой выборки бессмысленно, а вот если делать много анализов генома у сотен больных с определенной генетической болезнью (например, если это требуется по работе) и у сотен здоровых людей, то всего лишь загнав данные в систему машинного обучения — есть шанс найти корреляции генов и данной болезни, что может быть полезным (даже при том что корреляция еще не означает следствия). То есть глобальные цели — так не достичь, а локальные на медицинскую статью — вполне можно.
stychos
03.08.2017 14:08Вот и тема для стартапа: сделать библиотеку бесплатных днк-библиотек, openDNA.

Kriminalist
04.08.2017 16:18Бренд занят — http://opendna.ai/, а идея давно не стартап — https://www.openhumans.org

BelerafonL
01.08.2017 12:28+1Автору нужно из сигнала матрицы «фотокамеры» получить кусочки ДНК длиной 300 нуклеотидов — «риды». Здесь и речи не идет о получении целого ДНК человека. Хорошо если вирус получится прочитать за несколько заходов.
Нейросеть можно было бы обучить анализировать сигнал этой матрицы и собирать из них эти «риды», но для этого нужно точно знать, какой кусок ДНК там секвенируется априори для «обучения с учителем». И сделать скажем 10000 таких опытов с разными «точно известными» ридами, набрать базу обучения и тогда можно попробовать дать это нейросети.
Судя по описанию, что чтобы прочитать один (один!) нуклеотид одного рида нужно капать на матрицу реагент нужного типа, а потом его смывать (судя по описанной технологии PGM), потом капать другой… то в домашних условиях получить такую выборку не видится возможным.

nikosias
01.08.2017 11:15Я имел ввиду, что можно пред очищать данные если я праельно понял то данных с чипа идет много часть из них повторяется часть из за того что чип не совсем хорошо очищен может быть искажена. И с помощью нейросетей которые хорошо работают с искаженными данными, попытаться их вытащить.
Для того чтобы развить свою мысль и попросил уточнить в каком виде приходят данные.

barbanel
02.08.2017 14:43+3Что значит «прогнать через нейросеть»?
Пффф! Очень просто!
«Посмотреть в компьютере» же надо!

kgbplus
01.08.2017 11:20+1Мне кажется, вы не представляете какой там объем данных. Там SVM годы будет работать…
carpaccio
31.07.2017 18:24+22Вы сделали классическую работу целого советского НИИ в одиночку, очень круто. Единственная проблема в том, что надо сделать validation на большом количестве последовательностей чтобы быть уверенным в том, что полученные результаты достаточно точны. А когда и если вы решите эту проблему, то окажется, что сделанный прибор сильно устарел, и на рынке есть горздо более эффективные решения. Но проект замечательный!

sintech
31.07.2017 19:00+7Не согласен. На выходе НИИ была бы технология производства аналогов таких чипов в промышленных масштабах. А сейчас автор при всем к нему уважении, зависим от отходов коммерческих секвенаторов. Производители которых могут в любой момент сделать так, чтобы чипы нельзя было использовать повторно. Но к сожалению таков капитализм.
carpaccio
31.07.2017 19:12+7Чипы использовать повторно крайне чревато. В отличие от заправки картриджей косяки тут глазом увидеть невозможно.

redpax
07.08.2017 13:37Почему же? Многократное сравнение с результатами с нового чипа вполне себе даст понимание точности на БУ чипах при рассмотрении одинакового материала.

NiTr0_ua
01.08.2017 01:50+2Ну ок, 2/3 работы — по спецификациям/схемам/послойным шаблонам сделать чип-клон таки намного проще, чем имея чип (без какой-либо обвязки) получить схему и разводку слоев, а также — протокол управления.

AlexanderS
01.08.2017 10:54+3Вы слишком гипертрофируете. У меня на работе тоже каждый человек ведёт свою часть работы, которые в советские времена делало по доброй половине НИИ. А за пару месяцев мы делаем больше, чем раньше целые почтовые ящики пилили годами)
Автор молодец, всё это способствует прокачиванию понимания многих вещей и вообще — когда узнаёшь что-то новое и делаешь сам — это чертовски интересно! Я понимаю — сам немножко такой)

censor2005
31.07.2017 18:24+15Для меня эта статья подобна «Как собрать термоядерный реактор в чулане за 100$». Звучит просто невероятно!
Куча вопросов родилась. Сколько времени ушло на реверс-инжиниринг чипа по фото? Делали это вручную, или есть какие то программы облегчающие построение схемы? И сколько элементов примерно в микросхеме? Насколько точным будет секвенирование ДНК в б.у. чипе? Спасибо большое!
Navij
31.07.2017 19:25+4Ну так-то не в чулане, у автора был доступ к лабораторному оборудованию всё-таки. Что, впрочем, не умаляет невероятной крутости того, что он сделал.

robo2k
31.07.2017 18:26+22Как подается биоматериал на анализ? Как ДНК извлекается из ядра? Как ДНК делится на сегменты? Так много вопросов, и так мало ответов. Да и вообще, чувствую себя пещерным человеком на лекции в ВУЗе.

BlessYourHeart
31.07.2017 18:45+6В этой технологии чип — одна из главных деталей, и один из самых дорогих расходников.
Как принтер, в котором картридж стоит относительно дорого.
Собственно и промывка чипов похожа на перезаправку картриджей — для частных лиц и отдельных способов применения промывка подойдет, но ни одна лицензированная лаборатория не будет использовать отмытые чипы в своей деятельности.Garbus
31.07.2017 19:26К принтерам производители «догадались» приспособить СНПЧ, и тут со временем наладят промывку и повторное использование. Вычислительные мощности ныне позволяют вытягивать информацию порой из отвратительных исходников. Путь чип станет не вечным, а на десяток-другой циклов — уже приятное удешевление.

BlessYourHeart
31.07.2017 19:53+1Все же это гораздо более узкий рынок, чем принтеры.
Опять же вопрос в том, что для данного вида чипов автор подобрал промывку, а как промывать чипы со специфическими антителами, и не будет ли это дороже, чем покупать новые?.. Это тоже ставит вопросы о том, насколько этот рынок мытых чипов широк.
Garbus
31.07.2017 21:06Ну не рассказывать же о том, как бизнес «ленится», став монополистом? Может некоторый стимул, в виде призрачных недорогих наборов для промывки, заставит выпустить более удобное и недорогое решение пораньше. Думаю рынок пока далек от насыщения, наверняка использование сильно сдерживает цена.

BlessYourHeart
31.07.2017 23:53+4Недорогие наборы для промывки подойдут для энтузиастов, для "домашнего" применения, для некоторых видов исследований. Это относительно маленький рынок дня технологии, ну просто потому, что энтузиастов разного рода мало, для "домашнего" применения надо придумать прикладные задачи (это крайне сложно для такой узкоспециализированной системы), а исследователи обычно могут позволить себе покупать оригинальные расходники.
Повторюсь, что в медицинское применение "отмытых" чипов я не верю, так как это крайне зарегулированная сфера.
А сам пост автора меня наводит на мысли о жутком разрыве между возможностями современных технологий, их доступности и сложностью их осознания и понимания их возможностей. Одни люди разбираются в домашних условиях с, буквально, нуклеотидами, а другие боятся ГМО и верят в заговор учёных...
Garbus
01.08.2017 04:02+2Думаю потенциальный рынок «домашних» исследований все же не мал. Селекционеров любителей и профессионалов хватает, неужели они откажутся от недорогого и удобного прибора, пусть даже с «китайским» уклоном? Правда нужно хорошее ПО и информационная база, но уже проще в развитии, когда есть что анализировать.
Ну или другие исследования, прорабатываем теорию на дешевых расходниках, а подтверждаем на оригинальных. Тоже ведь неплохая экономия.
AllexIn
31.07.2017 21:48+4Почему узкий-то? Определение генетических заболеваний — это очень востребованная штука.
Впрочем я и 1000 готов заплатить за это. А если будет 100$ — рынок значительно вырастет. ИМХО
BlessYourHeart
31.07.2017 22:40+1Я к сожалению не знаком с применением секвенирования для прямого определения генетических заболеваний.
Насколько я знаю, сейчас используются маркерные методы, а расшифровка днк даёт только некоторую вероятность и/или склонность к заболеваниям, что, согласитесь, от диагностических методов далеко.
А рынок любопытствующих, которые готовы заплатить за свой профиль и знать свои особенности, включая склонность к патологиям, по моему мнению сейчас достаточно покрыт существующими компаниями, вроде того же Атласа.
golf2109
31.07.2017 19:32максимальная скорость передачи – 115200 бод
921600 уже давно широко применяетсяя

kablag
31.07.2017 19:36+1Александр, Вы с этой идеей в компанию Синтол не обращались? Российский секвенатор по Сэнгеру они уже сделали совместно с Институтом аналитического приборостроения РАН — может и на полногеномный замахнутся.

read2only
31.07.2017 20:06+10Пожалуй, это самый интересный и, к сожалению, крайне редкий теперь вид публикаций на Geektimes.
Удачи вам Александр!

Valerij56
31.07.2017 20:16+2Мне тоже кажется, что автор где-то не там работает. Думаю, его уже взяли а карандаш хэдхнтеры.
И, да, Александр, это неимоверно круто. Удачи вам, и хватит изобретать велосипеды!

ra3vdx
31.07.2017 20:18+4Даа, впечатляет! Это не LM317 исследовать)
Всё же правы биологи — мы живём в самой лавине третьей — геномной — революции. Осталось научиться интерпретировать все данные (или хотя бы бОльшую часть). Поскольку знаем мы сегодня крайне мало о регуляторных последовательностях и о межгенном взаимодействии.
А вот видео, где Фёдор Кондрашов про эту технологию рассказывает, кому интересно (с меткой времени):


DROS
Как скучно я живу. Удачи, даже не знаю в чем, да во всем вообще. Ваша энергия пошла в нужном русле. как мне кажется. Это нереально круто. Ждем продолжения.