Расскажем, почему появился этот инструмент и что он умеет.

Нехватка алгоритмов
Одна из ключевых задач машинного обучения — снижение размерности данных. Дата-саентисты сокращают число переменных, вычленяя среди них значения, наибольшим образом влияющие на результат. После этой операции модель машинного обучения требует меньше памяти, работает быстрее и качественнее. Пример ниже показывает, что исключение дублирующих признаков увеличивает точность классификации с 0,903 до 0,943.
>>> from sklearn.linear_model import SGDClassifier
>>> from ITMO_FS.embedded import MOS
>>> X, y = make_classification(n_samples=300, n_features=10, random_state=0, n_informative=2)
>>> sel = MOS()
>>> trX = sel.fit_transform(X, y, smote=False)
>>> cl1 = SGDClassifier()
>>> cl1.fit(X, y)
>>> cl1.score(X, y)
0.9033333333333333
>>> cl2 = SGDClassifier()
>>> cl2.fit(trX, y)
>>> cl2.score(trX, y)
0.9433333333333334Существует два подхода к уменьшению размерности — конструирование и выбор признаков. В областях вроде биоинформатики и медицины чаще используют последний, так как он позволяет выделить значимые признаки с сохранением семантики, то есть не меняет исходный смысл признаков. Однако в самых распространенных библиотеках машинного обучения на Python — scikit-learn, pytorch, keras, tensorflow — нет полноценного набора методов выбора признаков.
Для решения этой проблемы студенты и аспиранты Университета ИТМО разработали открытую библиотеку — ITMO_FS. Над ней трудится команда под руководством Ивана Сметанникова, доцента факультета информационных технологий и программирования, заместителя заведующего лабораторией Машинного обучения. Ведущий разработчик — Никита Пильненьский, закончивший магистратуру «Машинное обучение и анализ данных». Теперь он поступает в аспирантуру.
«За последние несколько лет в нашу лабораторию приходили запросы на решение задач, для которых не подходил стандартный инструментарий. Например, нам требовались ансамблирующие алгоритмы на основе объединения фильтров, или алгоритмы, учитывающие наличие заранее известных (экспертно-размеченных) значимых признаков.
Посмотрев на существующие решения, мы пришли к выводу, что они не только не содержат необходимые нам инструменты, но и не являются достаточно гибкими для их возможной мягкой интеграции. В контексте того, что среди таких библиотек конкуренция слаба, мы решили сделать свою библиотеку, исправляющую большинство недостатков».
— Иван Сметанников
Что умеет библиотека
ITMO_FS реализована на Python и совместима со scikit-learn, которая де-факто считается основным инструментом анализа данных. Ее селекторы признаков принимают те же параметры:
data: array-like (2-D list, pandas.Dataframe, numpy.array);
targets: array-like (1-D list, pandas.Series, numpy.array).Библиотека поддерживает все классические подходы к отбору признаков — фильтры, обертки и встраиваемые методы. Среди них числятся такие алгоритмы, как фильтры на основе корреляций Спирмена и Пирсона, критерий соответствия (Fit Criterion), QPFS, hill climbing filter и другие.

Также библиотека поддерживает обучение ансамблей за счет объединения алгоритмов выбора признаков по используемым в них мерам значимости. Такой подход позволяет получать более высокие прогностические результаты при низких временных затратах.
Какие есть аналоги
Библиотек алгоритмов выбора признаков существует не так много — особенно на Python. Одной из крупных считают разработку инженеров из Государственного университета Аризоны (ASU). Она поддерживает большое количество алгоритмов, но последнее время почти не обновляется.
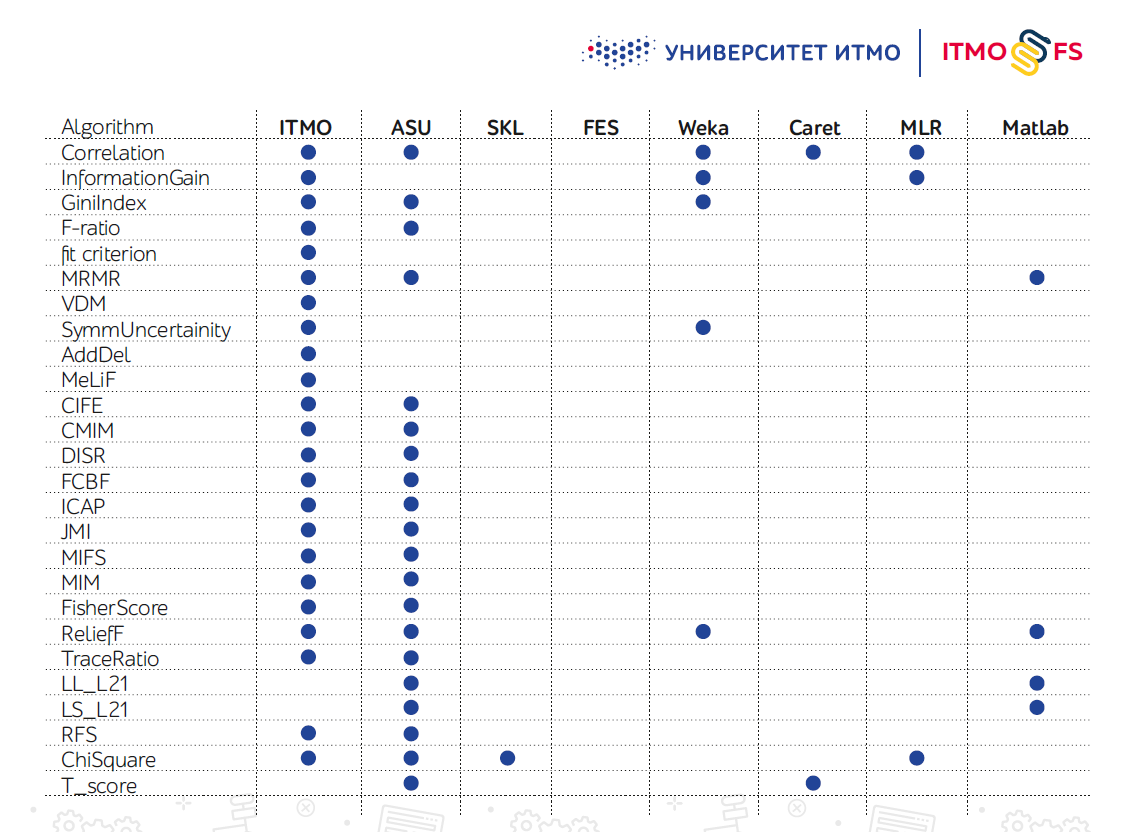
В самой scikit-learn также есть несколько механизмов выбора признаков, но на практике их оказывается недостаточно.
«Вообще за последние пять–семь лет фокус сильно сместился в сторону ансамблирующих алгоритмов выбора признаков, но они особо не представлены в подобных библиотеках, что мы также хотим исправить».
— Иван Сметанников
Перспективы проекта
Авторы ITMO_FS планируют интегрировать свой продукт с scikit-learn, добавив его в список официально совместимых библиотек. На данный момент библиотека уже содержит наибольшее среди всех библиотек число алгоритмов выбора признаков, но их добавление продолжается. Дальше на дорожной карте добавление новых алгоритмов, включая собственные разработки.
В более далеких планах стоят задачи внедрить библиотеку в систему мета-обучения, добавить алгоритмы прямой работы с матричными данными (заполнение пропусков, генерация данных мета-признакового пространства и т. д.), а также графический интерфейс. Параллельно с этим будут проходить хакатоны с использованием библиотеки, чтобы заинтересовать в продукте большее число разработчиков и получить фидбек.
Ожидается, что ITMO_FS найдет применение в областях медицины и биоинформатики — в таких задачах, как диагностика различных раковых заболеваний, построение прогностических моделей фенотипических характеристик (например, возраста человека) и синтез лекарственных препаратов.
Где скачать
Если вас заинтересовал проект ITMO_FS, вы можете скачать библиотеку и опробовать её на практике — вот репозиторий на GitHub. Начальная версия документации доступна на readthedocs. Там же можно посмотреть инструкцию по установке (поддерживается pip). Мы будем рады любой обратной связи.
Дополнительные материалы из нашего блога на Хабре:


NeoPhix
Я бы на вашем месте подумал над сменой названия.
UPPER CASE ни разу не способствует популяризации библиотеки… А еще FS больше напоминает File System, а не Feature Selection.
Но все равно круто, удачи вам :)