
Про биоинформатику слышали многие. Кто-то знает больше, кто-то меньше. Мы постарались раскрыть вопрос этой, относительно новой, науки. Так сказать, дать общие представления читателю об основных вехах развития, методах и проблемах: решённых и существующих на нынешнее время.
Автор сообщества Фанерозой, генетик, ботаник Степан Иванов.
С развитием информационных технологий появилось множество удобств самого разного характера. Не обошли стороной эти удобства и научные исследования. Совершенно неудивительно, что и в биологии появился и нашёл применение целый огромный раздел науки — биоинформатика. Хотя следует отметить, что это и произошло совсем недавно. Так, датой начала использования биоинформатики в биологии можно назвать 1970 год. Тогда Полина Хогевег (нидерландский биолог) и Бен Хеспер ввели термин «биоинформатика», определив его как изучение информационных процессов в биотических системах.
С этого момента биоинформатика развивалась стремительно и неумолимо. В 1970 году Нидельманом-Вуншем была предложена система сравнения аминокислотных и нуклеотидных последовательностей. Эти алгоритмы быстро находили применение в огромном количестве исследований и стали фундаментом для новых открытий. Одним из наиболее важных прорывов стали алгоритмы и программы, позволяющие предсказывать вторичные структуры белков, а на их основе — предсказывать функции отдельных доменов и белков в целом (мы писали об этом здесь).

Вообще, стандартным решением многих биологических вопросов является сравнение полученных данных с эталоном, и информационные технологии, позволяющие перенести эти расчёты на компьютеры. Исследования в этой области знания приносят очень прорывные и технологические плоды.
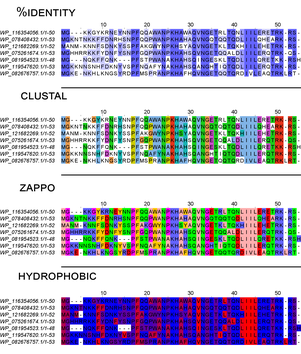
Визуализированный результат работы алгоритмов выравнивания аминокислотных последовательностей:
Вместе с развитием применения информационных технологий развивались и сами методы получения этих данных. Происходило их накопление. Например, на новый уровень выходили методы секвенирования, получали целые геномы модельных организмов, а их расшифровка и аннотирование во многом ложилось на вычислительную технику. Не углубляясь в историю развития методов NGS (next generation sequencing) отметим лишь, что сейчас есть возможность получать до 2 миллионов пар нуклеотидов за одно прочтение по весьма низкой себестоимости (в случае секвенирования методом MinION — стоимость одного миллиона пар нуклеотидов колеблется в районе 1$).

Чем больше становилось референсных данных, тем больше была точность работы программ. Конечно, развивались и алгоритмы, обрабатывающие эти данные. И постепенно интерес в биоинформатике сместился с изучения отдельных частиц к исследованию взаимодействий разных генов. Отсюда даже выделился новый раздел биоинформатики — системная биология (объект — как система связанных процессов и компонентов взаимодействующих друг с другом).

Новые технологии позволили получать все больше данных, в свою очередь, эти массивы охватить человеческим сознанием уже было невозможно, а многие расчёты на компьютерах до сих пор занимают дни и недели (например, сборки больших геномов).
Чтобы не быть голословным, приведём несколько примеров исследований с использованием методов биоинформатики:
-
Предсказание трёхмерной структуры белкаТак, команда DeepMind буквально год назад решила вопрос по предсказанию трёхмерной структуры белка по его аминокислотной последовательности. Хотя сама проблема считалась буквально фундаментальной и сформулированной ещё в 1970-х.Сама идея о том, что аминокислотная последовательность белка несёт в себе информацию о его функциях была высказана нобелевским лауреатом по химии Кристианом Анфинсном. С тех пор это стало вызовом для компьютерных технологий, поскольку альтернативой для выяснения трёхмерной структуры (а значит, и функции белка) были лишь долгие эксперименты, тянувшиеся порой десятки лет для выяснения функции лишь одного белка. В 1969 году проблему обозначил Сайрус Левинталь, оценивший количество конформаций для типичного белка как 10 в 300 степени вариантов. Разработанный программистами ИИ AlphaFold позволяет весьма быстро (2–3 дня расчётов для типичного белка на не самом мощном компьютере) решать вопрос третичной конформации белка, а значит и его функции. Конечно, и здесь остаются проблемы, одна из основных — ИИ обучался на тех белках, для которых третичной структуры уже известны и верификация расчётов для отличных белков остаётся под вопросом. Но даже так, это однозначно прорыв в фундаментальной, структурной биологии. И, однозначно, найдёт своё применение в разработке многих лекарств. Больше можно почитать про ИИ на этом сайте .
-
Сборка геномовНаверняка многие слышали про NGS — секвенирование нового поколения. Методы NGS позволяют получать огромные нуклеотидные последовательности. При этом для того или иного участка по итогу секвенирования может существовать много тысяч прочтений и выравнивание этих фрагментов относительно друг друга сделать человеческими силами просто невозможно. Однако же существует ряд программ, в которых любой исследователь может получить консенсусные последовательности, просто внеся нужные параметры и переложив все вычисления на компьютер.
- Биоинформатические методы в традиционных исследованиях
Помимо обработки молекулярных данных, информационные технологии находят своё применение и в традиционных исследованиях. Например, в исследовании по черепной коробке Bissektipelta archibaldi были использованы матрицы признаков, обработка которых полностью переложена на компьютерные вычисления (Kuzmin I. et al., 2020). Также в работе представлены методы 3D моделирования, которые, безусловно, в данном случае представляют собой аналогичные вычислительные методы, без которых порой не обойтись.
Иным примером использования биоинформатики в традиционных исследованиях может послужить орнитологическая работа исследователей из СПбГУ (Bojarinova J. & Babushkina O., 2015). Ещё в 2006 году были начаты эксперименты по влиянию фотопериоду на двигательную активность воробьиных птиц ( сначала длиннохвостых синиц, а после, и иных видов). Отловленные на Ладожской Орнитологической станции птицы помещались в цилиндрические клетки, с установленными камерами и лампами на таймере. Все движения птиц записывались, а отдельная программа обсчитывала, в какую сторону движения птиц были наиболее активными. Безусловно, сбор такого материала мог быть проведён и самими исследователями, путём ежедневного наблюдения за клетками, но это отняло бы гораздо больше времени, а также сильно увеличилась бы погрешность в данных.
Ещё более интересные и сложные примеры можно посмотреть в данной статье. Это сложно, но интересующимся в теме может быть очень занятно..Эти примеры я вставил не просто так. Ведь та часть биоинформатики, которая является разделом биологии, вносит огромный вклад в развитие этой науки. Отдельно стоит отметить роль статистических методов, которые позволяют делать совершенно новые выводы из накопленных данных. Фундаментальные исследования, даже основанные на экспериментальных данных, все больше прибегают к накоплению данных и применению статистических методов обработки этих данных.
Но биоинформатика не панацея. В этой области науки до сих пор остаются нерешённые и актуальные проблемы. Отвлекаясь от частностей, можно сказать, что одной из проблем остаётся верификация прогнозируемых компьютером данных. Так те же предсказания третичной структуры белка (упомянутая выше работа DeepMind) по аминокислотной последовательности получаются лишь при обучении нейросетей на имеющихся данных по весьма коротким аминокислотным последовательностям. В то время как в живых клетках представлены и намного более сложные белки, чьи функции и конформация до сих пор существуют лишь в виде неподтвержденных моделей. И на нынешний момент подтвердить или опровергнуть те или иные модели современными методами невозможно.По итогу, самым насущным вопросом биоинформатики остаётся совершенствование алгоритмов вычислений и разного рода расчётов по моделям, увеличения статистической поддержки тех или иных вычислений и, как ни странно, накопление эмпирических данных традиционными методами биологии. Это всё делается для того, чтобы впоследствии эти данные могли быть применены для обучения разного рода нейросетей.

Нейросеть обучается. Щепотка плоского юмора.
Информационные технологии находят своё применение в построении экологических моделей. И одной из основополагающих работ в этой области можно назвать работу Лотки-Вольтерры. В работе рассматривается закрытый ареал, в котором обитают два вида — травоядные жертвы и хищники. Модель взаимодействия этих видов описывается математическими уравнениями. Конечно, модель эта неточна и имеет много допущений (пищи для травоядных «жертв» в избытке, паразиты и иные хищники отсутствуют, а ареал закрытый. Комплекс этих допущений в живой природе невозможен), но даже такая имитация представляет существенный интерес для биологии как науки и закладывает основы для развития алгоритмов прогнозирования динамики и эволюции разного рода экосистем.
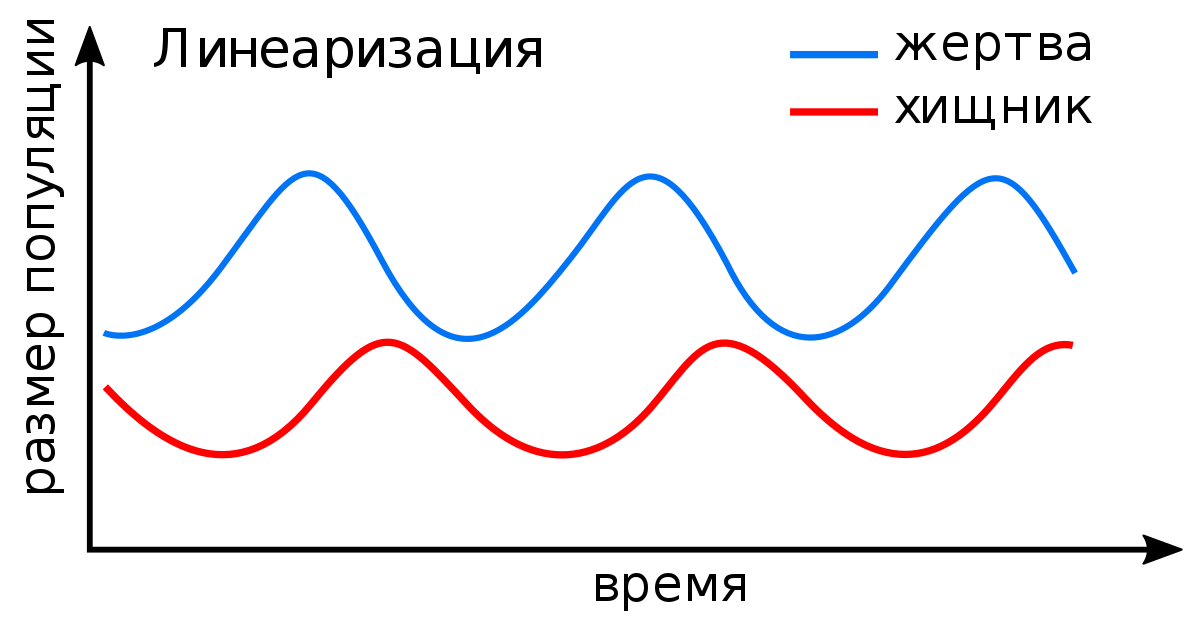 Простая иллюстрация модели Лотки-Вольтерра.
Простая иллюстрация модели Лотки-Вольтерра.Ранее мы уже упоминали, что одно из применений биоинформатика находит в области молекулярной биологии. Работы, основанные на сборке геномов, сравнении и аннотировании и анализе последовательностей, так или иначе, прибегают к методам биоинформатики. Однако же в приближении, оказывается, что решение многих задач берёт своё начало в поиске наибольших совпадений между двумя последовательностями белковыми или нуклеотидными. В случае белковых рассматриваются последовательности аминокислот, в случае нуклеотидных — последовательности нуклеотидов соответственно. Одним из самых первых алгоритмов можно назвать алгоритм Нидлмана–Вунша, разработанный и опубликованный в 1970 году. Иные его названия: алгоритм оптимального соответствия или метод глобального выравнивания. Он применим для выравнивания любых двух строк, будь то аминокислотная, нуклеотидная последовательность или же просто набор букв, цифр.
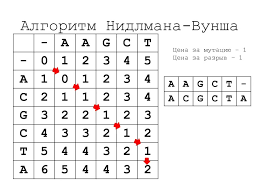
По своей сути компьютер (или человек) даёт оценку всем возможным выравниваниям (соотнося друг с другом последовательности всеми возможными вариантами). И по этой оценки в «баллах» (начисляемых или убавляемых за те или иные варианты) предлагает оптимальный вариант соотнесения представленных строк. Изначально задача может показаться простой, но в случае ДНК(РНК) и белков, вероятны разного рода замены, повторы, инделы (пропуски) в последовательностях, которые могут быть не отражены на сиквенсах (в силу разных причин) и создают большое количество самых разных вариантов соотнесения двух строк. И даже в случае использования мощных компьютеров — такие выравнивания могут занимать не один день.
Несмотря на высокое качество выравниваний при использовании алгоритмов, основанных на алгоритме Нидлмана-Вунша, потраченное на вычисление время, зачастую не соразмерно получаемым данным. И в этом случае на помощь приходят эвристические и вероятностные способы обработки данных. Такие методы не дают высокой точности по сравнению с описанным выше, однако сильно экономят на вычислительной мощности.Эвристические алгоритмы основаны на функции, которая ранжирует альтернативы на каждом шаге ветвления вариантов (ранжирование происходит на основе изначально установленных и введённых данных, то есть, основываясь на уже имеющейся информации) и, таким образом, конечный ответ будет приблизительно соответствовать оптимальному варианту. Такое решение не является абсолютно верным, но остаётся ценным в силу скорости получения ответа. На сходном принципе основана вся эвристика.
Вероятностные же методы, помимо использования основных алгоритмов выравнивания учитывают разные вероятности тех или иных замен. В этом случае строятся так называемые матрицы замен. Разные аминокислоты заменяются в процессе эволюции с разной вероятностью. И для учёта этой неравной вероятности замен используются матрицы. Не вдаваясь в частности, отметим, что и у этих методов есть свои недостатки. Так, точность вероятностных методов сильно уменьшается с увеличением дистанции между таксонами.
WinClada — одна из программ, используемая при работе с матрицами
Мы уже писали про использование матриц признаков выше. Как правило, такой подход используется в морфологических исследованиях. Метод используется в описательной биологии, когда, к примеру, мы хотим установить весь комплекс признаков для 2 или более видов. Безусловно, в приближении идея выглядит довольно просто. Давая количественную или качественную оценку разным признакам у представителей разных видов, мы можем достаточно быстро составить нужную матрицу. Однако же при работе с большими коллекциями или множеством видов (или признаков, а может быть и того и другого) проанализировать получившиеся данные становится крайне затруднительно человеческими силами. И на помощь приходят разные программы для обработки таких данных: TNT, WinClada, PAUP. С помощью них любой человек может провести кладистический анализ имеющихся матриц признаков.
Учитывая разнообразие задач, встающих перед биоинформатиками в разных областях биологии — появление огромного спектра самых разнообразных программ для обеспечения расчётов и анализа моделей — становится неизбежным. Зачастую эти программы не адаптированы для использования обывателем, содержат ошибки, а их оптимизация недостаточно хороша. Примером может служить программа PAST, используемая изначально для обсчёта палеонтологических данных, а в дальнейшем и для решения самых разных статистических задач, притом что там остаются ошибки в алгоритмах некоторых вычислений.Конечно, для стандартных задач уже существует немало понятных программ, с доступным руководством. Большое количество библиотек на таких языках программирования как Python или R. Но всё же круг задач в биологии, для которых использование методов биоинформатике могло бы сильно продвинуть исследования в этих областях — остаётся весьма широк. И востребованность специалистов в этой сфере всё ещё очень высока.
▍Места, где можно обучиться в России. Программы и курсы.
Отдельным плюсом биоинформатики можно отметить её доступность. Практически любой программист или математик может в весьма короткие сроки перепрофилироваться в эту область. Кроме того, любой из наших читателей может познакомиться с некоторыми принципами и методами этой науки. А при большом интересе и найти первую работу в этой области, так как спрос во многом превышает предложение и востребованность таких специалистов велика.
Многие курсы по биоинформатике доступны на разных онлайн-площадках подобных площадке Stepic. Мы предложим лишь несколько курсов, которые нам самим нравятся:
- Этот курс позволяет получить самые начальные навыки в биоинформатике и возможно подтолкнёт новичка к дальнейшему развитию своих навыков;
- А этот курс подойдёт тем, кто хочет получить продвинутые навыки программирования в подсистемах семейства Linux;
- Институт биоинформатики. У них Вы можете найти расширенный список курсов. И, возможно даже поступить на обучение.
▍Заключение.
На множественных примерах мы видим, что биоинформатика является важным разделом в современной биологии. Она оказывает огромное влияние на медицину и биологию, как фундаментальную, так и практическую. Статистические методы открывают небывалые перспективы для многих областей этих наук. При этом новое применение находят многие мониторинговые исследования, не имевшие большого применения в прошлом. Теперь эти огромные массивы данных могут быть проанализированы с применением вычислительной техники. Как это нередко бывает, применение методов на стыке дисциплин и междисциплинарные исследования приносят порой самые неожиданные результаты, которые оказывают большое влияние на развитие науки.
Следует отметить, что биоинформатика до сих принимается некоторыми учёными лишь за метод (к тому же не самый точный) помогающий в исследованиях. И, конечно, у них есть право так считать. На то есть комплекс причин, одна из самых очевидных: несовершенность моделей. Так, на данный момент времени создать идеальную модель той или иной живой системы и учесть все факторы и взаимодействие их влияний — практически невозможно.Мы же, в свою очередь, уверены, что информационные технологии хоть и не являются панацеей, но переводят многие исследования на совершенно новый уровень по скорости и точности исполнения. Это открывает (уже открыло) для человечества совершенно новые перспективы для развития как медицины, так и биологии в целом. А раз так, то мы и не вправе называть биоинформатику просто методом!
- Kuzmin, I., Petrov, I., Averianov, A., Boitsova, E., Skutschas, P., & Sues, H.-D. (2020). The braincase of Bissektipelta archibaldi — new insights into endocranial osteology, vasculature, and paleoneurobiology of ankylosaurian dinosaurs. Biological Communications, 65(2), 85–156. doi.org/10.21638/spbu03.2020.201
- Julia Bojarinova, Olga Babushkina, Photoperiodic conditions affect the level of locomotory activity during autumn migration in the Long-tailed Tit (Aegithalos c. caudatus), The Auk, Volume 132, Issue 2, 1 April 2015, Pages 370–379, doi.org/10.1642/AUK-14-155.1

Комментарии (25)

Vsevo10d
10.02.2022 02:12+2Я вижу надуманной проблему "наука, а не инструмент". То, что в обиходе биоинформатикой называют любой подход, позволяющий перелопатить огромное количество данных, не низводит ее из науки о данных к "всего лишь инструменту". Другое дело, что без адекватного понимания фундаментальных принципов биологии вы не сможете правильно интерпретировать полученные данные.
Вы можете омиксными подходами вытащить полный проетом или метаболом крови, например. Но вы не скажете только по этим данным, у вас некий белок повышен в результате заболевания, или же заболевание развивается от повышения этого белка. Вы можете говорить только о корелляции, а дальше вы обязаны копать в сторону экспрессии генов и метаболических цепей, чтобы понять, что причина, а что - следствие.
Так что биоинформатика - это безусловно наука, но для фундаментальной биологии она - инструмент, как и статистика, например. Или как гидродинамика - это наука, но в то же время инструмент, например, химической технологии.

phanerozoi_evidence Автор
10.02.2022 09:11+1Приветствуем. Ну вообще вы по факту все сказали. Нечего добавить. У нас примерно о том же. Кто-то, только как инструмент воспринимает, кто-то, включая пишущих алгоритмы, считает за науку=)

victor_1212
10.02.2022 02:36+1> .... дальше вы обязаны копать в сторону экспрессии генов и метаболических цепей, чтобы понять, что причина, а что - следствие
хорошо сказано, но не факт что это можно будет понять в обозримом будущем, хотя прогресс последние 20-30 лет большой, но скорее всего то, что мы видим только вершина айсберга, так что конечно наука и скорее всего фундаментальная, ясно одно, если есть возможность этим стоит заняться

phanerozoi_evidence Автор
10.02.2022 09:14+1В принципе мы специально и добавили ссылки на курсы, надеюсь они будут кому-нибудь полезны

VoronaDragon
10.02.2022 09:47+1Наука или инструмент? Есть универсальное название, которое устроит обе стороны - направление))

ArtBulatov
10.02.2022 15:51А наука это не инструмент?

phanerozoi_evidence Автор
10.02.2022 23:09Разница между наукой и инструментом тонка, конечно. Но если кратко попробовать обрисовать, то наука как инструмент к познанию мира намного шире, любого частного инструмента. Так как представляет постоянно пополняющуюся систему знаний. В то время как инструмент частный, типо той же световой микроскопии, например, имеет свои пределы. Будучи расширенным до какого-то предела - натыкается на ограничение ( например длину волны видимого спектра) и дальше лишь находит различные применения, но не расширяется по своей сути как система знаний.
Goupil
10.02.2022 15:55Я занимаюсь анализом биомедицинских изображений, в том числе с помощью ML. И я вот все не понимаю, я все таки биоинформатик или нет?

phanerozoi_evidence Автор
10.02.2022 23:11Если биомедицинские изображения обрабатываются с помощью информационных технологий, то биоинформатик, конечно. Статья как раз про то, что любое действие на стыке этих дисциплин и будет биоинформатикой

Loskechos
10.02.2022 19:04курсы на степик - самое бесполезное занятие которым я когда либо занимался. курсы построены так, что существует огромный пласт интернета с решениями этих заданий. Темы и задания вообще никоим образом не коррелируют.
я не совсем понял как старая добрая модель Лотке Вольтерра попала в эту статью? тем более что нормальные биофизики от нее уже давно не в восторге

phanerozoi_evidence Автор
10.02.2022 23:37курсы на степик - самое бесполезное занятие которым я когда либо занимался. курсы построены так, что существует огромный пласт интернета с решениями этих заданий. Темы и задания вообще никоим образом не коррелируют.
Ну во-первых, стоит прочитать текст внимательнее, ибо мы выбрали три, которые нам нравятся
Многие курсы по биоинформатике доступны на разных онлайн-площадках подобных площадке Stepic. Мы предложим лишь несколько курсов, которые нам самим нравятся:
Во-вторых, мы от части согласны с вами по поводу степика, именно поэтому в спискепонравившихся у нас нет. Однако, несмотря на то, что там есть безусловно тупые так сказать курсы, там есть и хорошие, по сертификатам которых открываются многие двери. Так что мы считаем, что не стоит быть столь категоричным, тем более, если подумать, даже к более продвинутым курсам можно найти ответы, ну задача курса не в том, чтобы все гуглили ответы, а в том чтоб человек разобрался сам. И если ты сам разобрался и решил задачу, то уж наверное какие-то знания после этого должны появиться и закрепиться.
я не совсем понял как старая добрая модель Лотке Вольтерра попала в эту статью? тем более что нормальные биофизики от нее уже давно не в восторге
Модель Лоттки-Вольтера к биофизике вообще по сути не имеет отношения. Но, возможно тут, вы случайно опечатались, по нашему предположению. Кроме того, в статье сказано, что модель просто одна из первых, опять же, в статье об этом сказано не для красного словца.. Конечно, она более чем не актуальна для каких-то точных расчетов на нынешний момент. На время своей публикации - это была вполне рабочая вещь показывающая неочевидную взаимосвязь двух популяций с вполне ясной и наглядной аргументацией.

W0lfgang
10.02.2022 20:33+1Зря Вы включили пример про экологию, так как он создает несколько неверное представление о биоинформатике как о науке. Все-таки у каждой науки должна быть некая область знаний, ограниченная конкретным признаком. И в случае с биоинформатикой это не любые биологические данные, к которым применимы информационные технологии. Если судить по образовательным программам, которые зовутся биоинформатикой, и по самоопределению членов данного научного сообщества, то биоинформатика должна изучать данные, относящиеся к молекулярной биологии, генетике и отчасти к биохимии. В итоге, учитывая конкретную область знаний, научную философию и, что немаловажно, научное сообщество, биоинформатику с уверенностью можно назвать наукой. А информационные технологии, которые, как Вы говорите "не панацея" применяются в биоинформатике только там, где это имеет смысл, иначе бы это было совсем ненаучно. Так, биоинформатика характеризуется не только информационными технологиями, но и конкретной областью знаний и связанной с ней парадигмой, что делает ее полноценной наукой со множеством разделов

phanerozoi_evidence Автор
10.02.2022 23:46Мы считаем, что в экологии есть биоинформатика, поскольку любые обсчеты со спутника, количества хлорофилла по цвету воды, например, определенно относится к экологии и информационным технологиям. И поскольку, биоинформатика является междисциплинарной областью, имеющей прямое отношение, как к общей биологии (а общая биология включает и раздел экологии), так и к молекулярной биологии и плюсом к компьютерным наукам и математике, то с каких пор это не биоинформатика?
Если только потому, что большинство в этой области занимаются обсчетом молекулярных данных, то это не совсем аргументированно. Водитель мотоцикла - тоже водитель, хотя автомобилей в мире больше. И если какой-то автомобилист не считает мотоциклиста водителем, то тот не перестаем им быть... Полагаю, такой пример уместен и немного раскрывает суть проблемы.

W0lfgang
11.02.2022 00:12Мы же (биоинформаты) считаем, что область знания каждой науки определяет в первую очередь научное сообщество, ограничивая ее конкретным признаком. Биоинформатика как синтез любого биологического направления и информационных технологий, - это определение, которое приводите именно Вы, но оно не является общепринятым в среде биоинформатов по крайней мере. Биоинформатика в большинстве случаев понимается как синтез гораздо меньшего числа биологических направлений с ИТ, чем приводите Вы.
Несмотря на Ваш отдаленный от темы пример с мотоциклом, по слову не всегда можно точно угадать его значение. Например, молекулярная биология (в российском понимании) - это не биология любых макромолекул в биологических системах. А ботаника и микология в общем случае (так уж исторически сложилась), как Вы знаете, изучают не только растения и грибы соотвественно.
Также в наше время почти в каждой науке наблюлается некое срастание с ИТ в силу технологического прогресса, но это далеко не всегда образует новое направление, так как многие операции до этого осуществлялись и без ИТ. Ваш график с численностью хищников и жертв существовал задолго до появления компьютера, как и модели органов и наблюдения за движениями птиц. Ученые просто использовали камеру, компьютер в качестве нового инструмента в рамках уже существующей дисциплины. А то, что относится к биоинформатике в общепринятом понимании как раз появилось благодаря новым методам, отчего и образовало новую область знаний. Примерно так же зародилась и молекулярная биология, выделившись благодаря довольно общим методам в узкое направление

phanerozoi_evidence Автор
11.02.2022 07:57Мы же (биоинформаты) считаем, что область знания каждой науки определяет в первую очередь научное сообщество, ограничивая ее конкретным признаком. Биоинформатика как синтез любого биологического направления и информационных технологий, - это определение, которое приводите именно Вы, но оно не является общепринятым в среде биоинформатов по крайней мере. Биоинформатика в большинстве случаев понимается как синтез гораздо меньшего числа биологических направлений с ИТ, чем приводите Вы.
аппеляция к себе и обобщение себя ко всем, это конечно интересно,но работы, на которые мы ссылались говорят об обратном, т.е. определение, которое мы дали вполне общепринятое. У нас нет желания спорить, потому что, скорее всего мы уйдем в область философских изречений, но так или иначе научное сообщество уже давно считает общеприянятым определение биоиформатики в том смысле, в котором обозначили его мы. Это же определение общедоступно и на английской википедии, которая весьма точно передает мысль научного сообщества.

W0lfgang
11.02.2022 12:00-1Вы ответили с новыми аргументами, хотя написали, что не хотите спорить. Что ж, продолжим тогда. Здесь речь идет не только об апелляции к себе, ведь Вы ответили только на первый абзац моего комментария.
Вы же апеллируете к абзацу Википедии с пометкой "clarification needed". Если Вам нравится Википедия, то лучше бы Вы обратились к немецкой версии, которая известна исключительно хорошей модерацией. Там как раз говорится, что в англоязычном мире биоинформатика часто противопоставляется более общей вычислительной биологии, хотя иногда эти термины выступают как синонимы. Подобная ситуация с молекулярной биологией: в англоязычном мире она изучает молекулярную основу вне~ и внутриклеточных процессов, а у нас в большей степени привязана к центральной догме.
Отчего я вообще решил прокомментировать Вашу работу. Тема, в которой Вы рассуждаете относится не столько к науке, сколько к научной философии, и является весьма дискуссионной, учитывая, что биоинформатика находится на заре развития. Во-первых, Вы привели не очень типичные для биоинформатики примеры, которые меня и заинтересовали. Во-вторых, именно они - те, что Вы здесь привели - в наименьшей степени порождают новую область знаний и выглядят скорее как методы, примененные в существующей дисциплине. Об этом я писал выше

phanerozoi_evidence Автор
11.02.2022 22:26Вы же апеллируете к абзацу Википедии с пометкой "clarification needed". Если Вам нравится Википедия, то лучше бы Вы обратились к немецкой версии, которая известна исключительно хорошей модерацией.
1) Можно пруфы, что модерация англовики плоха
2) Можно пруфы, что биоформатические методы в экологии не являтся отраслью биоформатики?

W0lfgang
12.02.2022 00:181. "Модерация англовики плоха" - это Ваш тезис, а не мой. Я сравнил ее с немецкой версией, так как последняя привлекательна прозрачной системой отметок хороших (проверенных и рецензированных) статей, а также наград за статьи и приложения. Меня в принципе удивило, что ваш выбор ссылки на авторитетный источник пал на Википедию. Примеры же из иных версий той же Википедии я привел в ответ, так как хотел проиллюстрировать действительно разный подход к определению, зависящий от локального научного сообщества. И это актуально не только для биоинформатики (см. выше).
Мой тезис заключался в том, что для определения направления, возникшего благодаря новым методам, в качестве науки необходимо порождение этими методами принципиально новой области знаний. Как я говорил выше, именно те примеры из Вашей работы, которыми Вы расширяете понятие биоинформатики, демонстрируют это меньше всего, отчего и привлекли мое внимание. К слову, именно это и является важным моментом в определении биоинформатики как науки. Отдельного внимания стоит Ваш конклюжн, который не полностью раскрывает Ваше мнение на жтот счет и плохо связан с приведенными примерами.
Если хорошим доказательством для Вас будет ссылка на авторитетный источник, отражающий мнение научного сообщества, то приведу ее на статью из Британской энциклопедии. В первом абзаце биоинформатика действительно определяется, как "гибридная наука, связывающая биологические данные [какие?] с методами их хранения, передачи и анализа". Во втором говорится, какие конкретно биологические данные имеются в виду: различного рода сиквенсы, модели макромолекулярных структур, "омиксные" данные, включая метагеномику (она действительно может относится к экологии, но не тем же образом, что Ваш пример, который сложно назвать биоинформатическим), которая по сути та же работа с сиквенсами, только из лужи.
Хочу повторить, что тема, которую Вы выбрали для Вашей статьи действительно является дискуссионной и требует привлечения дополнительных ссылок и понимания философии науки. Также Вас не должны в таком случае удивлять иные мнения на этот счет. Вы действительно пишете по делу, но в этой ветке почему-то избирательно реагируете на приводимые аргументы. Надеюсь, мне удалось прояснить для Вас свою точку зрения касательно этой непростой темы

W0lfgang
11.02.2022 12:05Отднльная статья из любимой Вами Википедии по вычислительной биологии. В ней говорится о большем и меньшем применении биоинформатики в тех или иных ее разделах. В русскоязычном сообществе это менее расхожий термин, но он тоже всегда является более общим понятием относительно биоинформатики

phanerozoi_evidence Автор
11.02.2022 09:06Также советуем ознакомиться с такой областью биоинформатики, как биоинформатика биразнообразия, которая в свою очередь порой уходит очень далеко в экологию. Вот пример статьи с использованием биоинформатики не только в геномике

AlexanderS
11.02.2022 10:52Так, команда DeepMind буквально год назад решила вопрос по предсказанию трёхмерной структуры белка по его аминокислотной последовательности.
Имеется ввиду фолдинг белков? Но ведь это реально очень сложная задача — для её обсчитывания целые распределенные вычислительные сети трудятся. Тот же Folding@home, например. Сляпать ПО с априорным нейросетевым обучением, обозвать его модным словом «ИИ» и презентовать как решение… ± и оно работать же не будет, так как четкой модели происходящего процесса нет или хотя бы понимание алгоритмического перебора.
phanerozoi_evidence Автор
11.02.2022 11:49А кто-то говорил, что это задача реально легкая?Команда deepmind поэтому реально крута, так как у них все работает на небольших белках с довольно высокой точностью. И это было очень сложно


shadrap
и должен отметить, что считают эти "противные ученые" в основном по другой причине...
никто не спорит с важностью вклада биоинформатики в развитие знаний о генетике и тп, но как и во всяком развивающемся деле, бывают перекосы, связанные с желанием "побыстрее натянуть сову на глобус". Например получившее широкое распространение Imputation genetics, когда за отсутствием реальных данных (сиквенса или еще чего-то) добавляются статистические, основанные на популяционном анализе. Т.е. взяли дешевое генотипирование на чипах , добавили туда что-то из статистики и опс.... люди потом спрашивают, а зачем полногеномное секвенирование , когда и так все видно, за гораздо меньшие деньги....??? Забывая при этом , что например, серповидно-клеточная анемия это мутация одного нуклеотида...
Может я не очень наглядный пример привел, но , те люди , которые пытаются молекулярный анализ поставить воглаву персонифицированного подхода, справедливо недовольны, когда некоторые начинают собирать данные доказательной медицины , что бы объяснить генетические оберрации. Т.е. - давайте опросим всех у кого есть SNP mutation Nxxxxxx , чувствуют ли они запах миндаля , на этом выводе построим недостающие данные и на будущее примем , что у всех их будет частично растворимый белок ХХХ , что является причиной аносмии (пример виртуальный ).
Действия этих людей понятны - им хочется быстрый результат за меньшие деньги, но... это подрывает стремление разобраться в деталях , понять механизм..