С момента появления COVID-19 мы застали множество “волн” и новых вспышек вируса. Помимо очевидной тяжести заболевания и невероятной скорости передачи, SARS-CoV-2 также отличается большим количеством различных мутаций, уклоняющихся от иммунных реакций.
Несмотря на то, что сейчас ситуация с коронавирусом стабилизировались, вирус продолжает развиваться и мутировать, что все равно сохраняет большую опасность для населения, в связи с трудностью и затратностью отслеживания его эволюции.
В данной статье я расскажу, как с помощью машинного обучения и новых методов кластеризации исследователям удалось встать на путь обнаружения новых вариантов вируса SARS-CoV-2, вызывающего COVID-19, со значительным временным и вычислительным выигрышем, по сравнению с существующими методами.
Введение в исследование
SARS-CoV-2 (вирус, вызывающий COVID-19), как и другие РНК-вирусы, имеет высокую скорость мутаций и короткое время генерации, что означает, что он эволюционирует так же быстро, как и распространяется. По этой причине филогенетический анализ стал мощным инструментом для отслеживания эволюции и распространения SARS-CoV-2.
РНК-вирусы — это вирусы, генетический материал которых представлен в виде РНК (рибонуклеиновая кислота), в отличие от более распространенных организмов, геном которых содержит ДНК (дезоксирибонуклеиновая кислота). РНК-вирусы включают в себя такие патогены, как вирус гриппа, вирус гепатита С, вирус ВИЧ, вирус гепатита B, коронавирусы (например, рассматриваемый SARS-CoV-2) и другие. Генетическая информация в РНК-вирусах может быть прямо использована для синтеза белков и для размножения вирусных частиц внутри клетки хозяина.
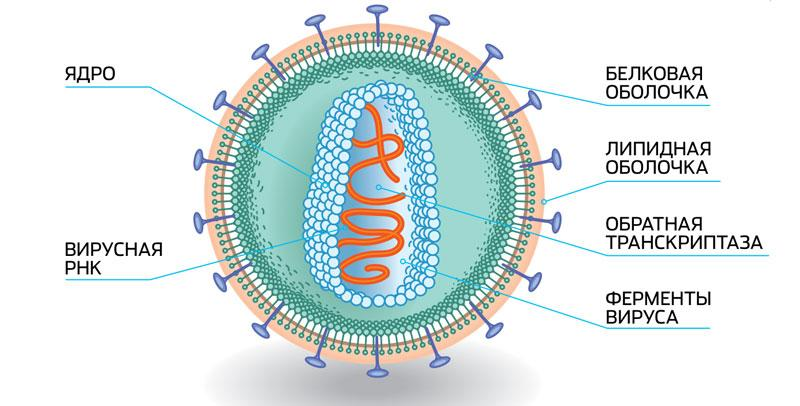
Филогенетический анализ — это изучение эволюции вида, группы организмов, геномов, белков и некодирующих последовательностей. С помощью филогенетического анализа ученые строят деревья родства, которые показывают, какие виды более тесно связаны и как происходило их эволюционное развитие. Этот метод позволяет понять и классифицировать организмы, а также изучать их эволюционные процессы.

Большинство точечных мутаций, или однонуклеотидных полиморфизмов (SNP), которые появляются на филогенетических древах, нейтральны или почти нейтральны, что означает, что сами мутации практически не влияют на передачу и на то, будут ли линии расти или вымрут. Однако некоторые мутации действительно обладают избирательным преимуществом, поскольку они обеспечивают более эффективную передачу в популяции в момент появления.
Однонуклеотидные полиморфизмы (SNP) — это небольшие генетические изменения, которые происходят в одном нуклеотиде (базовой паре) ДНК. Например, если у одного человека в определенном месте генома может быть аденин (A), а у другого человека в том же месте — цитозин (C), то это будет представлять собой однонуклеотидный полиморфизм.
Одной из особенностей эволюции SARS-CoV-2 становится появление и распространение вариантов VOCs (Variants of Concern) и некоторых их основных сублиний, каждые из которых имеют большое количество провоцирующих линию мутаций (+которые обеспечили этим вирусным линиям преимущества в передаче).
VOCs – это варианты вируса, которые вызывают беспокойство из-за их способности передаваться легче, вызывать более тяжелые формы болезни или снижать эффективность вакцин.
Можно выделить наиболее значимые для распространения ковида:
Alpha (B.1.1.7) - впервые обнаружен в Великобритании;
Beta (B.1.351) - впервые обнаружен в Южной Африке;
Gamma (P.1) - впервые обнаружен в Бразилии;
Delta (B.1.617.2) - впервые обнаружен в Индии.
Выявление вирусных линий, которые могут стать проблематичными в будущем, требует значительных усилий, включая выравнивание последовательностей по некоторому эталону перед их включением в филогенетическое дерево; назначение новых линий (в частности, присвоение им номенклатуры, основанной на их местоположении); и идентификацию линий с потенциально проблемными мутациями.
Создание филогений, содержащих все высококачественные геномы SARS-CoV-2, могло бы помочь в автоматизации этого процесса, но при наличии почти 16 миллионов последовательностей, доступных в базе данных GISAID, выравнивание значительной части последовательностей и создание единой филогении возможно только при чрезвычайно больших вычислительных ресурсах.
В данном исследовании рассматриваются менее трудоемкие в вычислительном отношении методы неконтролируемого машинного обучения, которые могут работать с огромными объемами генетических данных и могут быть использованы для выявления растущих значимых линий.
Решением стал анализ последовательностей без выравнивания.
Подходы к сравнению последовательностей без выравнивания можно определить как любой метод количественной оценки сходства/различия последовательностей, который не использует или не производит выравнивание (назначение соответствия остатков) на любом этапе применения алгоритма.
С самого начала такое ограничение ставит подходы без выравнивания в выгодное положение, поскольку они не зависят от динамического программирования, они вычислительно менее затратны (поскольку, как правило, имеют линейную сложность, зависящую только от длины запроса) и, следовательно, подходят для сравнения целых геномов.
Подходы без выравнивания можно в целом разделить на две группы: методы, основанные на частотах подпоследовательностей определенной длины (методы, основанные на статистике слов) и методы, оценивающие информационное содержание между последовательностями полной длины (методы, основанные на теории информации). Нас будет интересовать первая группа.
Методы, основанные на статистике слов
Суть этих методов проста: похожие последовательности содержат схожие слова/ k-mers (подпоследовательности длиной k), и математические операции с частотой встречаемости этих слов предоставляют хорошую относительную меру различия последовательностей.

Сначала сравниваемые последовательности должны быть разделены на наборы уникальных слов заданной длины. Например, две последовательности ДНК x = ATGTGTG и y = CATGTG и размер слова в три нуклеотида (3-mers) создают две коллекции уникальных слов: Wx = {ATG, TGT, GTG} и Wy = {CAT, ATG, TGT, GTG}. Поскольку некоторые слова часто присутствуют в одной последовательности, но не в другой (например, CAT в y, но не в x), создается полный набор слов, которые принадлежат по крайней мере к W3x или W3y для дальнейшего упрощения вычислений, в результате чего получаем набор объединения W3 = {ATG, CAT, GTG, TGT}.
Далее идет преобразование каждой последовательности в массив чисел (вектор) (например, путем подсчета количества раз, когда каждое конкретное слово встречается в последовательностях). Для последовательностей x и y идентифицируют два действительных вектора: C3x = (1, 0, 2, 2) и C3y = (1, 1, 1, 1).
Последний шаг включает количественную оценку различий между последовательностями посредством применения функции расстояния к векторам, представляющим последовательность, C3x и 3. Это различие очень часто вычисляется с помощью евклидова расстояния. Чем выше значение различия, тем более отдалены последовательности; таким образом, две идентичные последовательности приведут к расстоянию, равному 0.
Перейдем к методам и результатам исследования!

Методы и результаты исследования
Использовалась полная база данных GISAID, загруженная 19 января 2023 года, содержащая 14 617 387 последовательностей SARS-CoV-2.
GISAID (Global Initiative on Sharing All Influenza Data) – глобальная научная инициатива, созданная в 2008 году для обеспечения доступа к геномным данным вирусов гриппа. База данных была расширена за счёт включения коронавируса, ответственного за пандемию COVID-19, а также других патогенов.

Имеющиеся последовательности были отфильтрованы, чтобы включить только полные (содержащие более 29000 нуклеотидов) и с высоким перекрытием (<1% локусов идентифицированы как N) последовательности человеческого происхождения. В общей сложности отобрали 5,726,839 экземпляров.
*Символ "N" в генетической последовательности обычно используется для обозначения не идентифицированных нуклеотидов или "дырок" в последовательности, которые не удалось полностью определить в процессе секвенирования из-за различных технических или биологических причин.
Локусы в генетике – это конкретные места на хромосомах, где расположены гены. Каждый локус обозначает конкретное место на геноме, где может находиться аллель (вариант гена).
Как и указывалось выше, методы на основе статистики слов могут быть полезны, поскольку они позволяют анализировать последовательности быстро и на больших масштабах без необходимости проведения времязатратного и сложного процесса выравнивания. Таким образом, исследователи могли быстро оценить генетическую структуру популяций SARS-CoV-2, используя эти методы, и сравнить полученные результаты с традиционными методами филогенетического анализа.
Так, исследователи сначала сравнили проекцию 3-mers (k=3) из не выровненных последовательностей с филогенетическим деревом, построенным методом максимальной парсимонии с использованием IQTree, на основании выравнивания тех же последовательностей.
Метод максимальной парсимонии — это метод построения филогенетических деревьев, который основывается на принципе экономии (парсимонии). Согласно этому принципу, предпочтительнейшим считается тот сценарий эволюционного развития, который требует наименьшего количества эволюционных изменений (таких как мутации) для объяснения наблюдаемых различий в генетических последовательностях изучаемых организмов.
IQTree (IQ-TREE - Efficient Tree Reconstruction) – это программный инструмент для построения филогенетических деревьев на основе выравнивания последовательностей. Он использует методы максимального правдоподобия для построения наиболее достоверных филогенетических деревьев из молекулярных данных. IQTree предлагает широкий выбор моделей замены и методов оценки деревьев, что позволяет учитывать различные эволюционные процессы и дает возможность получить более точные результаты.
Ввиду высоких вычислительных затрат на создание филогенетических деревьев, исследователи использовали не выровненные последовательности для их анализа с помощью метода снижения размерности – PaCMAP для визуализации и выявления кластерной структуры данных. Это позволило быстро оценить генетическую структуру без необходимости проведения полного выравнивания.
Probabilistic and Compositional Multidimensional Analysis of Paired Data (PaCMAP) – это метод анализа данных, который используется для визуализации и понимания сложных многомерных данных. PaCMAP позволяет сжимать и представлять данные высокой размерности в двумерном или трехмерном пространстве, сохраняя при этом относительные расстояния между объектами.
Результат проецирования последовательностей в двумерном пространстве с помощью PaCMAP:
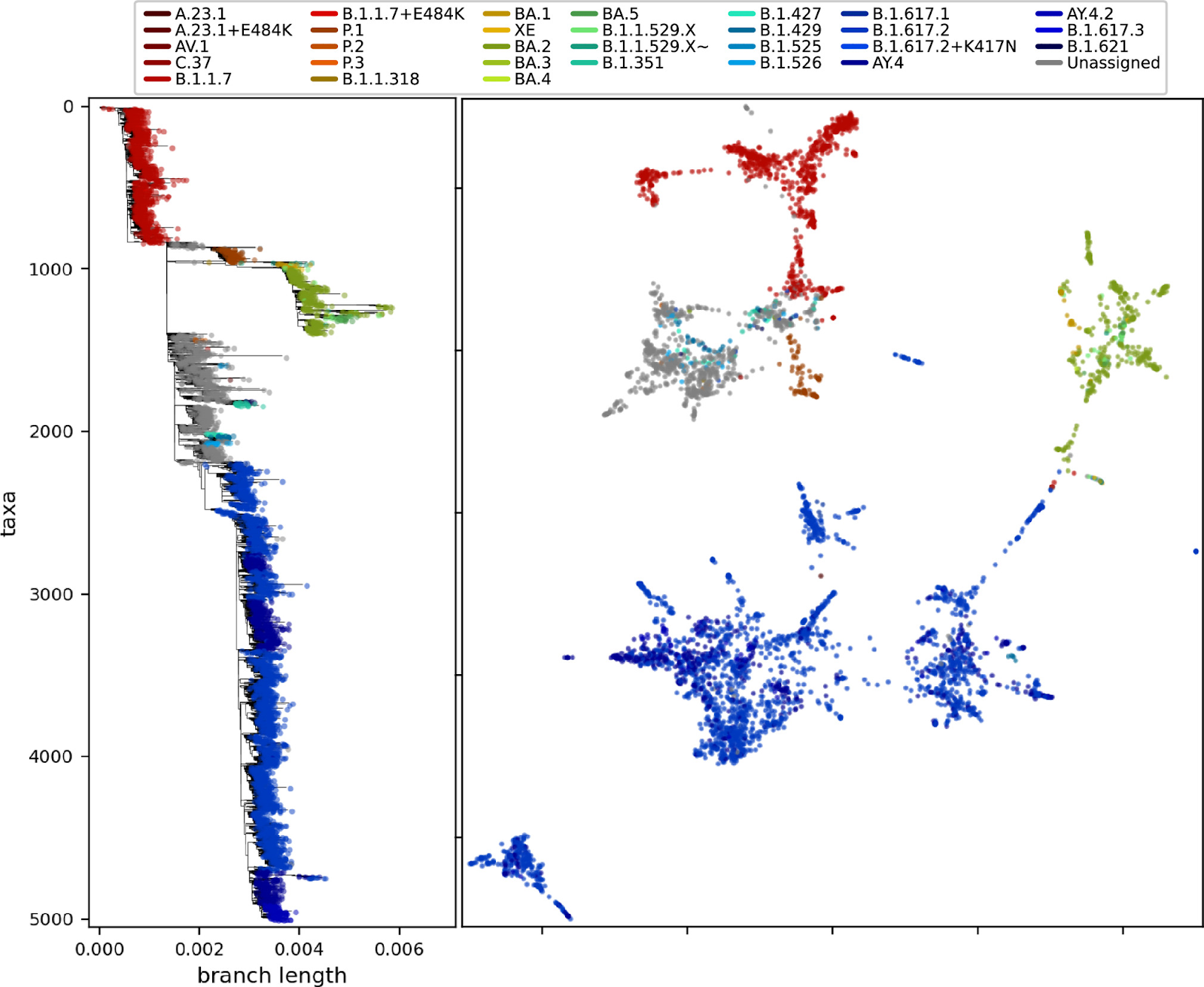
Можно обнаружить близость между важными вариантами вируса (кластерами), такими как “Альфа” (B.1.1.7), “Дельта” (B.1.617.2), “Омикрон” (BA.1) и “Гамма” (P.1) на двумерной проекции PaCMAP и филогенетическом дереве. Это демонстрирует потенциал комбинации методов, основанных на статистике слов, и средств снижения размерности для создания интерпретируемых структур для распределения значимых вариантов в генетическом пространстве.
Далее была сгенерирована проекция последовательностей на трехмерное пространство для дальнейшего анализа.
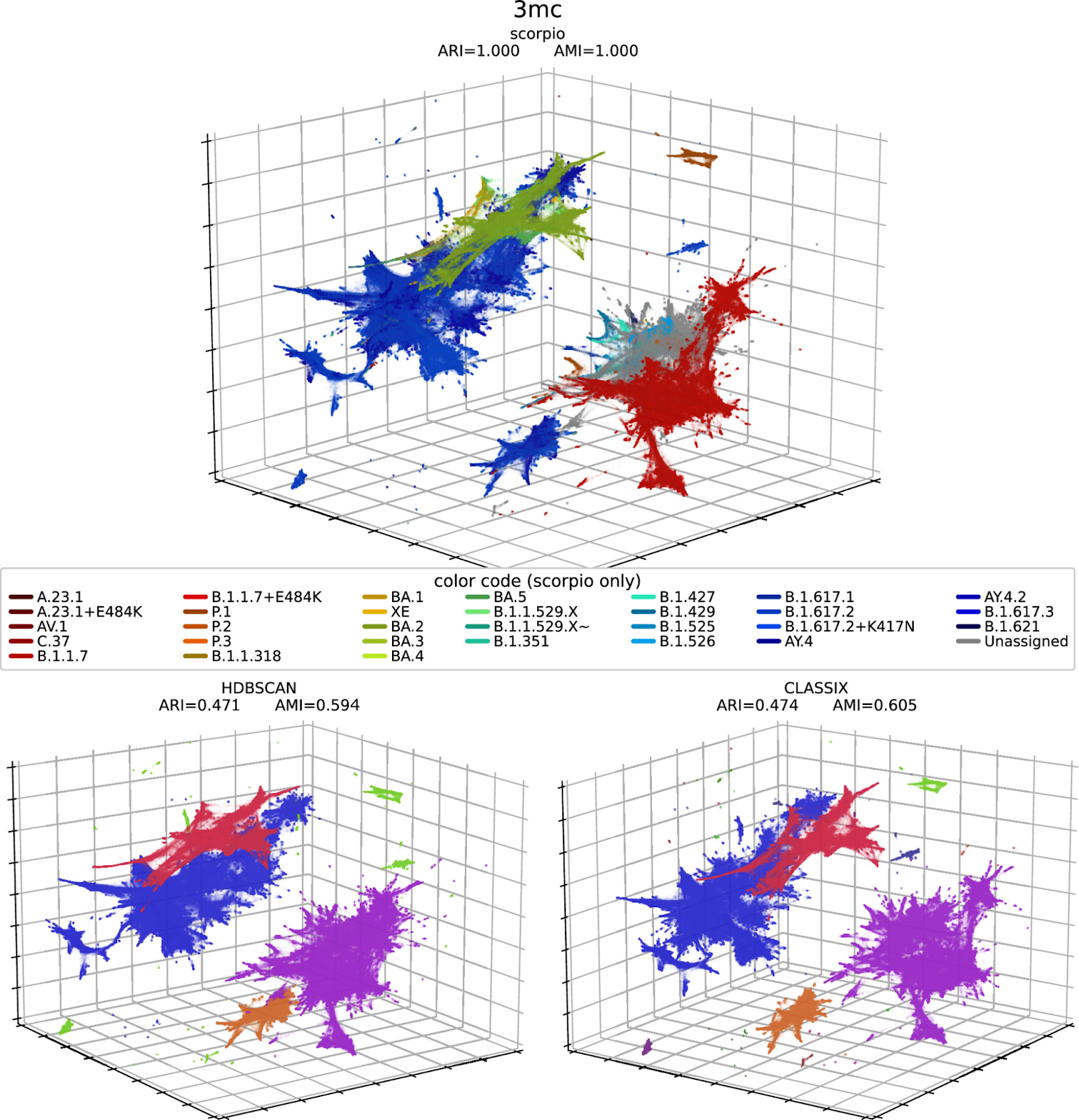
Для количественной оценки качества кластеров, сформированных в трехмерной проекции PaCMAP, реализовали два алгоритма кластеризации – HDBSCAN и CLASSIX. Эти алгоритмы предназначены для выявления кластеров в наборах данных на основе распределения плотности точек данных. В отличие от некоторых других методов кластеризации, здесь не требуется предварительное указание количества кластеров (что делает этот алгоритм более адаптируемым к различным наборам данных).
Метки Scorpio, полученные с помощью Pangolin, использовали в качестве "истины" для расчета метрик сходства кластеров: скорректированного индекса Рэнда (ARI) и скорректированного индекса взаимной информации (AMI) для матриц кластеризации. В обеих метриках значение 1 означало бы, что обнаруженные кластеры полностью эквивалентны "истинным" кластерам.
Adjusted Rand Index (ARI) – измеряет сходство между двумя назначениями кластеров, учитывая все пары выборок и подсчитывая пары, которые либо сгруппированы в один и тот же кластер, либо в разные кластеры в обоих назначениях.
Adjusted Mutual Information (AMI) – измеряет взаимную информацию между двумя назначениями кластеров, но корректируется на ожидаемое значение взаимной информации при случайном назначении кластеров.
Метки Scorpio получены в рамках программного пакета Pangolin (Phylogenetic Assignment of Named Global Outbreak LINeages). Pangolin используется для классификации штаммов вирусов, таких как SARS-CoV-2. Этот инструмент присваивает штаммам вируса определенные метки на основе их геномных последовательностей, позволяя идентифицировать и отслеживать распространение различных линий и вариантов вируса.
Здесь CLASSIX показал более высокие результаты по сравнению с более устоявшимся методом HDBSCAN, с ARI и AMI значениями 0.474 и 0.605 для CLASSIX по сравнению с 0.471 и 0.594 для HDBSCAN.
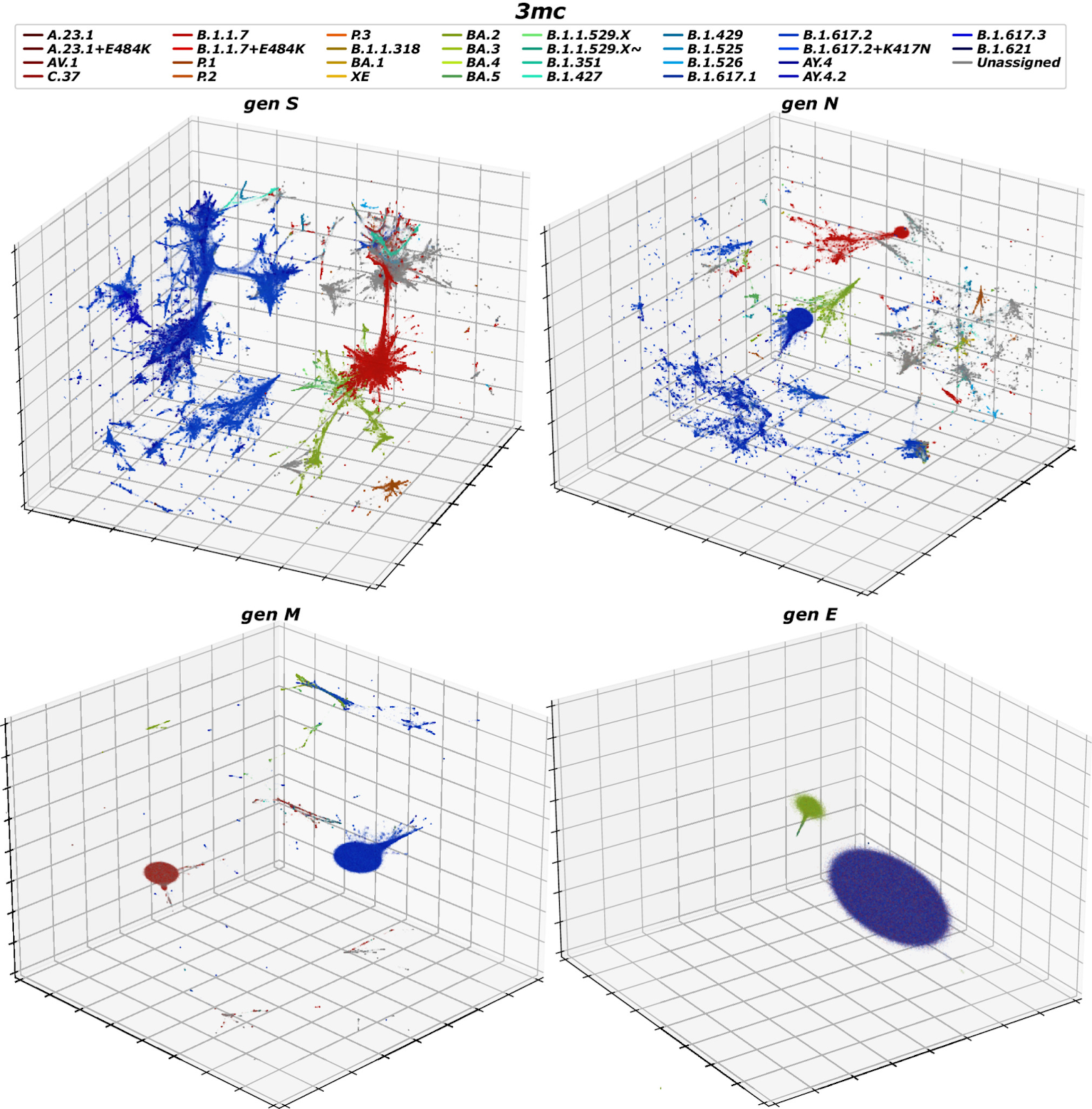
Затем исследователей заинтересовало, как рассматриваемая проекция будет работать, если ограничиться только основными структурными белковыми регионами. Для точного определения различных генных регионов использовали выровненные последовательности. Эта проекция была выполнена с использованием только параметров PaCMAP, поскольку цель этого анализа заключалась в сравнении регионов генома, а не в идентификации кластеров.
Основные структурные белковые регионы в контексте вирусов, таких как SARS-CoV-2, относятся к ключевым белкам, которые образуют структуру вирусной частицы. Эти белки включают:
S (Spike) — шиповидный белок, который отвечает за связывание вируса с рецептором на поверхности клетки-хозяина и последующее проникновение в клетку.
E (Envelope) — оболочечный белок, участвующий в сборке и выпуске вирусных частиц.
M (Membrane) — мембранный белок, который помогает формировать вирусную оболочку и способствует взаимодействию с другими структурными белками.
N (Nucleocapsid) — нуклеокапсидный белок, связывающий РНК вируса и формирующий нуклеокапсид.
Графики на рисунке ниже показывают, что хотя регионы S и N разбиваются на множество кластеров, связанных с различными метками Scorpio, регионы E и M гораздо более однородны. Относительная однородность, наблюдаемая для E и M, может быть частично обусловлена их относительно короткой длиной (соответственно 227 и 668 нуклеотидов) по сравнению с S и N (соответственно 1,259 и 3,821 нуклеотидов), а также возможно подверженностью более сильной адаптивной эволюции, в частности наличию антител, связывающихся с S и N.
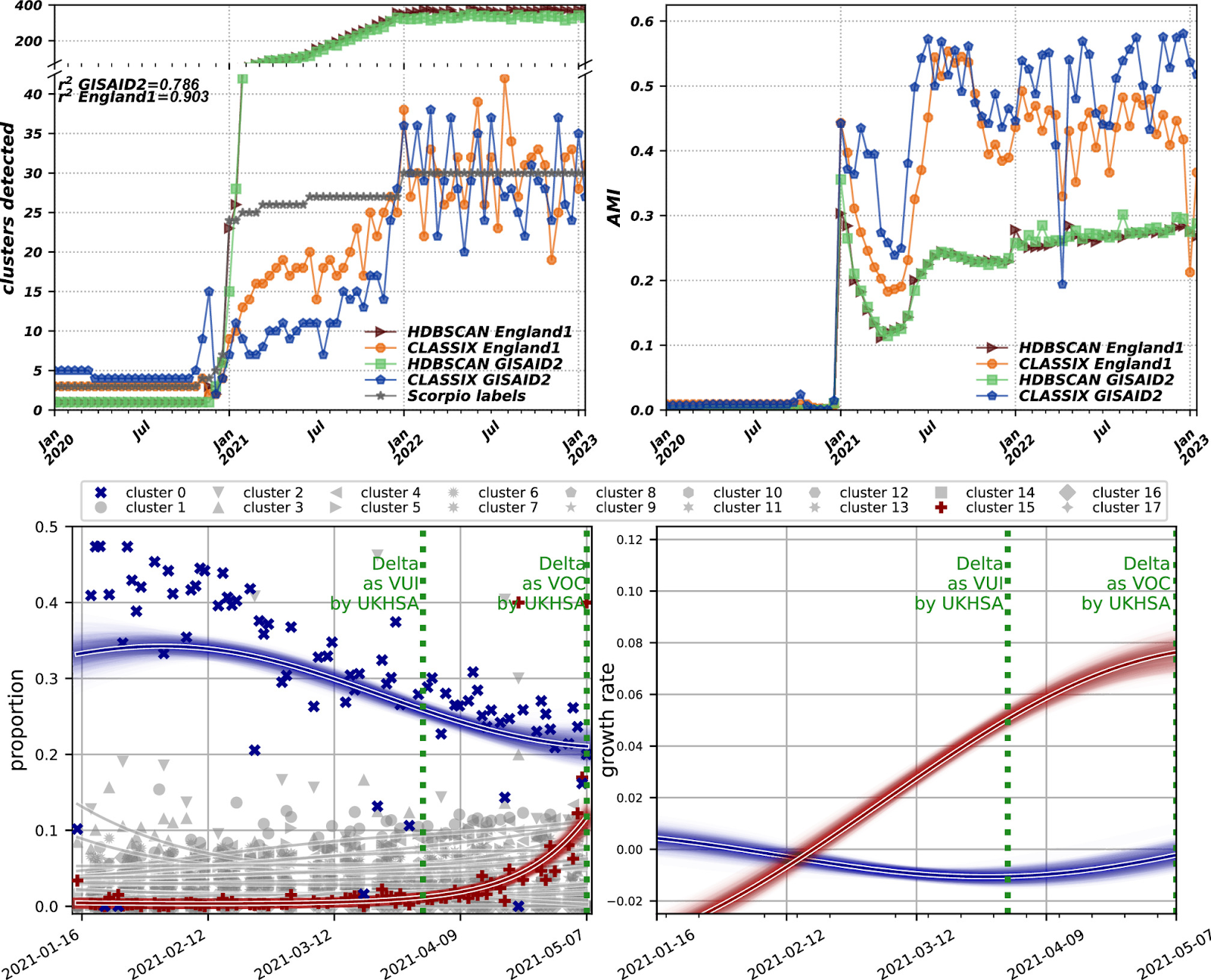
Исследователи проверили использование этого пайплайна для обнаружения появления новых вариантов, исходя из гипотезы, что они будут формировать новые кластеры по мере их появления. Для этого взяли подвыборку последовательностей, собранных в Англии (n=982,496), из которой были отобраны подмножества, формирующие временную кумулятивную прогрессию с двухнедельными шагами на основе даты их представления.
Временная кумулятивная прогрессия – это последовательность событий или изменений, которые накапливаются или увеличиваются по мере прохождения времени. Этот термин часто используется в контексте анализа данных или статистики для описания тенденции накопления чего-то со временем. Таким образом, исследователи начинают с анализа набора данных, собранных за первые две недели. Затем, для следующего анализа, они добавляют данные, собранные в следующие две недели, к уже существующей кумулятивной выборке, и так далее. С каждым шагом объем данных увеличивается, что позволяет отслеживать развитие и появление новых вариантов вируса с течением времени.
Также были внесены некоторые корректировки в параметры алгоритма кластеризации. Параметры HDBSCAN были настроены таким образом, чтобы повысить чувствительность к малым кластерам, что помогло бы более точно обнаруживать новые варианты. Параметры для CLASSIX оставили без изменений, как в первоначальной базе данных GISAID (GISAID1). В этот раз была введена новая параметрическая настройка (GISAID2), чтобы улучшить результаты.
Результаты можно увидеть в верхнем ряду рисунка 4, где видно, что HDBSCAN следует за Scorpio по числу кластеров, прежде чем превысить его обнаружение, в то время как CLASSIX остается близким, хотя и растет более постепенно в начале 2021 года.
Дальнейшая корректировка параметров PaCMAP привела к увеличению коэффициента корреляции Пирсона (r2) между обнаружением кластеров CLASSIX и маркировкой Scorpio с 0.785 до 0.903. Однако это изменение параметров снизило значение AMI в среднем на 0.049 с среднего значения 0.323 до 0.274, что указывает на снижение качества кластеров. Эта последняя параметрическая настройка была названа England1.
Коэффициент корреляции Пирсона (также известный как Pearson's r или r-коэффициент) — это статистический показатель, который измеряет линейную связь между двумя количественными переменными. Он показывает, насколько сильно изменения одной переменной соответствуют изменениям другой.
С точки зрения вычислительных требований, весь процесс от извлечения последовательностей до обнаружения кластеров может быть выполнен примерно за 30 часов на современном ноутбуке. С помощью метода статистики слов последовательность с высоким покрытием может быть обработана примерно за 0,12 секунды (используя параллелизацию, весь набор данных из 5,7 миллионов последовательностей может быть обработан примерно за 14 часов). Затем проекция PaCMAP занимает приблизительно 17 часов, в то время как кластеризация может быть выполнена менее чем за 5 минут.
Чтобы смоделировать условия обнаружения роста кластеров в "реальном времени", исследователи обработали серию накопительных подмножеств для создания "снимков", а именно проекцию 3mers-PaCMAP, используя параметрическую настройку GISAID1 и применяя эти алгоритмы к подмножеству последовательностей с датой предоставления равной или более ранней, чем заданная дата. После определения кластеров в любом данном "снимке" были рассчитаны ежедневные подсчеты последовательностей, принадлежащих каждому кластеру.
Исследователи применили это к периоду, в течение которого появился значимый вариант вируса – Дельта, раскрывая динамику среди кластеров, аналогичную динамике замещения варианта Альфа вариантом Дельта, которая наблюдалась в то время внутри вирусной популяции.

Наконец, была применена регрессия гауссовского процесса (GPR) для сглаживания тенденций подсчетов кластеров и для расчета темпов роста интересующих кластеров (доминирующего "кластера 0" и быстрорастущего "кластера 15"). Это дополнительно подтверждает потенциал этих методов для идентификации появления значимых линий. Эти результаты подытожены на рисунке 4.
Регрессия Гауссовского процесса (Gaussian Process Regression, GPR) — это метод машинного обучения, используемый для выполнения задач регрессии, то есть предсказания непрерывных значений. В отличие от других регрессионных моделей, которые могут быть ограничены определенными функциональными формами (например, линейной регрессией), GPR может аппроксимировать практически любую функцию благодаря своей гибкости и непараметрической природе.

Выводы
Вкратце подведем итоги!

Таким образом, благодаря данному исследованию мы стали ближе к решению проблемы необходимости ухода от сложных, перегруженных в вычислительном плане, филогенетических анализов.
Авторы работы успешно использовали статистические характеристики не выровненных последовательностей для анализа генетической структуры вирусных популяций, что позволило им обрабатывать огромные наборы данных с высоким покрытием на обычном ноутбуке в короткие сроки.
Применение таких инструментов, как проекция 3mers-PaCMAP и алгоритмов кластеризации типа HDBSCAN и CLASSIX, показало их способность к формированию интерпретируемых структур, которые отражают распределение значимых вариантов вируса в генетическом пространстве. Особенно интересным стало использование регрессии Гауссовского процесса для сглаживания тенденций в изменении численности кластеров и расчета темпов роста, что подтверждает потенциал этих методов в идентификации появления значимых линий вируса.
Автор исследования, Роберто Кауанци, также высказался об итогах проделанной работы: “Хотя филогенетика остается "золотым стандартом" для понимания вирусной родословной, эти методы машинного обучения могут охватить на несколько порядков больше последовательностей, чем текущие филогенетические методы, и при низких вычислительных затратах”.
На этом все! Будем ждать Вас в комментариях!
Спасибо за прочтение :)


shadrap
исследование из разряда "сова на глобус".
конечно когда есть практические данные по вариантам "Voc" можно попробовать подгонять под них статистику. И зачем действительно все эти сложности с филогенетикой и исследованием зооносных и обратных переходов, когда можно просто поиграть последовательностями и получить результат? А если подумать что одна и та же последовательность может подразумевать разную третичную структуру? и это уже другой белок с другими свойствами и он по другому отразится в мутационной активности? Для того что бы это учесть - нужно понимать с какого вида пришла мутация... и так мы возвращаемся назад ...)