Экспериментальный протокол и реализация алгоритма сортировки на программируемом ДНК-компьютере
Учёные давно ведут эксперименты с хранением информации в ДНК и с обработкой этой информации. Например, учёные из Вашингтонского университета и Microsoft недавно построили «первый в мире DNA-винчестер» (фото). Эта конструкция способна впервые обеспечить запись и считывание информации в ДНК-хранилище без участия человека. Весьма значительное достижение, если учесть, что в ДНК можно записывать информацию с плотностью 2,2 петабайта на грамм. ДНК — компактный контейнер с плотностью записи в тысячи раз больше, чем у существующих носителей.
Однако у всех существующих ДНК-систем есть проблема: всё это уникальные проприетарные разработки, у которых напрочь отсутствует какая бы ни было гибкость. Если сравнить с кремниевой техникой, то каждая группа исследователей с нуля разрабатывает новую архитектуру компьютера, для которого нужно писать новый софт. Но ситуация может измениться благодаря первому программируемому ДНК-компьютеру, разработанному в Калифорнийском университете в Дейвисе (UC Davis), Калифорнийского технологического института и Университета Мейнут.
Первый программируемый компьютер на ДНК описан в научной статье, которая опубликована 20 марта 2019 года в журнале Nature. Авторы показали, что с помощью простого триггера один и тот же базовый набор молекул ДНК способен реализовать множество различных алгоритмов. Хотя исследование представляет собой чисто лабораторный эксперимент, но программируемые молекулярные алгоритмы в будущем могут быть использованы, например, для программирования ДНК-роботов, которые уже успешно доставляют лекарства в раковые клетки.
«Это одна из знаковых работ в данной области, — говорит Торстен-Ларс Шмидт, доцент по экспериментальной биофизике в Университете штата Кент, который не участвовал в исследованиях. — Раньше уже демонстрировали алгоритмическую самосборку, но не до такой степени сложности».
В электронных компьютерах биты — это двоичные единицы информации. Они представляют собой дискретное физическое состояние базового оборудования, например, наличие или отсутствие электрического тока. Эти биты, или, скорее, электрические сигналы пропускаются через схемы, состоящие из логических элементов, которые выполняют операцию на одном или нескольких входных битах и производят один бит в качестве выхода.
Объединяя эти простые строительные блоки снова и снова, компьютеры могут запускать удивительно сложные программы. Идея ДНК-вычислений заключается в замене электрических сигналов химическими связями, а кремния — нуклеиновыми кислотами для создания биомолекулярного программного обеспечения.
Абстрактная иерархия архитектуры и практической реализации полной 6-битной логической схемы IBC (Iterated Boolean Circuit)
По словам Эрика Винфри, учёного из Калифорнийского технологического института и соавтора статьи, молекулярные алгоритмы используют естественные возможности обработки информации в ДНК, но вместо того, чтобы позволить природе взять бразды правления в свои руки, вычисления в ДНК производятся в соответствии с программой, написанной человеком.
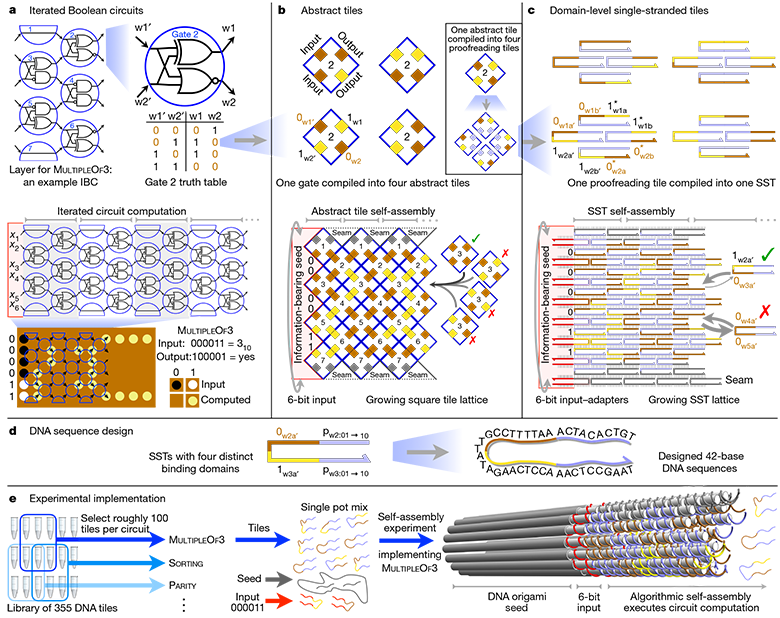
За последние 20 лет было проведено несколько успешных экспериментов с молекулярными алгоритмами, например, для игры в крестики-нолики или сборки молекул различной формы. В каждом случае требовалась тщательная разработка последовательности ДНК, чтобы выполнить один конкретный алгоритм, который будет генерировать структуру ДНК. В данном случае отличие состоит в том, что исследователи разработали систему, в которой одни и те же базовые фрагменты ДНК могут быть упорядочены для создания совершенно разных алгоритмов — и, следовательно, получения совершенно разного результата.
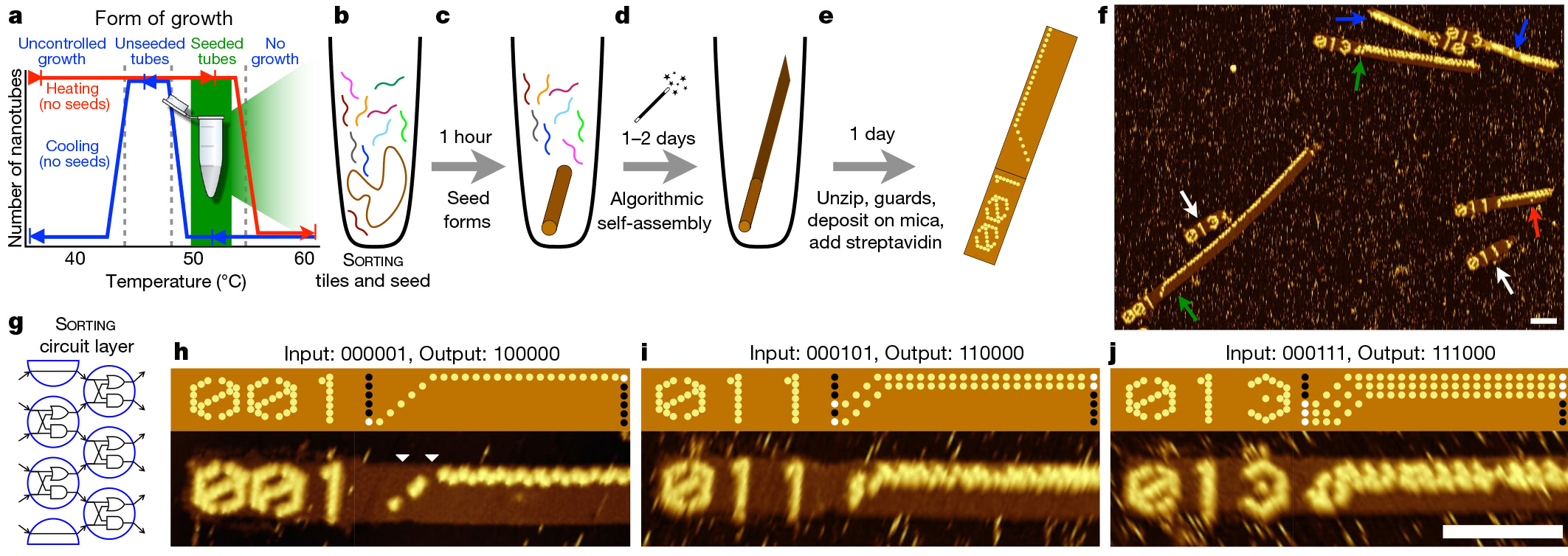
Процесс начинается с техники ДНК-оригами, то есть складывания длинной цепочки ДНК в желаемую форму. Этот сложенный кусочек работает как seed, который запускает алгоритмическую сборочную линию. Seed остаётся практически неизменным, независимо от алгоритма. Для каждого эксперимента в него вносят лишь небольшие изменения в несколько последовательностей.
Перепрограммирование логической схемы
После создания «семени» оно добавляется в раствор с сотней других нитей ДНК, известных как ДНК-плитки (DNA tiles). Учёные разработали 355 таких плиток. У каждой уникальное расположение азотистых оснований. Соответственно, для каждого алгоритма исследователи просто выбирают другой набор стартовых плиток. Поскольку эти фрагменты ДНК соединяются в процессе сборки, они образуют схему, которая реализует выбранный молекулярный алгоритм на входных битах, предоставляемых «семенем».
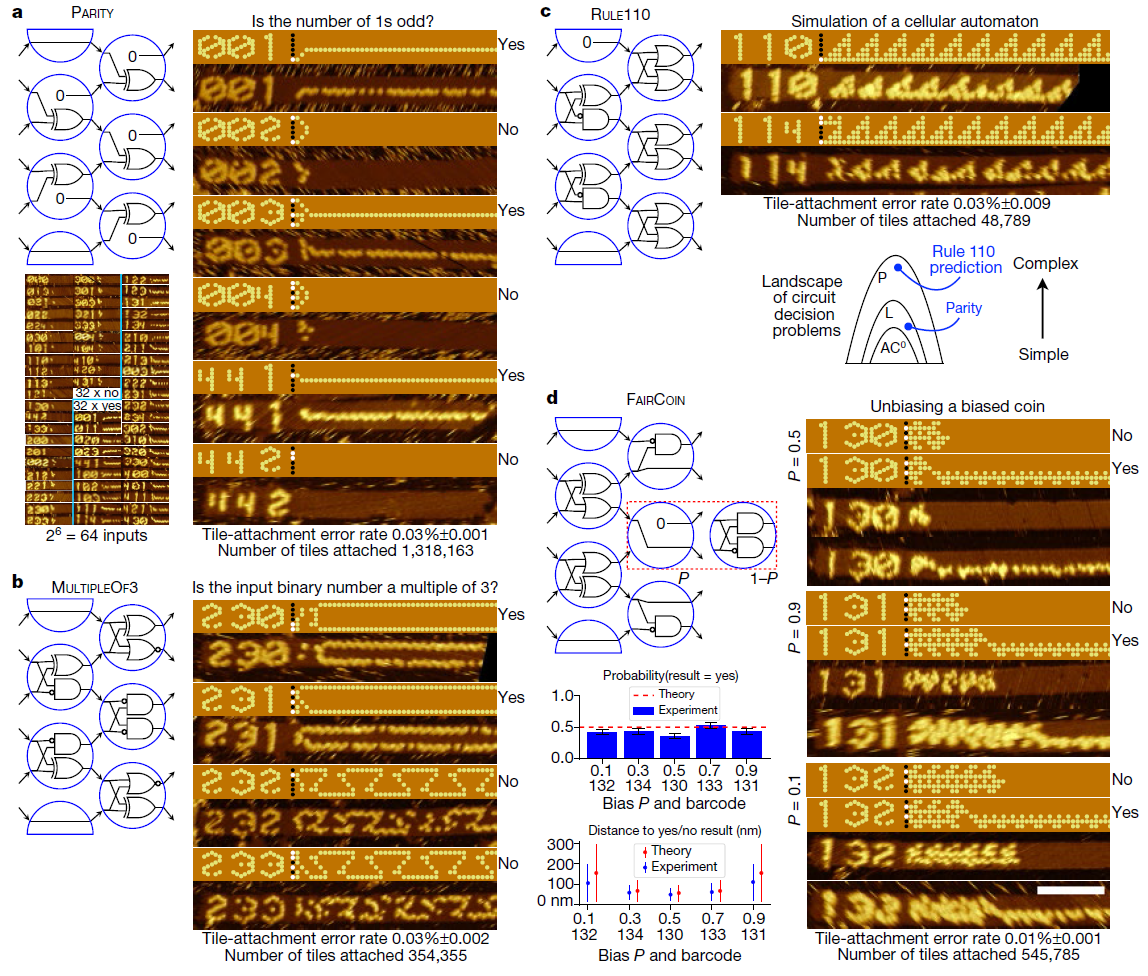
Используя эту систему, исследователи разработали и проверили 21 алгоритм для выполнения таких задач, как распознавание деления на три, выбор лидера, генерация паттернов и счёт с 0 до 63. Все эти алгоритмы реализованы с использованием различных комбинаций одних и тех же 355 плиток ДНК.
Конечно, непросто писать код, сбрасывая в пробирку фрагменты ДНК, но если автоматизировать процесс, то будущим молекулярным программистам даже не придётся задумываться о биомеханике, как сегодняшним программистам необязательно понимать физику транзисторов, чтобы писать хорошие программы.
Комментарии (15)

HellKaim
22.03.2019 18:03-1Интересно другое: мы ни как не можем добиться модели памяти и формирования нейронных связей, но, как топором, пытаемся программировать на ДНК.
Вопрос, конечно, что раньше случится… Но то, что это не эффективный метод, очевидно уже сей час (но самый доступный).

pallada92
22.03.2019 20:22+1Проблема подхода реализации компьютеров в духе Wang tiles «в лоб» на ДНК в том, что при таких вычислениях часто возникают ошибки. В этой статье указана частота 1 ошибка на 3000 плиток (я мог что-то напутать т.к. читал только abstract по диагонали). С учетом того, что число используемых плиток для самых простых алгоритмов пропорционально размеру входа в битах, умноженному на «число тактов» работы алгоритма, т.е. >100 – это довольно часто.
Вот картинка из одной из первых статей на эту тему от этих же авторов, где делали треугольник СерпинскогоЕсли увеличить правильный кусок, то складывается впечатление, что все работает идеально, однако вокруг происходит хаос из-за этих самых ошибок


DjSapsan
22.03.2019 20:27-1учитывая, что в одном грамме ДНК можно хранить петабайты, можно использовать такие компьютеры для хорошо паралерируемых задах. Может даже майнить Белкоины )

HellKaim
22.03.2019 20:49-1Именно, я выше об этом писал.
Наиболее целесообразно — базы данных, хранилища и т.п. где мало записи, но много чтения.
А вот "считать" так не очень выгодно.

HellKaim
22.03.2019 20:59Еще интересно попробовать синтез белков с люминисценцией. Тогда можно лазером читать.
Если запрограммировать синтез белка, части которого имеют разную люминисценцию, результат читать будет на порядок проще.
А вот как программировать процесс вычислений тут вопрос.


AxisPod
А какова скорость работы? Зачем он нужен, если вручную будет не особо медленее.
EvilGenius18
Как это «зачем он нужен»?
В разработке прорывных технологий неважно насколько быстро работает первая версия прототипа (как впрочем и его стоимость/размер/объем).
Когда создавались первые диски объемом несколько мегабайт, они и мечтать не могли, что 50 лет спустя у нас будут диски объемом в несколько терабайт, размером 1 см2 (SD диски) — 10 см2 (NVMe диски), имеющие скорость записи/чтения 3500 МБ/с
AxisPod
Скорость синтеза белков ну очень не быстрый процесс, как ускорять? Где здесь потенциал?
HellKaim
Скорость синтеза, кстати, огромна и хорошо запараллеливается.
Эти машины как раз и могут решить классическую проблему множественного доступа "by design"
AxisPod
20 аминоксислот в секунду, о да, очень быстро ) Уж простите, в любом случае химическая реакция по скорости будет куда медленее. Как параллелить? Особенно учитывая тот факт, что белок — нить. В разных «банках» запускать? Если всё в «одной», как контролировать это?
HellKaim
Эм.
"Репликационная вилка движется со скоростью порядка 100 000 пар нуклеотидов в минуту у прокариот и 500—5000 — у эукариот".
Обычно запускают чтение подряд или, если записано несколько раз подряд — одновременно несколько фрагментов.
Физику не изменить: читать и писать можно только в 1 поток что кремнием, что магнитом, что светом, что молекулой.
Вопрос только в том, на сколько компактно можно хранить данные.
ЗЫ. В суперспирализированнлм состоянии ДНК действительно очень компактно, но для чтения ее разворачивают в месте начала чтения и идут далее по молекуле (напоминает сломанную молнию, которая движется, но не открывает при этом шва).
HellKaim
Погуглите такое понятие как секвенирование ДНК.
То, что представлено на рисунке — похоже хроматография (вернее ее фото).
EvilGenius18
Прогресс в любой области/технологии так всегда и происходил, не так ли?
1. Мы создаем что-то новое
2. Многим людям кажется что это будет невозможно существенно улучшить в ближайшие сотни лет
3. В течении нескольких лет исследователи находят несколько способов улучшить изобретение в десятки — миллионы раз, применяя открытия из других научных областей
К примеру:
— Устройства хранения информации за 50 лет были улучшены в миллионы раз (килобайты => терабайты)
— Алгоритмы распознавания изображений за несколько десятков лет улучшились с «не существуют» до «лучше, чем люди». Многие даже и не мечтали, что ИИ будет лучше ученых в распознавания рака и управлении машинами.
— Микро чипы за несколько десятков лет также были усовершенствованы в тысячи раз (1 ядро@20mhz@микрометры => 64 ядер@3,000mhz@7нм)
— Ракеты только за последние 16 лет были улучшены в десятки 1-й компанией (SpaceX), мы перешли от 1 запуска до десятков запусков (Falcon), хотя об этом даже и не мечтали, многие думали что это невыполнимая задача (даже Nasa не осмелились начать решать эту проблему, до сих пор летают на одноразовых ракетах)
amarao
Он не нужен. Ни какое научное достижение не нужно, потому что его создание стоило безумных денег, оно медленное и неудобное.
Надо просто обесценивать всё. Художник начал рисовать — самое время подойти и сказать «ну и нафига снова это рисовать, уже же всё нарисовали, и получше чем это». Писатель начал писать — можно заметить, что есть куда более известные и талантливые произведения.
Искусство обесценивания, одним словом.